+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 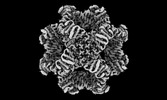 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Fast and versatile sequence- independent protein docking for nanomaterials design using RPXDock | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | octahedra / oligomer / de novo design / rosetta / cryoEM / interface / DE NOVO PROTEIN | |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
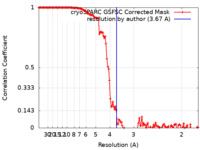
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.67 Å | |||||||||
 データ登録者 データ登録者 | Skotheim R / Borst AJ / Baker D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: PLoS Comput Biol / 年: 2023 ジャーナル: PLoS Comput Biol / 年: 2023タイトル: Fast and versatile sequence-independent protein docking for nanomaterials design using RPXDock. 著者: William Sheffler / Erin C Yang / Quinton Dowling / Yang Hsia / Chelsea N Fries / Jenna Stanislaw / Mark D Langowski / Marisa Brandys / Zhe Li / Rebecca Skotheim / Andrew J Borst / Alena ...著者: William Sheffler / Erin C Yang / Quinton Dowling / Yang Hsia / Chelsea N Fries / Jenna Stanislaw / Mark D Langowski / Marisa Brandys / Zhe Li / Rebecca Skotheim / Andrew J Borst / Alena Khmelinskaia / Neil P King / David Baker /   要旨: Computationally designed multi-subunit assemblies have shown considerable promise for a variety of applications, including a new generation of potent vaccines. One of the major routes to such ...Computationally designed multi-subunit assemblies have shown considerable promise for a variety of applications, including a new generation of potent vaccines. One of the major routes to such materials is rigid body sequence-independent docking of cyclic oligomers into architectures with point group or lattice symmetries. Current methods for docking and designing such assemblies are tailored to specific classes of symmetry and are difficult to modify for novel applications. Here we describe RPXDock, a fast, flexible, and modular software package for sequence-independent rigid-body protein docking across a wide range of symmetric architectures that is easily customizable for further development. RPXDock uses an efficient hierarchical search and a residue-pair transform (RPX) scoring method to rapidly search through multidimensional docking space. We describe the structure of the software, provide practical guidelines for its use, and describe the available functionalities including a variety of score functions and filtering tools that can be used to guide and refine docking results towards desired configurations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
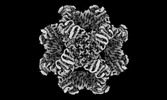
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29502.map.gz emd_29502.map.gz | 197.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29502-v30.xml emd-29502-v30.xml emd-29502.xml emd-29502.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29502_fsc.xml emd_29502_fsc.xml | 12.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29502.png emd_29502.png | 69.7 KB | ||
| Filedesc metadata |  emd-29502.cif.gz emd-29502.cif.gz | 5.5 KB | ||
| その他 |  emd_29502_additional_1.map.gz emd_29502_additional_1.map.gz emd_29502_additional_2.map.gz emd_29502_additional_2.map.gz emd_29502_half_map_1.map.gz emd_29502_half_map_1.map.gz emd_29502_half_map_2.map.gz emd_29502_half_map_2.map.gz | 103.1 MB 178.7 MB 194 MB 194 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29502 http://ftp.pdbj.org/pub/emdb/structures/EMD-29502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29502 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29502 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29502.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29502.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.885 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_29502_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: DeepEMhancer
| ファイル | emd_29502_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | DeepEMhancer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_29502_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_29502_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : O43-rpxdock-EK1
| 全体 | 名称: O43-rpxdock-EK1 |
|---|---|
| 要素 |
|
-超分子 #1: O43-rpxdock-EK1
| 超分子 | 名称: O43-rpxdock-EK1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: O43-rpxdoc-EK1_A
| 分子 | 名称: O43-rpxdoc-EK1_A / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 26.624115 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EEAELAYLLG ELAYKLGEYR IAIRAYRIAL KRDPNNAEAW YNLGNAYTKQ GDYDEAIEYY LRALVLDPNN AEAATNLGQA YMNQGDKDR AKLMLLLALK LDPNNDSARV ILGVAKVGIE ELAKLASQAQ QEGDSEKQKA IELAAEAARV AQEVGDPELE K LALEAARR ...文字列: EEAELAYLLG ELAYKLGEYR IAIRAYRIAL KRDPNNAEAW YNLGNAYTKQ GDYDEAIEYY LRALVLDPNN AEAATNLGQA YMNQGDKDR AKLMLLLALK LDPNNDSARV ILGVAKVGIE ELAKLASQAQ QEGDSEKQKA IELAAEAARV AQEVGDPELE K LALEAARR GDSEKAKAIL LAAEAARVAK EVGDPELIKL ALEAARRGDS EKARAILEAA ERAREAKERG DPEQIKKARE LA KR |
-分子 #2: O43-rpxdoc-EK1_B
| 分子 | 名称: O43-rpxdoc-EK1_B / タイプ: protein_or_peptide / ID: 2 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 15.268709 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DVSALAYVML GLLLSLLNRL SLAAEAYKKA IELDPNDALA WLLLGSVLEK LKRLDEAAEA YKKAIELKPN DASAWKELGK VLEKLGRLD EAAEAYLIAI MLDPEDAEAA KELGKVLEKL GELEMAEEAY KLAIKLDPND |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/2 / 支持フィルム - 材質: CARBON |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X