+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Langya Virus Fusion Protein (LayV-F) in Post-Fusion Conformation | |||||||||
 マップデータ マップデータ | Half Map B | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Glycoprotein / Fusion / Paramyxovirus / Henipavirus / Viral Protein | |||||||||
| 生物種 |  Langya virus (ウイルス) Langya virus (ウイルス) | |||||||||
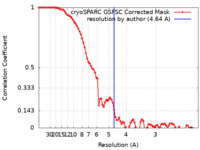
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.64 Å | |||||||||
 データ登録者 データ登録者 | May AJ / Pothula KR / Janowska K / Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2023 ジャーナル: J Virol / 年: 2023タイトル: Structures of Langya Virus Fusion Protein Ectodomain in Pre- and Postfusion Conformation. 著者: Aaron J May / Karunakar Reddy Pothula / Katarzyna Janowska / Priyamvada Acharya /  要旨: Langya virus (LayV) is a paramyxovirus in the genus, closely related to the deadly Nipah (NiV) and Hendra (HeV) viruses, that was identified in August 2022 through disease surveillance following ...Langya virus (LayV) is a paramyxovirus in the genus, closely related to the deadly Nipah (NiV) and Hendra (HeV) viruses, that was identified in August 2022 through disease surveillance following animal exposure in eastern China. Paramyxoviruses present two glycoproteins on their surface, known as attachment and fusion proteins, that mediate entry into cells and constitute the primary antigenic targets for immune response. Here, we determine cryo-electron microscopy (cryo-EM) structures of the uncleaved LayV fusion protein (F) ectodomain in pre- and postfusion conformations. The LayV-F protein exhibits pre- and postfusion architectures that, despite being highly conserved across paramyxoviruses, show differences in their surface properties, in particular at the apex of the prefusion trimer, that may contribute to antigenic variability. While dramatic conformational changes were visualized between the pre- and postfusion forms of the LayV-F protein, several domains remained invariant, held together by highly conserved disulfides. The LayV-F fusion peptide (FP) is buried within a highly conserved, hydrophobic interprotomer pocket in the prefusion state and is notably less flexible than the rest of the protein, highlighting its "spring-loaded" state and suggesting that the mechanism of pre-to-post transition must involve perturbations to the pocket and release of the fusion peptide. Together, these results offer a structural basis for how the Langya virus fusion protein compares to its Henipavirus relatives and propose a mechanism for the initial step of pre- to postfusion conversion that may apply more broadly to paramyxoviruses. The genus is quickly expanding into new animal hosts and geographic locations. This study compares the structure and antigenicity of the Langya virus fusion protein to other henipaviruses, which have important vaccine and therapeutic development implications. Furthermore, the study proposes a new mechanism to explain the early steps of the fusion initiation process that can be more broadly applied to the family. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29032.map.gz emd_29032.map.gz | 167.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29032-v30.xml emd-29032-v30.xml emd-29032.xml emd-29032.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29032_fsc.xml emd_29032_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29032.png emd_29032.png | 30.7 KB | ||
| Filedesc metadata |  emd-29032.cif.gz emd-29032.cif.gz | 5.9 KB | ||
| その他 |  emd_29032_additional_1.map.gz emd_29032_additional_1.map.gz emd_29032_half_map_1.map.gz emd_29032_half_map_1.map.gz emd_29032_half_map_2.map.gz emd_29032_half_map_2.map.gz | 167.4 MB 165.1 MB 165.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29032 http://ftp.pdbj.org/pub/emdb/structures/EMD-29032 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29032 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29032 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29032_validation.pdf.gz emd_29032_validation.pdf.gz | 702.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29032_full_validation.pdf.gz emd_29032_full_validation.pdf.gz | 702.2 KB | 表示 | |
| XML形式データ |  emd_29032_validation.xml.gz emd_29032_validation.xml.gz | 19.2 KB | 表示 | |
| CIF形式データ |  emd_29032_validation.cif.gz emd_29032_validation.cif.gz | 24.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29032 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29032 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29032 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29032 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29032.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29032.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Supplemental map prior to final local refinement.
| ファイル | emd_29032_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Supplemental map prior to final local refinement. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Final 3D Refinement
| ファイル | emd_29032_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final 3D Refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A
| ファイル | emd_29032_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Langya Virus Fusion Protein Ectodomain Post-fusion Conformation
| 全体 | 名称: Langya Virus Fusion Protein Ectodomain Post-fusion Conformation |
|---|---|
| 要素 |
|
-超分子 #1: Langya Virus Fusion Protein Ectodomain Post-fusion Conformation
| 超分子 | 名称: Langya Virus Fusion Protein Ectodomain Post-fusion Conformation タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Langya virus (ウイルス) Langya virus (ウイルス) |
| 分子量 | 理論値: 180 KDa |
-分子 #1: Fusion Protein
| 分子 | 名称: Fusion Protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Langya virus (ウイルス) Langya virus (ウイルス) |
| 分子量 | 理論値: 59.02577 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: SLHYDSLSKV GIIKGLTYNY KIKGSPSTKL MVVKLIPNID GVRNCTQKQF DEYKNLVKNV LEPVKLALNA MLDNVKSGNN KYRFAGAIM AGVALGVATA ATVTAGIALH RSNENAQAIA NMKNAIQNTN EAVKQLQLAN KQTLAVIDTI RGEINNNIIP V INQLSCDT ...文字列: SLHYDSLSKV GIIKGLTYNY KIKGSPSTKL MVVKLIPNID GVRNCTQKQF DEYKNLVKNV LEPVKLALNA MLDNVKSGNN KYRFAGAIM AGVALGVATA ATVTAGIALH RSNENAQAIA NMKNAIQNTN EAVKQLQLAN KQTLAVIDTI RGEINNNIIP V INQLSCDT IGLSVGIKLT QYYSEILTAF GPALQNPVNT RITIQAISSV FNRNFDELLK IMGYTSGDLY EILHSGLIRG NI IDVDVEA GYIALEIEFP NLTLVPNAVV QELMPISYNV DGDEWVTLVP RFVLTRTTLL SNIDTSRCTV TESSVICDND YAL PMSYEL IGCLQGDTSK CAREKVVSSY VPRFALSDGL VYANCLNTIC RCMDTDTPIS QSLGTTVSLL DNKKCLVYQV GDIL ISVGS YLGEGEYSAD NVELGPPVVI DKIDIGNQLA GINQTLQNAE DYIEKSEEFL KGINPSIGSG YIPEAPRDGQ AYVRK DGEW VLLSTFLGRS LEVLFQGPGH HHHHHHHSAW SHPQFEKGGG SGGGGSGGSA WSHPQFEK |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 6179 / 平均電子線量: 56.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: SwissModel / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL |
| 得られたモデル |  PDB-8fel: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)