+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a Zinc-loaded asymmetrical TMD D70A mutant of the YiiP-Fab complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報zinc efflux active transmembrane transporter activity / cadmium ion transmembrane transporter activity / ferrous iron transmembrane transporter activity / intracellular zinc ion homeostasis / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Shewanella oneidensis (バクテリア) / Shewanella oneidensis (バクテリア) /  Shewanella oneidensis MR-1 (バクテリア) / Shewanella oneidensis MR-1 (バクテリア) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
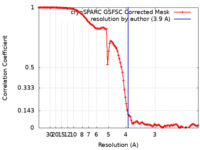
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Lopez-Redondo ML / Hussein AK / Stokes DL | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Characterization of individual Zn2+ binding sites of YiiP 著者: Lopez-Redondo ML / Hussein AK / Stokes DL | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28883.map.gz emd_28883.map.gz | 129.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28883-v30.xml emd-28883-v30.xml emd-28883.xml emd-28883.xml | 21.7 KB 21.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28883_fsc.xml emd_28883_fsc.xml | 10.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28883.png emd_28883.png | 36.7 KB | ||
| マスクデータ |  emd_28883_msk_1.map emd_28883_msk_1.map | 137.1 MB |  マスクマップ マスクマップ | |
| その他 |  emd_28883_half_map_1.map.gz emd_28883_half_map_1.map.gz emd_28883_half_map_2.map.gz emd_28883_half_map_2.map.gz | 127.2 MB 127.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28883 http://ftp.pdbj.org/pub/emdb/structures/EMD-28883 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28883 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28883 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28883_validation.pdf.gz emd_28883_validation.pdf.gz | 781.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28883_full_validation.pdf.gz emd_28883_full_validation.pdf.gz | 781.3 KB | 表示 | |
| XML形式データ |  emd_28883_validation.xml.gz emd_28883_validation.xml.gz | 19.4 KB | 表示 | |
| CIF形式データ |  emd_28883_validation.cif.gz emd_28883_validation.cif.gz | 25.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28883 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28883 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28883 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28883 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28883.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28883.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.079 Å | ||||||||||||||||||||
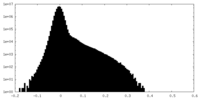
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28883_msk_1.map emd_28883_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
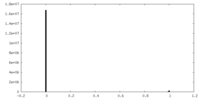
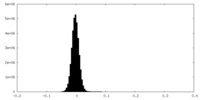
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28883_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
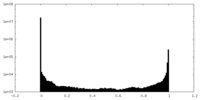
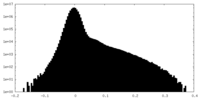
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28883_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
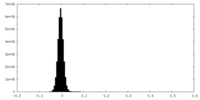
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : D70A asymmetrical Zinc-loaded YiiP-Fab complex
| 全体 | 名称: D70A asymmetrical Zinc-loaded YiiP-Fab complex |
|---|---|
| 要素 |
|
-超分子 #1: D70A asymmetrical Zinc-loaded YiiP-Fab complex
| 超分子 | 名称: D70A asymmetrical Zinc-loaded YiiP-Fab complex / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Shewanella oneidensis (バクテリア) Shewanella oneidensis (バクテリア) |
| 分子量 | 理論値: 163.024 KDa |
-分子 #1: Cadmium and zinc efflux pump FieF
| 分子 | 名称: Cadmium and zinc efflux pump FieF / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Shewanella oneidensis MR-1 (バクテリア) Shewanella oneidensis MR-1 (バクテリア) |
| 分子量 | 理論値: 32.441203 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTQTSQYDFW VKLASRASVA TALTLITIKL LAWLYSGSAS MLASLTDSFA DTLASIINFI AIRYAIVPAA HDHRYGHGKA EPLAALAQS AFIMGSAFLL LFYGGERLLN PSPVENATLG VVVSVVAIVL TLALVLLQKR ALAATNSTVV EADSLHYKSD L FLNAAVLL ...文字列: MTQTSQYDFW VKLASRASVA TALTLITIKL LAWLYSGSAS MLASLTDSFA DTLASIINFI AIRYAIVPAA HDHRYGHGKA EPLAALAQS AFIMGSAFLL LFYGGERLLN PSPVENATLG VVVSVVAIVL TLALVLLQKR ALAATNSTVV EADSLHYKSD L FLNAAVLL ALVLSQYGWW WADGLFAVLI ACYIGQQAFD LGYRSIQALL DRELDEDTRQ RIKLIAKEDP RVLGLHDLRT RQ AGKTVFI QFHLELDGNL SLNEAHSITD TTGLRVKAAF EDAEVIIHQD PVQVEPTTQ |
-分子 #2: Fab2r light chain
| 分子 | 名称: Fab2r light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.580242 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQIWSWPLIT FGQGTKVEIK RTVAAPSVFI FPPSDSQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQIWSWPLIT FGQGTKVEIK RTVAAPSVFI FPPSDSQLKS GTASVVCLLN NFYPREAKVQ WKVDNALQSG N SQESVTEQ DSKDSTYSLS STLTLSKADY EKHKVYACEV THQGLSSPVT KSFNRGEC |
-分子 #3: Fab2r heavy chain
| 分子 | 名称: Fab2r heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.406352 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF TIYSSSIHWV RQAPGKGLEW VASIYSSSGS TYYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARQSYSGLSP RRHWSYGAMD YWGQGTLVTV FNQIKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF TIYSSSIHWV RQAPGKGLEW VASIYSSSGS TYYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARQSYSGLSP RRHWSYGAMD YWGQGTLVTV FNQIKGPSVF PLAPSSKSTS GGTAALGCLV K DYFPEPVT VSWNSGALTS GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKKV EPKSCDKTHT |
-分子 #4: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 4 / コピー数: 6 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 7.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 5 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.02 kPa / 詳細: PELCO easiGLOW | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 3420 / 平均露光時間: 2.5 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X