+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Gentamicin bound aminoglycoside efflux pump AcrD | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | aminoglycoside efflux pump / AcrD / E.coli / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | Hydrophobe/amphiphile efflux-1 HAE1 / Acriflavin resistance protein / Multidrug efflux transporter AcrB TolC docking domain, DN/DC subdomains / AcrB/AcrD/AcrF family / efflux transmembrane transporter activity / xenobiotic transmembrane transporter activity / plasma membrane / Efflux pump membrane transporter 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
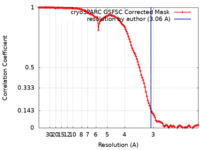
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.06 Å | |||||||||
 データ登録者 データ登録者 | Zhang Z | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: mBio / 年: 2023 ジャーナル: mBio / 年: 2023タイトル: Cryo-EM Structures of AcrD Illuminate a Mechanism for Capturing Aminoglycosides from Its Central Cavity. 著者: Zhemin Zhang / Christopher E Morgan / Meng Cui / Edward W Yu /  要旨: The Escherichia coli acriflavine resistance protein D (AcrD) is an efflux pump that belongs to the resistance-nodulation-cell division (RND) superfamily. Its primary function is to provide resistance ...The Escherichia coli acriflavine resistance protein D (AcrD) is an efflux pump that belongs to the resistance-nodulation-cell division (RND) superfamily. Its primary function is to provide resistance to aminoglycoside-based drugs by actively extruding these noxious compounds out of E. coli cells. AcrD can also mediate resistance to a limited range of other amphiphilic agents, including bile acids, novobiocin, and fusidic acids. As there is no structural information available for any aminoglycoside-specific RND pump, here we describe cryo-electron microscopy (cryo-EM) structures of AcrD in the absence and presence of bound gentamicin. These structures provide new information about the RND superfamily of efflux pumps, specifically, that three negatively charged residues central to the aminoglycoside-binding site are located within the ceiling of the central cavity of the AcrD trimer. Thus, it is likely that AcrD is capable of picking up aminoglycosides via this central cavity. Through the combination of cryo-EM structural determination, mutagenesis analysis, and molecular simulation, we show that charged residues are critically important for this pump to shuttle drugs directly from the central cavity to the funnel of the AcrD trimer for extrusion. Here, we report cryo-EM structures of the AcrD aminoglycoside efflux pump in the absence and presence of bound gentamicin, posing the possibility that this pump is capable of capturing aminoglycosides from the central cavity of the AcrD trimer. The results indicate that AcrD utilizes charged residues to bind and export drugs, mediating resistance to these antibiotics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
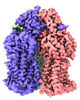
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28857.map.gz emd_28857.map.gz | 157.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28857-v30.xml emd-28857-v30.xml emd-28857.xml emd-28857.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28857_fsc.xml emd_28857_fsc.xml | 11.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28857.png emd_28857.png | 172.7 KB | ||
| Filedesc metadata |  emd-28857.cif.gz emd-28857.cif.gz | 6.5 KB | ||
| その他 |  emd_28857_additional_1.map.gz emd_28857_additional_1.map.gz emd_28857_half_map_1.map.gz emd_28857_half_map_1.map.gz emd_28857_half_map_2.map.gz emd_28857_half_map_2.map.gz | 83.9 MB 154.2 MB 154.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28857 http://ftp.pdbj.org/pub/emdb/structures/EMD-28857 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28857 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28857 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28857_validation.pdf.gz emd_28857_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28857_full_validation.pdf.gz emd_28857_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_28857_validation.xml.gz emd_28857_validation.xml.gz | 20.6 KB | 表示 | |
| CIF形式データ |  emd_28857_validation.cif.gz emd_28857_validation.cif.gz | 26.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28857 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28857 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28857 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28857 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28857.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28857.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||
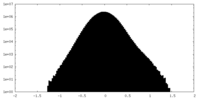
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_28857_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
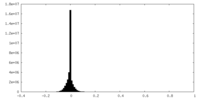
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28857_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
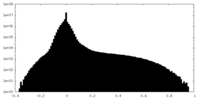
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28857_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
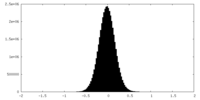
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AcrD
| 全体 | 名称: AcrD |
|---|---|
| 要素 |
|
-超分子 #1: AcrD
| 超分子 | 名称: AcrD / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Efflux pump membrane transporter
| 分子 | 名称: Efflux pump membrane transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 113.139891 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MANFFIDRPI FAWVLAILLC LTGTLAIFSL PVEQYPDLAP PNVRVTANYP GASAQTLENT VTQVIEQNMT GLDNLMYMSS QSSGTGQAS VTLSFKAGTD PDEAVQQVQN QLQSAMRKLP QAVQNQGVTV RKTGDTNILT IAFVSTDGSM DKQDIADYVA S NIQDPLSR ...文字列: MANFFIDRPI FAWVLAILLC LTGTLAIFSL PVEQYPDLAP PNVRVTANYP GASAQTLENT VTQVIEQNMT GLDNLMYMSS QSSGTGQAS VTLSFKAGTD PDEAVQQVQN QLQSAMRKLP QAVQNQGVTV RKTGDTNILT IAFVSTDGSM DKQDIADYVA S NIQDPLSR VNGVGDIDAY GSQYSMRIWL DPAKLNSFQM TAKDVTDAIE SQNAQIAVGQ LGGTPSVDKQ ALNATINAQS LL QTPEQFR DITLRVNQDG SEVRLGDVAT VEMGAEKYDY LSRFNGKPAS GLGVKLASGA NEMATAELVL NRLDELAQYF PHG LEYKVA YETTSFVKAS IEDVVKTLLE AIALVFLVMY LFLQNFRATL IPTIAVPVVL MGTFSVLYAF GYSVNTLTMF AMVL AIGLL VDDAIVVVEN VERIMSEEGL TPREATRKSM GQIQGALVGI AMVLSAVFVP MAFFGGTTGA IYRQFSITIV AAMVL SVLV AMILTPALCA TLLKPLKKGE HHGQKGFFAW FNQMFNRNAE RYEKGVAKIL HRSLRWIVIY VLLLGGMVFL FLRLPT SFL PLEDRGMFTT SVQLPSGSTQ QQTLKVVEQI EKYYFTHEKD NIMSVFATVG SGPGGNGQNV ARMFIRLKDW SERDSKT GT SFAIIERATK AFNQIKEARV IASSPPAISG LGSSAGFDME LQDHAGAGHD ALMAARNQLL ALAAENPELT RVRHNGLD D SPQLQIDIDQ RKAQALGVAI DDINDTLQTA WGSSYVNDFM DRGRVKKVYV QAAAPYRMLP DDINLWYVRN KDGGMVPFS AFATSRWETG SPRLERYNGY SAVEIVGEAA PGVSTGTAMD IMESLVKQLP NGFGLEWTAM SYQERLSGAQ APALYAISLL VVFLCLAAL YESWSVPFSV MLVVPLGVIG ALLATWMRGL ENDVYFQVGL LTVIGLSAKN AILIVEFANE MNQKGHDLFE A TLHACRQR LRPILMTSLA FIFGVLPMAT STGAGSGGQH AVGTGVMGGM ISATILAIYF VPLFFVLVRR RFPLKPRPE UniProtKB: Efflux pump membrane transporter |
-分子 #2: (2R,3R,4R,5R)-2-((1S,2S,3R,4S,6R)-4,6-DIAMINO-3-((2R,3R,6S)-3-AMI...
| 分子 | 名称: (2R,3R,4R,5R)-2-((1S,2S,3R,4S,6R)-4,6-DIAMINO-3-((2R,3R,6S)-3-AMINO-6-(AMINOMETHYL)-TETRAHYDRO-2H-PYRAN-2-YLOXY)-2-HYDR OXYCYCLOHEXYLOXY)-5-METHYL-4-(METHYLAMINO)-TETRAHYDRO-2H-PYRAN-3,5-DIOL タイプ: ligand / ID: 2 / コピー数: 1 / 式: LLL |
|---|---|
| 分子量 | 理論値: 449.542 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 35.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X