+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | P53 monomer structure | ||||||||||||
 マップデータ マップデータ | P53 monomer structure | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  cancer (悪性腫瘍) / cancer (悪性腫瘍) /  tumor suppressor (がん抑制遺伝子) / tumor suppressor (がん抑制遺伝子) /  cell cycle (細胞周期) / cell cycle (細胞周期) /  apoptosis (アポトーシス) / apoptosis (アポトーシス) /  DNA repair (DNA修復) / DNA repair (DNA修復) /  ANTITUMOR PROTEIN ANTITUMOR PROTEIN | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria / negative regulation of pentose-phosphate shunt / ATP-dependent DNA/DNA annealing activity ...Loss of function of TP53 in cancer due to loss of tetramerization ability / Regulation of TP53 Expression / signal transduction by p53 class mediator / negative regulation of G1 to G0 transition / negative regulation of glucose catabolic process to lactate via pyruvate / Transcriptional activation of cell cycle inhibitor p21 / regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Activation of NOXA and translocation to mitochondria / negative regulation of pentose-phosphate shunt / ATP-dependent DNA/DNA annealing activity / negative regulation of helicase activity / regulation of cell cycle G2/M phase transition / intrinsic apoptotic signaling pathway in response to hypoxia / regulation of fibroblast apoptotic process / oxidative stress-induced premature senescence / oligodendrocyte apoptotic process / negative regulation of miRNA processing / positive regulation of thymocyte apoptotic process / glucose catabolic process to lactate via pyruvate /  regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death / regulation of tissue remodeling / positive regulation of mitochondrial membrane permeability / negative regulation of mitophagy / positive regulation of programmed necrotic cell death /  転写 (生物学) / bone marrow development / circadian behavior / histone deacetylase regulator activity / regulation of mitochondrial membrane permeability involved in apoptotic process / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / negative regulation of neuroblast proliferation / Regulation of TP53 Activity through Association with Co-factors / mitochondrial DNA repair / T cell lineage commitment / positive regulation of execution phase of apoptosis / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / positive regulation of cardiac muscle cell apoptotic process / TP53 Regulates Transcription of Caspase Activators and Caspases / T cell proliferation involved in immune response / cardiac septum morphogenesis / entrainment of circadian clock by photoperiod / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding / 転写 (生物学) / bone marrow development / circadian behavior / histone deacetylase regulator activity / regulation of mitochondrial membrane permeability involved in apoptotic process / RUNX3 regulates CDKN1A transcription / germ cell nucleus / regulation of DNA damage response, signal transduction by p53 class mediator / TP53 regulates transcription of additional cell cycle genes whose exact role in the p53 pathway remain uncertain / TP53 Regulates Transcription of Death Receptors and Ligands / Activation of PUMA and translocation to mitochondria / DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / negative regulation of glial cell proliferation / Formation of Senescence-Associated Heterochromatin Foci (SAHF) / negative regulation of neuroblast proliferation / Regulation of TP53 Activity through Association with Co-factors / mitochondrial DNA repair / T cell lineage commitment / positive regulation of execution phase of apoptosis / negative regulation of DNA replication / ER overload response / B cell lineage commitment / thymocyte apoptotic process / TP53 regulates transcription of several additional cell death genes whose specific roles in p53-dependent apoptosis remain uncertain / positive regulation of cardiac muscle cell apoptotic process / TP53 Regulates Transcription of Caspase Activators and Caspases / T cell proliferation involved in immune response / cardiac septum morphogenesis / entrainment of circadian clock by photoperiod / PI5P Regulates TP53 Acetylation / Association of TriC/CCT with target proteins during biosynthesis / Zygotic genome activation (ZGA) / necroptotic process / rRNA transcription / positive regulation of release of cytochrome c from mitochondria / TP53 Regulates Transcription of Genes Involved in Cytochrome C Release / TFIID-class transcription factor complex binding /  マイトファジー / SUMOylation of transcription factors / negative regulation of telomere maintenance via telomerase / general transcription initiation factor binding / intrinsic apoptotic signaling pathway by p53 class mediator / Transcriptional Regulation by VENTX / response to X-ray / DNA damage response, signal transduction by p53 class mediator / マイトファジー / SUMOylation of transcription factors / negative regulation of telomere maintenance via telomerase / general transcription initiation factor binding / intrinsic apoptotic signaling pathway by p53 class mediator / Transcriptional Regulation by VENTX / response to X-ray / DNA damage response, signal transduction by p53 class mediator /  ヘイフリック限界 / chromosome organization / neuroblast proliferation / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / cellular response to UV-C / : / hematopoietic stem cell differentiation / negative regulation of reactive oxygen species metabolic process / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / glial cell proliferation / embryonic organ development / positive regulation of RNA polymerase II transcription preinitiation complex assembly / ヘイフリック限界 / chromosome organization / neuroblast proliferation / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / cellular response to UV-C / : / hematopoietic stem cell differentiation / negative regulation of reactive oxygen species metabolic process / intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator / glial cell proliferation / embryonic organ development / positive regulation of RNA polymerase II transcription preinitiation complex assembly /  Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / cellular response to actinomycin D / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest / Pyroptosis / cis-regulatory region sequence-specific DNA binding / hematopoietic progenitor cell differentiation / cellular response to glucose starvation / cellular response to actinomycin D / TP53 Regulates Transcription of Genes Involved in G1 Cell Cycle Arrest /  somitogenesis / type II interferon-mediated signaling pathway / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding / somitogenesis / type II interferon-mediated signaling pathway / positive regulation of intrinsic apoptotic signaling pathway / negative regulation of stem cell proliferation / core promoter sequence-specific DNA binding /  gastrulation / negative regulation of fibroblast proliferation / MDM2/MDM4 family protein binding / cardiac muscle cell apoptotic process / transcription initiation-coupled chromatin remodeling / 14-3-3 protein binding / mitotic G1 DNA damage checkpoint signaling / Regulation of TP53 Activity through Acetylation / response to salt stress gastrulation / negative regulation of fibroblast proliferation / MDM2/MDM4 family protein binding / cardiac muscle cell apoptotic process / transcription initiation-coupled chromatin remodeling / 14-3-3 protein binding / mitotic G1 DNA damage checkpoint signaling / Regulation of TP53 Activity through Acetylation / response to salt stress類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 5.0 Å クライオ電子顕微鏡法 / 解像度: 5.0 Å | ||||||||||||
 データ登録者 データ登録者 | Solares M / Kelly DF | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Chembiochem / 年: 2022 ジャーナル: Chembiochem / 年: 2022タイトル: High-Resolution Imaging of Human Cancer Proteins Using Microprocessor Materials. 著者: Maria J Solares / G M Jonaid / William Y Luqiu / Samantha Berry / Janki Khadela / Yanping Liang / Madison C Evans / Kevin J Pridham / William J Dearnaley / Zhi Sheng / Deborah F Kelly /  要旨: Mutations in tumor suppressor genes, such as Tumor Protein 53 (TP53), are heavily implicated in aggressive cancers giving rise to gain- and loss-of-function phenotypes. While individual domains of ...Mutations in tumor suppressor genes, such as Tumor Protein 53 (TP53), are heavily implicated in aggressive cancers giving rise to gain- and loss-of-function phenotypes. While individual domains of the p53 protein have been studied extensively, structural information for full-length p53 remains incomplete. Functionalized microprocessor chips (microchips) with properties amenable to electron microscopy permitted us to visualize complete p53 assemblies for the first time. The new structures revealed p53 in an inactive dimeric state independent of DNA binding. Residues located at the protein-protein interface corresponded with modification sites in cancer-related hot spots. Changes in these regions may amplify the toxic effects of clinical mutations. Taken together, these results contribute advances in technology and imaging approaches to decode native protein models in different states of activation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28817.map.gz emd_28817.map.gz | 161.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28817-v30.xml emd-28817-v30.xml emd-28817.xml emd-28817.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28817.png emd_28817.png | 144.2 KB | ||
| Filedesc metadata |  emd-28817.cif.gz emd-28817.cif.gz | 6 KB | ||
| その他 |  emd_28817_half_map_1.map.gz emd_28817_half_map_1.map.gz emd_28817_half_map_2.map.gz emd_28817_half_map_2.map.gz | 192.5 KB 157.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28817 http://ftp.pdbj.org/pub/emdb/structures/EMD-28817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28817 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8f2iMC  8f2hC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28817.map.gz / 形式: CCP4 / 大きさ: 181.6 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28817.map.gz / 形式: CCP4 / 大きさ: 181.6 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | P53 monomer structure | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.3 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half Map 1
| ファイル | emd_28817_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
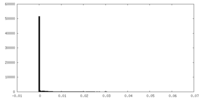
| 密度ヒストグラム |
-ハーフマップ: Half Map 2
| ファイル | emd_28817_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : P53 monomer isolated from U87-MG cells
| 全体 | 名称: P53 monomer isolated from U87-MG cells |
|---|---|
| 要素 |
|
-超分子 #1: P53 monomer isolated from U87-MG cells
| 超分子 | 名称: P53 monomer isolated from U87-MG cells / タイプ: cell / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Native protein, wild-type with no tags |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 器官: Brain / 組織: Brain tumor Homo sapiens (ヒト) / 器官: Brain / 組織: Brain tumor |
-分子 #1: Cellular tumor antigen p53
| 分子 | 名称: Cellular tumor antigen p53 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 器官: Brain tumor / 組織: Brain Homo sapiens (ヒト) / 器官: Brain tumor / 組織: Brain |
| 分子量 | 理論値: 43.711176 KDa |
| 配列 | 文字列: MEEPQSDPSV EPPLSQETFS DLWKLLPENN VLSPLPSQAM DDLMLSPDDI EQWFTEDPGP DEAPRMPEAA PPVAPAPAAP TPAAPAPAP SWPLSSSVPS QKTYQGSYGF RLGFLHSGTA KSVTCTYSPA LNKMFCQLAK TCPVQLWVDS TPPPGTRVRA M AIYKQSQH ...文字列: MEEPQSDPSV EPPLSQETFS DLWKLLPENN VLSPLPSQAM DDLMLSPDDI EQWFTEDPGP DEAPRMPEAA PPVAPAPAAP TPAAPAPAP SWPLSSSVPS QKTYQGSYGF RLGFLHSGTA KSVTCTYSPA LNKMFCQLAK TCPVQLWVDS TPPPGTRVRA M AIYKQSQH MTEVVRRCPH HERCSDSDGL APPQHLIRVE GNLRVEYLDD RNTFRHSVVV PYEPPEVGSD CTTIHYNYMC NS SCMGGMN RRPILTIITL EDSSGNLLGR NSFEVRVCAC PGRDRRTEEE NLRKKGEPHH ELPPGSTKRA LPNNTSSSPQ PKK KPLDGE YFTLQIRGRE RFEMFRELNE ALELKDAQAG KEPGGSRAHS SHLKSKKGQS TSRHKKLMFK TEGPDSD UniProtKB:  P53遺伝子 P53遺伝子 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM HEPES (pH 7.5), 140 mM NaCl, 2 mM MgCl2, 2 mM CaCl2, 5 mM imidazole |
| グリッド | モデル: Homemade / 材質: SILICON NITRIDE 詳細: Cleaned microchips were coated with 25% Ni-NTA-containing lipid monolayers. Aliquots (2 microliters) of p53 fractions were added to the Ni-NTA-coated microchips and incubated for 2 minute at room temperature. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK III 詳細: The microchip samples were loaded into a FEI Mark III Vitrobot and flash-frozen into liquid ethane.. |
| 詳細 | Sample was placed on Silicon nitride chips coated with Ni-NTA layers |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS F200C |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 142000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 142000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / 撮影したグリッド数: 10 / 実像数: 300 / 平均露光時間: 1.0 sec. / 平均電子線量: 5.0 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 8000 |
|---|---|
| 初期モデル | モデルのタイプ: NONE |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION |
| 最終 3次元分類 | クラス数: 1 / 平均メンバー数/クラス: 8000 / ソフトウェア - 名称: RELION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION / ソフトウェア - 名称: RELION |
| 最終 再構成 | 使用したクラス数: 1 / 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 5.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 詳細: validated in Rmeasure / 使用した粒子像数: 8000 |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 50 |
|---|---|
| 得られたモデル |  PDB-8f2i: |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X