+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01 FAB | |||||||||
 マップデータ マップデータ | main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | broadly neutralizing antibody / fusion peptide / HIV-1 / glycoprotein / viral protein / FP-targeting vaccines / VIRAL PROTEIN- IMMUNE SYSTEM complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Pletnev S / Kwong P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Antibody-directed evolution reveals a mechanism for enhanced neutralization at the HIV-1 fusion peptide site. 著者: Bailey B Banach / Sergei Pletnev / Adam S Olia / Kai Xu / Baoshan Zhang / Reda Rawi / Tatsiana Bylund / Nicole A Doria-Rose / Thuy Duong Nguyen / Ahmed S Fahad / Myungjin Lee / Bob C Lin / ...著者: Bailey B Banach / Sergei Pletnev / Adam S Olia / Kai Xu / Baoshan Zhang / Reda Rawi / Tatsiana Bylund / Nicole A Doria-Rose / Thuy Duong Nguyen / Ahmed S Fahad / Myungjin Lee / Bob C Lin / Tracy Liu / Mark K Louder / Bharat Madan / Krisha McKee / Sijy O'Dell / Mallika Sastry / Arne Schön / Natalie Bui / Chen-Hsiang Shen / Jacy R Wolfe / Gwo-Yu Chuang / John R Mascola / Peter D Kwong / Brandon J DeKosky /  要旨: The HIV-1 fusion peptide (FP) represents a promising vaccine target, but global FP sequence diversity among circulating strains has limited anti-FP antibodies to ~60% neutralization breadth. Here we ...The HIV-1 fusion peptide (FP) represents a promising vaccine target, but global FP sequence diversity among circulating strains has limited anti-FP antibodies to ~60% neutralization breadth. Here we evolve the FP-targeting antibody VRC34.01 in vitro to enhance FP-neutralization using site saturation mutagenesis and yeast display. Successive rounds of directed evolution by iterative selection of antibodies for binding to resistant HIV-1 strains establish a variant, VRC34.01_mm28, as a best-in-class antibody with 10-fold enhanced potency compared to the template antibody and ~80% breadth on a cross-clade 208-strain neutralization panel. Structural analyses demonstrate that the improved paratope expands the FP binding groove to accommodate diverse FP sequences of different lengths while also recognizing the HIV-1 Env backbone. These data reveal critical antibody features for enhanced neutralization breadth and potency against the FP site of vulnerability and accelerate clinical development of broad HIV-1 FP-targeting vaccines and therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28617.map.gz emd_28617.map.gz | 306.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28617-v30.xml emd-28617-v30.xml emd-28617.xml emd-28617.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28617_fsc.xml emd_28617_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28617.png emd_28617.png | 178.5 KB | ||
| マスクデータ |  emd_28617_msk_1.map emd_28617_msk_1.map | 325 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28617.cif.gz emd-28617.cif.gz | 7 KB | ||
| その他 |  emd_28617_half_map_1.map.gz emd_28617_half_map_1.map.gz emd_28617_half_map_2.map.gz emd_28617_half_map_2.map.gz | 301.7 MB 301.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28617 http://ftp.pdbj.org/pub/emdb/structures/EMD-28617 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28617 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28617 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28617_validation.pdf.gz emd_28617_validation.pdf.gz | 813.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28617_full_validation.pdf.gz emd_28617_full_validation.pdf.gz | 813.4 KB | 表示 | |
| XML形式データ |  emd_28617_validation.xml.gz emd_28617_validation.xml.gz | 23.7 KB | 表示 | |
| CIF形式データ |  emd_28617_validation.cif.gz emd_28617_validation.cif.gz | 30.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28617 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28617 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28617 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28617 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28617.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28617.map.gz / 形式: CCP4 / 大きさ: 325 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28617_msk_1.map emd_28617_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
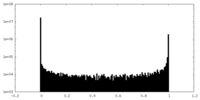
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_28617_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
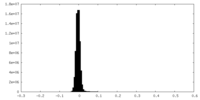
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_28617_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BG505 DS-SOSIP VRC34.01 FAB COMPLEX
| 全体 | 名称: BG505 DS-SOSIP VRC34.01 FAB COMPLEX |
|---|---|
| 要素 |
|
-超分子 #1: BG505 DS-SOSIP VRC34.01 FAB COMPLEX
| 超分子 | 名称: BG505 DS-SOSIP VRC34.01 FAB COMPLEX / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
-分子 #1: Envelope glycoprotein gp120
| 分子 | 名称: Envelope glycoprotein gp120 / タイプ: protein_or_peptide / ID: 1 / 詳細: BG505 DS-SOSIP GP120 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 54.086324 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPC VKLTPLCVTL QCTNVTNNIT DDMRGELKNC SFNMTTELRD KKQKVYSLFY RLDVVQINEN QGNRSNNSNK E YRLINCNT ...文字列: AENLWVTVYY GVPVWKDAET TLFCASDAKA YETEKHNVWA THACVPTDPN PQEIHLENVT EEFNMWKNNM VEQMHTDIIS LWDQSLKPC VKLTPLCVTL QCTNVTNNIT DDMRGELKNC SFNMTTELRD KKQKVYSLFY RLDVVQINEN QGNRSNNSNK E YRLINCNT SACTQACPKV SFEPIPIHYC APAGFAILKC KDKKFNGTGP CPSVSTVQCT HGIKPVVSTQ LLLNGSLAEE EV MIRSENI TNNAKNILVQ FNTPVQINCT RPNNNTRKSI RIGPGQAFYA TGDIIGDIRQ AHCNVSKATW NETLGKVVKQ LRK HFGNNT IIRFANSSGG DLEVTTHSFN CGGEFFYCNT SGLFNSTWIS NTSVQGSNST GSNDSITLPC RIKQIINMWQ RIGQ CMYAP PIQGVIRCVS NITGLILTRD GGSTNSTTET FRPGGGDMRD NWRSELYKYK VVKIEPLGVA PTRCKRRVVG RRRRR R UniProtKB: Envelope glycoprotein gp160 |
-分子 #2: Envelope glycoprotein gp41
| 分子 | 名称: Envelope glycoprotein gp41 / タイプ: protein_or_peptide / ID: 2 / 詳細: BG505 DS-SOSIP GP41 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 17.146482 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEA QQHLLKLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSG KLICCTNVPW NSSWSNRNLS EIWDNMTWLQ WDKEISNYTQ IIYGLLEESQ NQQEKNEQDL LALD UniProtKB: Envelope glycoprotein gp160 |
-分子 #3: VRC34.01 FAB variable heavy chain
| 分子 | 名称: VRC34.01 FAB variable heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.797615 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QEVLVQSGAE VKKPGASVKV SCRAFGYTFT GNALHWVRQA PGQGLEWLGW INPHSGDTTT SQKFQGRVYM TRDKSINTAF LDVTRLTSD DTGIYYCARD KYYGNEAVGM DVWGQGTSVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV T VSWNSGAL ...文字列: QEVLVQSGAE VKKPGASVKV SCRAFGYTFT GNALHWVRQA PGQGLEWLGW INPHSGDTTT SQKFQGRVYM TRDKSINTAF LDVTRLTSD DTGIYYCARD KYYGNEAVGM DVWGQGTSVT VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV T VSWNSGAL TSGVHTFPAV LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK KVEPK |
-分子 #4: VRC34.01 FAB variable light chain
| 分子 | 名称: VRC34.01 FAB variable light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.138766 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQLTQSPSF LSASVGDKVT ITCRASQGVR NELAWYQQKP GKAPNLLIYY ASTLQSGVPS RFSATGSGTH FTLTVSSLQP EDFATYFCQ HMSSYPLTFG GGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQLTQSPSF LSASVGDKVT ITCRASQGVR NELAWYQQKP GKAPNLLIYY ASTLQSGVPS RFSATGSGTH FTLTVSSLQP EDFATYFCQ HMSSYPLTFG GGTKVEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRG |
-分子 #8: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 8 / コピー数: 15 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: GOLD / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 8085 / 平均電子線量: 43.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8euu: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)