+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a delivery complex containing the SspB adaptor, an ssrA-tagged substrate, and the AAA+ ClpXP protease | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAA+ protease / ClpXP / SspB adaptor / HYDROLASE / CHAPERONE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報HslUV protease complex / endopeptidase Clp / endopeptidase Clp complex / positive regulation of programmed cell death / response to temperature stimulus / ATP-dependent peptidase activity / protein quality control for misfolded or incompletely synthesized proteins / proteasomal protein catabolic process / serine-type peptidase activity / proteolysis involved in protein catabolic process ...HslUV protease complex / endopeptidase Clp / endopeptidase Clp complex / positive regulation of programmed cell death / response to temperature stimulus / ATP-dependent peptidase activity / protein quality control for misfolded or incompletely synthesized proteins / proteasomal protein catabolic process / serine-type peptidase activity / proteolysis involved in protein catabolic process / ATP-dependent protein folding chaperone / response to radiation / serine-type endopeptidase inhibitor activity / unfolded protein binding / ATPase binding / peptidase activity / response to heat / protein dimerization activity / cell division / serine-type endopeptidase activity / ATP hydrolysis activity / proteolysis / extracellular space / zinc ion binding / ATP binding / identical protein binding / membrane / metal ion binding / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
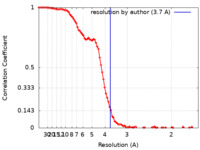
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Ghanbarpour A / Fei X / Davis JH / Sauer RT | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: The SspB adaptor drives structural changes in the AAA+ ClpXP protease during ssrA-tagged substrate delivery. 著者: Alireza Ghanbarpour / Xue Fei / Tania A Baker / Joseph H Davis / Robert T Sauer /  要旨: Energy-dependent protein degradation by the AAA+ ClpXP protease helps maintain protein homeostasis in bacteria and eukaryotic organelles of bacterial origin. In and many other proteobacteria, the ...Energy-dependent protein degradation by the AAA+ ClpXP protease helps maintain protein homeostasis in bacteria and eukaryotic organelles of bacterial origin. In and many other proteobacteria, the SspB adaptor assists ClpXP in degrading ssrA-tagged polypeptides produced as a consequence of tmRNA-mediated ribosome rescue. By tethering these incomplete ssrA-tagged proteins to ClpXP, SspB facilitates their efficient degradation at low substrate concentrations. How this process occurs structurally is unknown. Here, we present a cryo-EM structure of the SspB adaptor bound to a GFP-ssrA substrate and to ClpXP. This structure provides evidence for simultaneous contacts of SspB and ClpX with the ssrA tag within the tethering complex, allowing direct substrate handoff concomitant with the initiation of substrate translocation. Furthermore, our structure reveals that binding of the substrate·adaptor complex induces unexpected conformational changes within the spiral structure of the AAA+ ClpX hexamer and its interaction with the ClpP tetradecamer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28585.map.gz emd_28585.map.gz | 58 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28585-v30.xml emd-28585-v30.xml emd-28585.xml emd-28585.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28585_fsc.xml emd_28585_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28585.png emd_28585.png | 45.9 KB | ||
| マスクデータ |  emd_28585_msk_1.map emd_28585_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28585.cif.gz emd-28585.cif.gz | 6.7 KB | ||
| その他 |  emd_28585_half_map_1.map.gz emd_28585_half_map_1.map.gz emd_28585_half_map_2.map.gz emd_28585_half_map_2.map.gz | 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28585 http://ftp.pdbj.org/pub/emdb/structures/EMD-28585 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28585 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28585 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28585_validation.pdf.gz emd_28585_validation.pdf.gz | 933.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28585_full_validation.pdf.gz emd_28585_full_validation.pdf.gz | 933.3 KB | 表示 | |
| XML形式データ |  emd_28585_validation.xml.gz emd_28585_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_28585_validation.cif.gz emd_28585_validation.cif.gz | 20.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28585 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28585 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28585 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28585 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8et3MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28585.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28585.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28585_msk_1.map emd_28585_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28585_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_28585_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ClpXP AAA protease complex bound with SspB and GFP-ssrA
| 全体 | 名称: ClpXP AAA protease complex bound with SspB and GFP-ssrA |
|---|---|
| 要素 |
|
-超分子 #1: ClpXP AAA protease complex bound with SspB and GFP-ssrA
| 超分子 | 名称: ClpXP AAA protease complex bound with SspB and GFP-ssrA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: ATP-dependent Clp protease ATP-binding subunit ClpX
| 分子 | 名称: ATP-dependent Clp protease ATP-binding subunit ClpX / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 46.414848 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTDKRKDGSG KLLYCSFCGK SQHEVRKLIA GPSVYICDEC VDLCNDIIRE EIKEVAPHRE RSALPTPHEI RNHLDDYVIG QEQAKKVLA VAVYNHYKRL RNGDTSNGVE LGKSNILLIG PTGSGKTLLA ETLARLLDVP FTMADATTLT EAGYVGEDVE N IIQKLLQK ...文字列: MTDKRKDGSG KLLYCSFCGK SQHEVRKLIA GPSVYICDEC VDLCNDIIRE EIKEVAPHRE RSALPTPHEI RNHLDDYVIG QEQAKKVLA VAVYNHYKRL RNGDTSNGVE LGKSNILLIG PTGSGKTLLA ETLARLLDVP FTMADATTLT EAGYVGEDVE N IIQKLLQK CDYDVQKAQR GIVYIDEIDK ISRKSDNPSI TRDVSGEGVQ QALLKLIEGT VAAVPPQGGR KHPQQEFLQV DT SKILFIC GGAFAGLDKV ISHRVETGSG IGFGATVKAK SDKASEGELL AQVEPEDLIK FGLIPEFIGR LPVVATLNEL SEE ALIQIL KEPKNALTKQ YQALFNLEGV DLEFRDEALD AIAKKAMARK TGARGLRSIV EAALLDTMYD LPSMEDVEKV VIDE SVIDG QSEPLLIYGK PEAQQASGE UniProtKB: ATP-dependent Clp protease ATP-binding subunit ClpX |
-分子 #2: ATP-dependent Clp protease proteolytic subunit
| 分子 | 名称: ATP-dependent Clp protease proteolytic subunit / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO / EC番号: endopeptidase Clp |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 23.468869 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: LVPMVIEQTS RGERSFDIYS RLLKERVIFL TGQVEDHMAN LIVAQMLFLE AENPEKDIYL YINSPGGVIT AGMSIYDTMQ FIKPDVSTI CMGQAASMGA FLLTAGAKGK RFCLPNSRVM IHQPLGGYQG QATDIEIHAR EILKVKGRMN ELMALHTGQS L EQIERDTE ...文字列: LVPMVIEQTS RGERSFDIYS RLLKERVIFL TGQVEDHMAN LIVAQMLFLE AENPEKDIYL YINSPGGVIT AGMSIYDTMQ FIKPDVSTI CMGQAASMGA FLLTAGAKGK RFCLPNSRVM IHQPLGGYQG QATDIEIHAR EILKVKGRMN ELMALHTGQS L EQIERDTE RDRFLSAPEA VEYGLVDSIL THRNENLYFQ SLEHHHHHH UniProtKB: ATP-dependent Clp protease proteolytic subunit |
-分子 #3: Green fluorescent protein
| 分子 | 名称: Green fluorescent protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 30.855428 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH DYDIPTTENL YFQGSRKGEE LFTGVVPILV ELDGDVNGHK FSVSGEGEGD ATYGKLTLKF ICTTGKLPVP WPTLVTTFG YGVQCFARYP DHMKQHDFFK SAMPEGYVQE RTIFFKDDGN YKTRAEVKFE GDTLVNRIEL KGIDFKEDGN I LGHKLEYN ...文字列: MGSSHHHHHH DYDIPTTENL YFQGSRKGEE LFTGVVPILV ELDGDVNGHK FSVSGEGEGD ATYGKLTLKF ICTTGKLPVP WPTLVTTFG YGVQCFARYP DHMKQHDFFK SAMPEGYVQE RTIFFKDDGN YKTRAEVKFE GDTLVNRIEL KGIDFKEDGN I LGHKLEYN YNSHNVYIMA DKQKNGIKVN FKIRHNIEDG SVQLADHYQQ NTPIGDGPVL LPDNHYLSTQ SALSKDPNEK RD HMVLLEF VTAAGITHGM DELYKAANDE NYALAA UniProtKB: Serpin |
-分子 #4: Stringent starvation protein B
| 分子 | 名称: Stringent starvation protein B / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 18.279352 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDLSQLTPRR PYLLRAFYEW LLDNQLTPHL VVDVTLPGVQ VPMEYARDGQ IVLNIAPRAV GNLELANDEV RFNARFGGIP RQVSVPLAA VLAIYARENG AGTMFEPEAA YDEDTSIMND EEASADNETV MSVIDGDKPD HDDDTHPDDE PPQPPRGGRP A LRVVK UniProtKB: Stringent starvation protein B |
-分子 #5: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 4 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-分子 #6: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 75.98 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X