登録情報 データベース : EMDB / ID : EMD-28332タイトル Cryo-EM structure of human HSP90B in the closed state Cryo-EM structure of human HSP90B-AIPL1 complex 複合体 : HSP90B/AIPL1 complexタンパク質・ペプチド : Heat shock protein HSP 90-betaタンパク質・ペプチド : Aryl-hydrocarbon-interacting protein-like 1リガンド : PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTERリガンド : MAGNESIUM ION / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / Mus musculus (ハツカネズミ)手法 / / 解像度 : 3.9 Å Srivastava D / Artemyev NO 資金援助 Organization Grant number 国 National Institutes of Health/National Eye Institute (NIH/NEI) R01 EY-10843
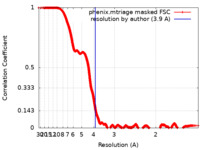
ジャーナル : Structure / 年 : 2023タイトル : Unique interface and dynamics of the complex of HSP90 with a specialized cochaperone AIPL1.著者 : Dhiraj Srivastava / Ravi P Yadav / Sneha Singh / Kimberly Boyd / Nikolai O Artemyev / 要旨 : Photoreceptor phosphodiesterase PDE6 is central for visual signal transduction. Maturation of PDE6 depends on a specialized chaperone complex of HSP90 with aryl hydrocarbon receptor-interacting ... Photoreceptor phosphodiesterase PDE6 is central for visual signal transduction. Maturation of PDE6 depends on a specialized chaperone complex of HSP90 with aryl hydrocarbon receptor-interacting protein-like 1 (AIPL1). Disruption of PDE6 maturation underlies a severe form of retina degeneration. Here, we report a 3.9 Å cryoelectron microscopy (cryo-EM) structure of the complex of HSP90 with AIPL1. This structure reveals a unique interaction of the FK506-binding protein (FKBP)-like domain of AIPL1 with HSP90 at its dimer interface. Unusually, the N terminus AIPL1 inserts into the HSP90 lumen in a manner that was observed previously for HSP90 clients. Deletion of the 7 N-terminal residues of AIPL1 decreased its ability to cochaperone PDE6. Multi-body refinement of the cryo-EM data indicated large swing-like movements of AIPL1-FKBP. Modeling the complex of HSP90 with AIPL1 using crosslinking constraints indicated proximity of the mobile tetratricopeptide repeat (TPR) domain with the C-terminal domain of HSP90. Our study establishes a framework for future structural studies of PDE6 maturation. 履歴 登録 2022年10月2日 - ヘッダ(付随情報) 公開 2023年1月25日 - マップ公開 2023年1月25日 - 更新 2024年6月19日 - 現状 2024年6月19日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報
 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) /
Homo sapiens (ヒト) / 
 データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Structure / 年: 2023
ジャーナル: Structure / 年: 2023
 構造の表示
構造の表示 ダウンロードとリンク
ダウンロードとリンク emd_28332.map.gz
emd_28332.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-28332-v30.xml
emd-28332-v30.xml emd-28332.xml
emd-28332.xml EMDBヘッダ
EMDBヘッダ emd_28332_fsc.xml
emd_28332_fsc.xml FSCデータファイル
FSCデータファイル emd_28332.png
emd_28332.png emd_28332_msk_1.map
emd_28332_msk_1.map マスクマップ
マスクマップ emd-28332.cif.gz
emd-28332.cif.gz emd_28332_half_map_1.map.gz
emd_28332_half_map_1.map.gz emd_28332_half_map_2.map.gz
emd_28332_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-28332
http://ftp.pdbj.org/pub/emdb/structures/EMD-28332 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28332
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28332 emd_28332_validation.pdf.gz
emd_28332_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_28332_full_validation.pdf.gz
emd_28332_full_validation.pdf.gz emd_28332_validation.xml.gz
emd_28332_validation.xml.gz emd_28332_validation.cif.gz
emd_28332_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28332
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28332 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28332
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28332

 F&H 検索
F&H 検索 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_28332.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_28332.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_28332_msk_1.map
emd_28332_msk_1.map 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト) Homo sapiens (ヒト)
Homo sapiens (ヒト)



 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)