+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tail tip structure of Staphylococcus phage Andhra | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | phage tail / VIRUS | |||||||||
| 機能・相同性 | CHAP domain profile. / CHAP domain / CHAP domain / Peptidase 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Staphylococcus phage Andhra (ファージ) Staphylococcus phage Andhra (ファージ) | |||||||||
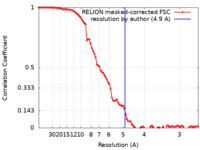
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.9 Å | |||||||||
 データ登録者 データ登録者 | Kizziah JL / Hawkins NC / Dokland T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Structure and host specificity of bacteriophage Andhra. 著者: N'Toia C Hawkins / James L Kizziah / Asma Hatoum-Aslan / Terje Dokland /  要旨: is an opportunistic pathogen of the human skin, often associated with infections of implanted medical devices. Staphylococcal picoviruses are a group of strictly lytic, short-tailed bacteriophages ... is an opportunistic pathogen of the human skin, often associated with infections of implanted medical devices. Staphylococcal picoviruses are a group of strictly lytic, short-tailed bacteriophages with compact genomes that are attractive candidates for therapeutic use. Here, we report the structure of the complete virion of -infecting phage Andhra, determined using high-resolution cryo-electron microscopy, allowing atomic modeling of 11 capsid and tail proteins. The capsid is a = 4 icosahedron containing a unique stabilizing capsid lining protein. The tail includes 12 trimers of a unique receptor binding protein (RBP), a lytic protein that also serves to anchor the RBPs to the tail stem, and a hexameric tail knob that acts as a gatekeeper for DNA ejection. Using structure prediction with AlphaFold, we identified the two proteins that comprise the tail tip heterooctamer. Our findings elucidate critical features for virion assembly, host recognition, and penetration. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28176.map.gz emd_28176.map.gz | 59.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28176-v30.xml emd-28176-v30.xml emd-28176.xml emd-28176.xml | 15.2 KB 15.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28176_fsc.xml emd_28176_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28176.png emd_28176.png | 77.5 KB | ||
| Filedesc metadata |  emd-28176.cif.gz emd-28176.cif.gz | 5.5 KB | ||
| その他 |  emd_28176_half_map_1.map.gz emd_28176_half_map_1.map.gz emd_28176_half_map_2.map.gz emd_28176_half_map_2.map.gz | 49.8 MB 49.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28176 http://ftp.pdbj.org/pub/emdb/structures/EMD-28176 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28176 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28176 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28176_validation.pdf.gz emd_28176_validation.pdf.gz | 938.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28176_full_validation.pdf.gz emd_28176_full_validation.pdf.gz | 938.3 KB | 表示 | |
| XML形式データ |  emd_28176_validation.xml.gz emd_28176_validation.xml.gz | 14.7 KB | 表示 | |
| CIF形式データ |  emd_28176_validation.cif.gz emd_28176_validation.cif.gz | 20.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28176 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28176 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28176 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28176 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28176.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28176.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.328 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28176_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28176_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Staphylococcus phage Andhra
| 全体 | 名称:  Staphylococcus phage Andhra (ファージ) Staphylococcus phage Andhra (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Staphylococcus phage Andhra
| 超分子 | 名称: Staphylococcus phage Andhra / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #2, #1 / NCBI-ID: 1958907 / 生物種: Staphylococcus phage Andhra / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-分子 #1: gp10, tail tip lysin (spike)
| 分子 | 名称: gp10, tail tip lysin (spike) / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage Andhra (ファージ) Staphylococcus phage Andhra (ファージ) |
| 分子量 | 理論値: 51.675402 KDa |
| 組換発現 | 生物種:  Staphylococcus phage Andhra (ファージ) Staphylococcus phage Andhra (ファージ) |
| 配列 | 文字列: MNDKEKIDKF IHSNLNDDFG LSVDDLVGKV KGIGRFSAWC GNSSTKIKQV LNAVKSIGVS PALFAAYEKN EGYNGSWGWL NHTSPQGNY LTDAQFVARK LVSQSRQAGT PSWIDAGNPV DFVPASVKRK GNYDFSHNMK NGKVGRAYIP LTAAATWAAY Y PEGLQASY ...文字列: MNDKEKIDKF IHSNLNDDFG LSVDDLVGKV KGIGRFSAWC GNSSTKIKQV LNAVKSIGVS PALFAAYEKN EGYNGSWGWL NHTSPQGNY LTDAQFVARK LVSQSRQAGT PSWIDAGNPV DFVPASVKRK GNYDFSHNMK NGKVGRAYIP LTAAATWAAY Y PEGLQASY NRVQNYGNPF LDAANTILGW GGKIDGKGGS SSGSSSSSSG TSGGLDVVAR AFEEFLKKLQ DSMQWDLHSI GT DKFFSNQ MFTITKTYNN TYRLNMNQKL LDEMKDLISR IDGGSGNDTG ADDSDGDHGG KAGKSVAPNG KSGRKIGGNW TYS NLPQKY KDAIEVPKFD PKYLAGSPFV NTGDTGQCTE LTWAYMHQIW GKRQPAWDNQ VTNGQRVWVV YRNQGARVTH RPTV GYGFS SKPNYLQAML PGVGHTGVVV AVFKDGSFLT ANYNVPPYWA PSRVVEYALI DGVPENAGDN IMFFSGIK UniProtKB: Peptidase |
-分子 #2: gp1, tail tip protein
| 分子 | 名称: gp1, tail tip protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Staphylococcus phage Andhra (ファージ) Staphylococcus phage Andhra (ファージ) |
| 分子量 | 理論値: 10.640743 KDa |
| 組換発現 | 生物種:  Staphylococcus phage Andhra (ファージ) Staphylococcus phage Andhra (ファージ) |
| 配列 | 文字列: VTNEKGQAYT EMLQLFNLLQ QWNDFYTAEN ANNLLVACQQ LLINYNEPVI KFINDENEDK SLLQYLAGDD GLAQWQFYKG FYNNYNVHI F |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 39.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X