+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of BIRC6/HtrA2-S306A | |||||||||
 マップデータ マップデータ | main map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ubiquitin / E3 ligase / Apoptosis / Autophagy / IAP / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報HtrA2 peptidase / pentacyclic triterpenoid metabolic process / negative regulation of mitophagy in response to mitochondrial depolarization / spongiotrophoblast layer development / positive regulation of cysteine-type endopeptidase activity involved in apoptotic signaling pathway / ceramide metabolic process / regulation of autophagy of mitochondrion / mitochondrial protein catabolic process / CD40 receptor complex / : ...HtrA2 peptidase / pentacyclic triterpenoid metabolic process / negative regulation of mitophagy in response to mitochondrial depolarization / spongiotrophoblast layer development / positive regulation of cysteine-type endopeptidase activity involved in apoptotic signaling pathway / ceramide metabolic process / regulation of autophagy of mitochondrion / mitochondrial protein catabolic process / CD40 receptor complex / : / labyrinthine layer development / ALK mutants bind TKIs / programmed cell death / positive regulation of extrinsic apoptotic signaling pathway in absence of ligand / Flemming body / serine-type endopeptidase complex / adult walking behavior / response to herbicide / positive regulation of protein targeting to mitochondrion / execution phase of apoptosis / microtubule organizing center / cysteine-type endopeptidase inhibitor activity / positive regulation of execution phase of apoptosis / ubiquitin conjugating enzyme activity / positive regulation of cysteine-type endopeptidase activity involved in apoptotic process / protein autoprocessing / negative regulation of cell cycle / regulation of multicellular organism growth / neuron development / cellular response to interferon-beta / negative regulation of oxidative stress-induced intrinsic apoptotic signaling pathway / cellular response to retinoic acid / forebrain development / Mitochondrial protein degradation / serine-type peptidase activity / regulation of cytokinesis / negative regulation of extrinsic apoptotic signaling pathway / mitochondrial membrane / protein catabolic process / RING-type E3 ubiquitin transferase / trans-Golgi network / mitochondrial intermembrane space / cytoplasmic side of plasma membrane / cellular response to growth factor stimulus / spindle pole / ubiquitin-protein transferase activity / intrinsic apoptotic signaling pathway in response to DNA damage / Signaling by ALK fusions and activated point mutants / unfolded protein binding / peptidase activity / cellular response to heat / regulation of cell population proliferation / cellular response to oxidative stress / midbody / neuron apoptotic process / negative regulation of neuron apoptotic process / cell population proliferation / cytoskeleton / protein ubiquitination / endosome / cell cycle / positive regulation of apoptotic process / cell division / protein phosphorylation / serine-type endopeptidase activity / centrosome / apoptotic process / positive regulation of cell population proliferation / endoplasmic reticulum membrane / chromatin / negative regulation of apoptotic process / endoplasmic reticulum / mitochondrion / proteolysis / identical protein binding / membrane / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.21 Å | |||||||||
 データ登録者 データ登録者 | Hunkeler M / Fischer ES | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Structures of BIRC6-client complexes provide a mechanism of SMAC-mediated release of caspases. 著者: Moritz Hunkeler / Cyrus Y Jin / Eric S Fischer /  要旨: Tight regulation of apoptosis is essential for metazoan development and prevents diseases such as cancer and neurodegeneration. Caspase activation is central to apoptosis, and inhibitor of apoptosis ...Tight regulation of apoptosis is essential for metazoan development and prevents diseases such as cancer and neurodegeneration. Caspase activation is central to apoptosis, and inhibitor of apoptosis proteins (IAPs) are the principal actors that restrain caspase activity and are therefore attractive therapeutic targets. IAPs, in turn, are regulated by mitochondria-derived proapoptotic factors such as SMAC and HTRA2. Through a series of cryo-electron microscopy structures of full-length human baculoviral IAP repeat-containing protein 6 (BIRC6) bound to SMAC, caspases, and HTRA2, we provide a molecular understanding for BIRC6-mediated caspase inhibition and its release by SMAC. The architecture of BIRC6, together with near-irreversible binding of SMAC, elucidates how the IAP inhibitor SMAC can effectively control a processive ubiquitin ligase to respond to apoptotic stimuli. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27841.map.gz emd_27841.map.gz | 157.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27841-v30.xml emd-27841-v30.xml emd-27841.xml emd-27841.xml | 27.5 KB 27.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27841_fsc.xml emd_27841_fsc.xml | 11.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27841.png emd_27841.png | 74.2 KB | ||
| マスクデータ |  emd_27841_msk_1.map emd_27841_msk_1.map emd_27841_msk_2.map emd_27841_msk_2.map | 166.4 MB 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-27841.cif.gz emd-27841.cif.gz | 9.8 KB | ||
| その他 |  emd_27841_half_map_1.map.gz emd_27841_half_map_1.map.gz emd_27841_half_map_2.map.gz emd_27841_half_map_2.map.gz | 154.6 MB 154.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27841 http://ftp.pdbj.org/pub/emdb/structures/EMD-27841 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27841 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27841 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27841_validation.pdf.gz emd_27841_validation.pdf.gz | 1.3 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27841_full_validation.pdf.gz emd_27841_full_validation.pdf.gz | 1.3 MB | 表示 | |
| XML形式データ |  emd_27841_validation.xml.gz emd_27841_validation.xml.gz | 20.5 KB | 表示 | |
| CIF形式データ |  emd_27841_validation.cif.gz emd_27841_validation.cif.gz | 26.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27841 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27841 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27841 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27841 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8e2kMC  8e2dC  8e2eC  8e2fC  8e2gC  8e2hC  8e2iC  8e2jC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27841.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27841.map.gz / 形式: CCP4 / 大きさ: 166.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | main map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27841_msk_1.map emd_27841_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
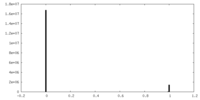
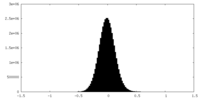
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_27841_msk_2.map emd_27841_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
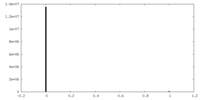
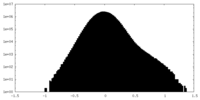
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 1
| ファイル | emd_27841_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
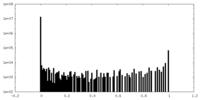
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_27841_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
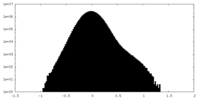
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Baculoviral IAP repeat-containing protein 6
| 全体 | 名称: Baculoviral IAP repeat-containing protein 6 |
|---|---|
| 要素 |
|
-超分子 #1: Baculoviral IAP repeat-containing protein 6
| 超分子 | 名称: Baculoviral IAP repeat-containing protein 6 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 1.174 MDa |
-分子 #1: Baculoviral IAP repeat-containing protein 6
| 分子 | 名称: Baculoviral IAP repeat-containing protein 6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: 合成酵素 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 535.317188 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGDYKDHDGD YKDHDIDYKD DDDKGGGSGG LEVLFQGPSR TMVTGGGAAP PGTVTEPLPS VIVLSAGRKM AAAAAAASGP GCSSAAGAG AAGVSEWLVL RDGCMHCDAD GLHSLSYHPA LNAILAVTSR GTIKVIDGTS GATLQASALS AKPGGQVKCQ Y ISAVDKVI ...文字列: MGDYKDHDGD YKDHDIDYKD DDDKGGGSGG LEVLFQGPSR TMVTGGGAAP PGTVTEPLPS VIVLSAGRKM AAAAAAASGP GCSSAAGAG AAGVSEWLVL RDGCMHCDAD GLHSLSYHPA LNAILAVTSR GTIKVIDGTS GATLQASALS AKPGGQVKCQ Y ISAVDKVI FVDDYAVGCR KDLNGILLLD TALQTPVSKQ DDVVQLELPV TEAQQLLSAC LEKVDISSTE GYDLFITQLK DG LKNTSHE TAANHKVAKW ATVTFHLPHH VLKSIASAIV NELKKINQNV AALPVASSVM DRLSYLLPSA RPELGVGPGR SVD RSLMYS EANRRETFTS WPHVGYRWAQ PDPMAQAGFY HQPASSGDDR AMCFTCSVCL VCWEPTDEPW SEHERHSPNC PFVK GEHTQ NVPLSVTLAT SPAQFPCTDG TDRISCFGSG SCPHFLAAAT KRGKICIWDV SKLMKVHLKF EINAYDPAIV QQLIL SGDP SSGVDSRRPT LAWLEDSSSC SDIPKLEGDS DDLLEDSDSE EHSRSDSVTG HTSQKEAMEV SLDITALSIL QQPEKL QWE IVANVLEDTV KDLEELGANP CLTNSKSEKT KEKHQEQHNI PFPCLLAGGL LTYKSPATSP ISSNSHRSLD GLSRTQG ES ISEQGSTDNE SCTNSELNSP LVRRTLPVLL LYSIKESDEK AGKIFSQMNN IMSKSLHDDG FTVPQIIEME LDSQEQLL L QDPPVTYIQQ FADAAANLTS PDSEKWNSVF PKPGTLVQCL RLPKFAEEEN LCIDSITPCA DGIHLLVGLR TCPVESLSA INQVEALNNL NKLNSALCNR RKGELESNLA VVNGANISVI QHESPADVQT PLIIQPEQRN VSGGYLVLYK MNYATRIVTL EEEPIKIQH IKDPQDTITS LILLPPDILD NREDDCEEPI EDMQLTSKNG FEREKTSDIS TLGHLVITTQ GGYVKILDLS N FEILAKVE PPKKEGTEEQ DTFVSVIYCS GTDRLCACTK GGELHFLQIG GTCDDIDEAD ILVDGSLSKG IEPSSEGSKP LS NPSSPGI SGVDLLVDQP FTLEILTSLV ELTRFETLTP RFSATVPPCW VEVQQEQQQR RHPQHLHQQH HGDAAQHTRT WKL QTDSNS WDEHVFELVL PKACMVGHVD FKFVLNSNIT NIPQIQVTLL KNKAPGLGKV NALNIEVEQN GKPSLVDLNE EMQH MDVEE SQCLRLCPFL EDHKEDILCG PVWLASGLDL SGHAGMLTLT SPKLVKGMAG GKYRSFLIHV KAVNERGTEE ICNGG MRPV VRLPSLKHQS NKGYSLASLL AKVAAGKEKS SNVKNENTSG TRKSENLRGC DLLQEVSVTI RRFKKTSISK ERVQRC AML QFSEFHEKLV NTLCRKTDDG QITEHAQSLV LDTLCWLAGV HSNGPGSSKE GNENLLSKTR KFLSDIVRVC FFEAGRS IA HKCARFLALC ISNGKCDPCQ PAFGPVLLKA LLDNMSFLPA ATTGGSVYWY FVLLNYVKDE DLAGCSTACA SLLTAVSR Q LQDRLTPMEA LLQTRYGLYS SPFDPVLFDL EMSGSSCKNV YNSSIGVQSD EIDLSDVLSG NGKVSSCTAA EGSFTSLTG LLEVEPLHFT CVSTSDGTRI ERDDAMSSFG VTPAVGGLSS GTVGEASTAL SSAAQVALQS LSHAMASAEQ QLQVLQEKQQ QLLKLQQQK AKLEAKLHQT TAAAAAAASA VGPVHNSVPS NPVAAPGFFI HPSDVIPPTP KTTPLFMTPP LTPPNEAVSV V INAELAQL FPGSVIDPPA VNLAAHNKNS NKSRMNPLGS GLALAISHAS HFLQPPPHQS IIIERMHSGA RRFVTLDFGR PI LLTDVLI PTCGDLASLS IDIWTLGEEV DGRRLVVATD ISTHSLILHD LIPPPVCRFM KITVIGRYGS TNARAKIPLG FYY GHTYIL PWESELKLMH DPLKGEGESA NQPEIDQHLA MMVALQEDIQ CRYNLACHRL ETLLQSIDLP PLNSANNAQY FLRK PDKAV EEDSRVFSAY QDCIQLQLQL NLAHNAVQRL KVALGASRKM LSETSNPEDL IQTSSTEQLR TIIRYLLDTL LSLLH ASNG HSVPAVLQST FHAQACEELF KHLCISGTPK IRLHTGLLLV QLCGGERWWG QFLSNVLQEL YNSEQLLIFP QDRVFM LLS CIGQRSLSNS GVLESLLNLL DNLLSPLQPQ LPMHRRTEGV LDIPMISWVV MLVSRLLDYV ATVEDEAAAA KKPLNGN QW SFINNNLHTQ SLNRSSKGSS SLDRLYSRKI RKQLVHHKQQ LNLLKAKQKA LVEQMEKEKI QSNKGSSYKL LVEQAKLK Q ATSKHFKDLI RLRRTAEWSR SNLDTEVTTA KESPEIEPLP FTLAHERCIS VVQKLVLFLL SMDFTCHADL LLFVCKVLA RIANATRPTI HLCEIVNEPQ LERLLLLLVG TDFNRGDISW GGAWAQYSLT CMLQDILAGE LLAPVAAEAM EEGTVGDDVG ATAGDSDDS LQQSSVQLLE TIDEPLTHDI TGAPPLSSLE KDKEIDLELL QDLMEVDIDP LDIDLEKDPL AAKVFKPISS T WYDYWGAD YGTYNYNPYI GGLGIPVAKP PANTEKNGSQ TVSVSVSQAL DARLEVGLEQ QAELMLKMMS TLEADSILQA LT NTSPTLS QSPTGTDDSL LGGLQAANQT SQLIIQLSSV PMLNVCFNKL FSMLQVHHVQ LESLLQLWLT LSLNSSSTGN KEN GADIFL YNANRIPVIS LNQASITSFL TVLAWYPNTL LRTWCLVLHS LTLMTNMQLN SGSSSAIGTQ ESTAHLLVSD PNLI HVLVK FLSGTSPHGT NQHSPQVGPT ATQAMQEFLT RLQVHLSSTC PQIFSEFLLK LIHILSTERG AFQTGQGPLD AQVKL LEFT LEQNFEVVSV STISAVIESV TFLVHHYITC SDKVMSRSGS DSSVGARACF GGLFANLIRP GDAKAVCGEM TRDQLM FDL LKLVNILVQL PLSGNREYSA RVSVTTNTTD SVSDEEKVSG GKDGNGSSTS VQGSPAYVAD LVLANQQIMS QILSALG LC NSSAMAMIIG ASGLHLTKHE NFHGGLDAIS VGDGLFTILT TLSKKASTVH MMLQPILTYM ACGYMGRQGS LATCQLSE P LLWFILRVLD TSDALKAFHD MGGVQLICNN MVTSTRAIVN TARSMVSTIM KFLDSGPNKA VDSTLKTRIL ASEPDNAEG IHNFAPLGTI TSSSPTAQPA EVLLQATPPH RRARSAAWSY IFLPEEAWCD LTIHLPAAVL LKEIHIQPHL ASLATCPSSV SVEVSADGV NMLPLSTPVV TSGLTYIKIQ LVKAEVASAV CLRLHRPRDA STLGLSQIKL LGLTAFGTTS SATVNNPFLP S EDQVSKTS IGWLRLLHHC LTHISDLEGM MASAAAPTAN LLQTCAALLM SPYCGMHSPN IEVVLVKIGL QSTRIGLKLI DI LLRNCAA SGSDPTDLNS PLLFGRLNGL SSDSTIDILY QLGTTQDPGT KDRIQALLKW VSDSARVAAM KRSGRMNYMC PNS STVEYG LLMPSPSHLH CVAAILWHSY ELLVEYDLPA LLDQELFELL FNWSMSLPCN MVLKKAVDSL LCSMCHVHPN YFSL LMGWM GITPPPVQCH HRLSMTDDSK KQDLSSSLTD DSKNAQAPLA LTESHLATLA SSSQSPEAIK QLLDSGLPSL LVRSL ASFC FSHISSSESI AQSIDISQDK LRRHHVPQQC NKMPITADLV APILRFLTEV GNSHIMKDWL GGSEVNPLWT ALLFLL CHS GSTSGSHNLG AQQTSARSAS LSSAATTGLT TQQRTAIENA TVAFFLQCIS CHPNNQKLMA QVLCELFQTS PQRGNLP TS GNISGFIRRL FLQLMLEDEK VTMFLQSPCP LYKGRINATS HVIQHPMYGA GHKFRTLHLP VSTTLSDVLD RVSDTPSI T AKLISEQKDD KEKKNHEEKE KVKAENGFQD NYSVVVASGL KSQSKRAVSA TPPRPPSRRG RTIPDKIGST SGAEAANKI ITVPVFHLFH KLLAGQPLPA EMTLAQLLTL LYDRKLPQGY RSIDLTVKLG SRVITDPSLS KTDSYKRLHP EKDHGDLLAS CPEDEALTP GDECMDGILD ESLLETCPIQ SPLQVFAGMG GLALIAERLP MLYPEVIQQV SAPVVTSTTQ EKPKDSDQFE W VTIEQSGE LVYEAPETVA AEPPPIKSAV QTMSPIPAHS LAAFGLFLRL PGYAEVLLKE RKHAQCLLRL VLGVTDDGEG SH ILQSPSA NVLPTLPFHV LRSLFSTTPL TTDDGVLLRR MALEIGALHL ILVCLSALSH HSPRVPNSSV NQTEPQVSSS HNP TSTEEQ QLYWAKGTGF GTGSTASGWD VEQALTKQRL EEEHVTCLLQ VLASYINPVS SAVNGEAQSS HETRGQNSNA LPSV LLELL SQSCLIPAMS SYLRNDSVLD MARHVPLYRA LLELLRAIAS CAAMVPLLLP LSTENGEEEE EQSECQTSVG TLLAK MKTC VDTYTNRLRS KRENVKTGVK PDASDQEPEG LTLLVPDIQK TAEIVYAATT SLRQANQEKK LGEYSKKAAM KPKPLS VLK SLEEKYVAVM KKLQFDTFEM VSEDEDGKLG FKVNYHYMSQ VKNANDANSA ARARRLAQEA VTLSTSLPLS SSSSVFV RC DEERLDIMKV LITGPADTPY ANGCFEFDVY FPQDYPSSPP LVNLETTGGH SVRFNPNLYN DGKVCLSILN TWHGRPEE K WNPQTSSFLQ VLVSVQSLIL VAEPYFNEPG YERSRGTPSG TQSSREYDGN IRQATVKWAM LEQIRNPSPC FKEVIHKHF YLKRVEIMAQ CEEWIADIQQ YSSDKRVGRT MSHHAAALKR HTAQLREELL KLPCPEGLDP DTDDAPEVCR ATTGAEETLM HDQVKPSSS KELPSDFQL UniProtKB: Baculoviral IAP repeat-containing protein 6 |
-分子 #2: Serine protease HTRA2, mitochondrial
| 分子 | 名称: Serine protease HTRA2, mitochondrial / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO / EC番号: HtrA2 peptidase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 35.961887 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAVPSPPPAS PRSQYNFIAD VVEKTAPAVV YIEILDRHPF LGREVPISNG SGFVVAADGL IVTNAHVVAD RRRVRVRLLS GDTYEAVVT AVDPVADIAT LRIQTKEPLP TLPLGRSADV RQGEFVVAMG SPFALQNTIT SGIVSSAQRP ARDLGLPQTN V EYIQTDAA ...文字列: MAVPSPPPAS PRSQYNFIAD VVEKTAPAVV YIEILDRHPF LGREVPISNG SGFVVAADGL IVTNAHVVAD RRRVRVRLLS GDTYEAVVT AVDPVADIAT LRIQTKEPLP TLPLGRSADV RQGEFVVAMG SPFALQNTIT SGIVSSAQRP ARDLGLPQTN V EYIQTDAA IDFGNAGGPL VNLDGEVIGV NTMKVTAGIS FAIPSDRLRE FLHRGEKKNS SSGISGSQRR YIGVMMLTLS PS ILAELQL REPSFPDVQH GVLIHKVILG SPAHRAGLRP GDVILAIGEQ MVQNAEDVYE AVRTQSQLAV QIRRGRETLT LYV TPEVTE HHHHHH UniProtKB: Serine protease HTRA2, mitochondrial |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 283.15 K / 装置: LEICA EM GP | ||||||||||||
| 詳細 | added CHAPSO to 0.4 mM |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 詳細 | Data collection in counting mode, using multi-shot scheme (9 holes per stage position, 3 movies per hole) |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 15309 / 平均露光時間: 2.5 sec. / 平均電子線量: 60.659 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-8e2k: |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X