+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 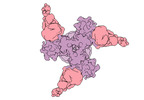 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Vaccine elicited Antibody MU89+S27Y bound to CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 Env trimer | |||||||||
 マップデータ マップデータ | Sharpened map from homogeneous C3 refinement in cryosparc | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 envelope / vaccine-induced antibody / broadly neutralizing antibody / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.32 Å | |||||||||
 データ登録者 データ登録者 | Stalls V / Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2024 ジャーナル: Cell Host Microbe / 年: 2024タイトル: Mutation-guided vaccine design: A process for developing boosting immunogens for HIV broadly neutralizing antibody induction. 著者: Kevin Wiehe / Kevin O Saunders / Victoria Stalls / Derek W Cain / Sravani Venkatayogi / Joshua S Martin Beem / Madison Berry / Tyler Evangelous / Rory Henderson / Bhavna Hora / Shi-Mao Xia / ...著者: Kevin Wiehe / Kevin O Saunders / Victoria Stalls / Derek W Cain / Sravani Venkatayogi / Joshua S Martin Beem / Madison Berry / Tyler Evangelous / Rory Henderson / Bhavna Hora / Shi-Mao Xia / Chuancang Jiang / Amanda Newman / Cindy Bowman / Xiaozhi Lu / Mary E Bryan / Joena Bal / Aja Sanzone / Haiyan Chen / Amanda Eaton / Mark A Tomai / Christopher B Fox / Ying K Tam / Christopher Barbosa / Mattia Bonsignori / Hiromi Muramatsu / S Munir Alam / David C Montefiori / Wilton B Williams / Norbert Pardi / Ming Tian / Drew Weissman / Frederick W Alt / Priyamvada Acharya / Barton F Haynes /   要旨: A major goal of HIV-1 vaccine development is the induction of broadly neutralizing antibodies (bnAbs). Although success has been achieved in initiating bnAb B cell lineages, design of boosting ...A major goal of HIV-1 vaccine development is the induction of broadly neutralizing antibodies (bnAbs). Although success has been achieved in initiating bnAb B cell lineages, design of boosting immunogens that select for bnAb B cell receptors with improbable mutations required for bnAb affinity maturation remains difficult. Here, we demonstrate a process for designing boosting immunogens for a V3-glycan bnAb B cell lineage. The immunogens induced affinity-matured antibodies by selecting for functional improbable mutations in bnAb precursor knockin mice. Moreover, we show similar success in prime and boosting with nucleoside-modified mRNA-encoded HIV-1 envelope trimer immunogens, with improved selection by mRNA immunogens of improbable mutations required for bnAb binding to key envelope glycans. These results demonstrate the ability of both protein and mRNA prime-boost immunogens for selection of rare B cell lineage intermediates with neutralizing breadth after bnAb precursor expansion, a key proof of concept and milestone toward development of an HIV-1 vaccine. #1:  ジャーナル: bioRxiv / 年: 2022 ジャーナル: bioRxiv / 年: 2022タイトル: Mutation-Guided Vaccine Design: A Strategy for Developing Boosting Immunogens for HIV Broadly Neutralizing Antibody Induction 著者: Wiehe K / Saunders KO / Stalls V / Cain D / Venkatayogi S / Martin Beem JS / Berry M / Evangelous T / Henderson R / Hora B / Xia S / Jiang C / Newman A / Bowman C / Lu X / Bryan ME / Bal J / ...著者: Wiehe K / Saunders KO / Stalls V / Cain D / Venkatayogi S / Martin Beem JS / Berry M / Evangelous T / Henderson R / Hora B / Xia S / Jiang C / Newman A / Bowman C / Lu X / Bryan ME / Bal J / Sanzone A / Chen H / Eaton A / Tomai MA / Fox CB / Tam Y / Barbosa C / Bonsignori M / Muramatsu H / Alam SM / Montefiori D / Williams WB / Pardi N / Tian M / Weisman D / Alt FW / Acharya P / Haynes BF | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
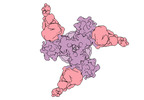
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27776.map.gz emd_27776.map.gz | 117.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27776-v30.xml emd-27776-v30.xml emd-27776.xml emd-27776.xml | 24 KB 24 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27776.png emd_27776.png | 49.9 KB | ||
| Filedesc metadata |  emd-27776.cif.gz emd-27776.cif.gz | 7.4 KB | ||
| その他 |  emd_27776_half_map_1.map.gz emd_27776_half_map_1.map.gz emd_27776_half_map_2.map.gz emd_27776_half_map_2.map.gz | 115.4 MB 115.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27776 http://ftp.pdbj.org/pub/emdb/structures/EMD-27776 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27776 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27776 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27776_validation.pdf.gz emd_27776_validation.pdf.gz | 744 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27776_full_validation.pdf.gz emd_27776_full_validation.pdf.gz | 743.5 KB | 表示 | |
| XML形式データ |  emd_27776_validation.xml.gz emd_27776_validation.xml.gz | 14.2 KB | 表示 | |
| CIF形式データ |  emd_27776_validation.cif.gz emd_27776_validation.cif.gz | 16.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27776 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27776 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27776 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27776 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27776.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27776.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map from homogeneous C3 refinement in cryosparc | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half A map from homogeneous C3 refinement in cryosparc
| ファイル | emd_27776_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half A map from homogeneous C3 refinement in cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half B map from homogeneous C3 refinement in cryosparc
| ファイル | emd_27776_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half B map from homogeneous C3 refinement in cryosparc | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : V3-glycan vaccine induced antibody MU89+S27Y in complex with CH84...
| 全体 | 名称: V3-glycan vaccine induced antibody MU89+S27Y in complex with CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV Env trimer |
|---|---|
| 要素 |
|
-超分子 #1: V3-glycan vaccine induced antibody MU89+S27Y in complex with CH84...
| 超分子 | 名称: V3-glycan vaccine induced antibody MU89+S27Y in complex with CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV Env trimer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 360 KDa |
-分子 #1: Envelope glycoprotein gp160
| 分子 | 名称: Envelope glycoprotein gp160 / タイプ: protein_or_peptide / ID: 1 / 詳細: CH848.D949.10.17_N133D_N138T.DS.SOSIP.664 HIV-1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 72.208086 KDa |
| 組換発現 | 生物種: Mammalia (両生類) |
| 配列 | 文字列: MGSLQPLATL YLLGMLVASV LAAENLWVTV YYGVPVWKEA KTTLFCASDA RAYEKEVHNV WATHACVPTD PSPQELVLGN VTENFNMWK NDMVDQMHED IISLWDQSLK PCVKLTPLCV TLICSDATVK TGTVEEMKNC SFNTTTEIRD KEKKEYALFY K PDIVPLSE ...文字列: MGSLQPLATL YLLGMLVASV LAAENLWVTV YYGVPVWKEA KTTLFCASDA RAYEKEVHNV WATHACVPTD PSPQELVLGN VTENFNMWK NDMVDQMHED IISLWDQSLK PCVKLTPLCV TLICSDATVK TGTVEEMKNC SFNTTTEIRD KEKKEYALFY K PDIVPLSE TNNTSEYRLI NCNTSACTQA CPKVTFEPIP IHYCAPAGYA ILKCNDETFN GTGPCSNVST VQCTHGIRPV VS TQLLLNG SLAEKEIVIR SENLTNNAKI IIVHLHTPVE IVCTRPNNNT RKSVRIGPGQ TFYATGDIIG DIKQAHCNIS EEK WNDTLQ KVGIELQKHF PNKTIKYNQS AGGDMEITTH SFNCGGEFFY CNTSNLFNGT YNGTYISTNS SANSTSTITL QCRI KQIIN MWQGVGRCMY APPIAGNITC RSNITGLLLT RDGGTNSNET ETFRPAGGDM RDNWRSELYK YKVVKIEPLG VAPTR CKRR VVGRRRRRRA VGIGAVFLGF LGAAGSTMGA ASMTLTVQAR NLLSGIVQQQ SNLLRAPEAQ QHLLKLTVWG IKQLQA RVL AVERYLRDQQ LLGIWGCSGK LICCTNVPWN SSWSNRNLSE IWDNMTWLQW DKEISNYTQI IYGLLEESQN QQEKNEQ DL LALD |
-分子 #2: MU89+S27Y Heavy Chain
| 分子 | 名称: MU89+S27Y Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.162116 KDa |
| 組換発現 | 生物種: Mammalia (両生類) |
| 配列 | 文字列: MGWSCIILFL VATATGVHSQ VQLVQSGAEV KKPGASVKVS CKASGYRFTD HYIHWVRQAP GQGPEWMGWI NTSSGRSNFA QKFQGRVTM TRDTSISTAY MELNRLKSDD TAVYYCTTGS WISLYYDSSG YPNFDYWGQG TLVTVT |
-分子 #3: MU89+S27Y Light Chain
| 分子 | 名称: MU89+S27Y Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.464601 KDa |
| 組換発現 | 生物種: Mammalia (両生類) |
| 配列 | 文字列: QSALTQPASV SGSPGQPITI SCTGTSYDVG NYDLVSWYQQ HPGNAPKYMI YEVTKRPAGI SNRFSGSKSG NTASLTISGL QAEDAADYY CCSYAGSSTV IFGGGTKVTV L |
-分子 #10: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 10 / コピー数: 9 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 1801 / 平均電子線量: 59.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X