+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of SARS-CoV-2 HexaPro Spike ectodomain in complex with DH1338 Fab, 3-RBD-up conformation | |||||||||
 マップデータ マップデータ | Sharp map from homogeneous C1 refinement in cryosparc | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SARS-CoV-2 / VIRAL PROTEIN / Antibody / Fab / RBD / 3-RBD-up | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Stalls V / Acharya P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM Structure of SARS-CoV-2 HexaPro Spike ectodomain in complex with DH1338 Fab, 3-RBD-up conformation 著者: Stalls V / Acharya P / Malewana D | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27606.map.gz emd_27606.map.gz | 96.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27606-v30.xml emd-27606-v30.xml emd-27606.xml emd-27606.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27606.png emd_27606.png | 48.6 KB | ||
| その他 |  emd_27606_half_map_1.map.gz emd_27606_half_map_1.map.gz emd_27606_half_map_2.map.gz emd_27606_half_map_2.map.gz | 94.8 MB 94.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27606 http://ftp.pdbj.org/pub/emdb/structures/EMD-27606 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27606 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27606 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27606_validation.pdf.gz emd_27606_validation.pdf.gz | 726.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27606_full_validation.pdf.gz emd_27606_full_validation.pdf.gz | 726.4 KB | 表示 | |
| XML形式データ |  emd_27606_validation.xml.gz emd_27606_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_27606_validation.cif.gz emd_27606_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27606 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27606 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27606 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27606 | HTTPS FTP |
-関連構造データ
| 関連構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27606.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27606.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharp map from homogeneous C1 refinement in cryosparc | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half B map from homogeneous C1 refinement in cryosparc
| ファイル | emd_27606_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half B map from homogeneous C1 refinement in cryosparc | ||||||||||||
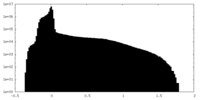
| 投影像・断面図 |
| ||||||||||||
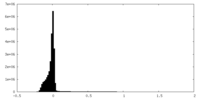
| 密度ヒストグラム |
-ハーフマップ: Half A map from homogeneous C1 refinement in cryosparc
| ファイル | emd_27606_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half A map from homogeneous C1 refinement in cryosparc | ||||||||||||
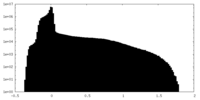
| 投影像・断面図 |
| ||||||||||||
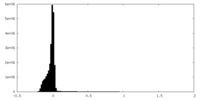
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ab026500 Fab bound to SARS-CoV-2 HexaPro Spike Trimer
| 全体 | 名称: Ab026500 Fab bound to SARS-CoV-2 HexaPro Spike Trimer |
|---|---|
| 要素 |
|
-超分子 #1: Ab026500 Fab bound to SARS-CoV-2 HexaPro Spike Trimer
| 超分子 | 名称: Ab026500 Fab bound to SARS-CoV-2 HexaPro Spike Trimer タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 556 KDa |
-分子 #1: Spike glycoprotein HexaPro
| 分子 | 名称: Spike glycoprotein HexaPro / タイプ: other / ID: 1 / 分類: other |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: VNLTTRTQLP PAYTNSFTRG VYYPDKVFRS SVLHSTQDLF LPFFSNVTWF HAIHVSGTNG TKRFDNPVLP FNDGVYFAST EKSNIIRGWI FGTTLDSKTQ SLLIVNNATN VVIKVCEFQF CNDPFLGVYY HKNNKSWMES EFRVYSSANN CTFEYVSQPF LMDLEGKQGN ...文字列: VNLTTRTQLP PAYTNSFTRG VYYPDKVFRS SVLHSTQDLF LPFFSNVTWF HAIHVSGTNG TKRFDNPVLP FNDGVYFAST EKSNIIRGWI FGTTLDSKTQ SLLIVNNATN VVIKVCEFQF CNDPFLGVYY HKNNKSWMES EFRVYSSANN CTFEYVSQPF LMDLEGKQGN FKNLREFVFK NIDGYFKIYS KHTPINLVRD LPQGFSALEP LVDLPIGINI TRFQTLLALH RSYLTPGDSS SGWTAGAAAY YVGYLQPRTF LLKYNENGTI TDAVDCALDP LSETKCTLKS FTVEKGIYQT SNFRVQPTES IVRFPNITNL CPFGEVFNAT RFASVYAWNR KRISNCVADY SVLYNSASFS TFKCYGVSPT KLNDLCFTNV YADSFVIRGD EVRQIAPGQT GKIADYNYKL PDDFTGCVIA WNSNNLDSKV GGNYNYLYRL FRKSNLKPFE RDISTEIYQA GSTPCNGVEG FNCYFPLQSY GFQPTNGVGY QPYRVVVLSF ELLHAPATVC GPKKSTNLVK NKCVNFNFNG LTGTGVLTES NKKFLPFQQF GRDIADTTDA VRDPQTLEIL DITPCSFGGV SVITPGTNTS NQVAVLYQDV NCTEVPVAIH ADQLTPTWRV YSTGSNVFQT RAGCLIGAEH VNNSYECDIP IGAGICASYQ TQTNSPGSAS SVASQSIIAY TMSLGAENSV AYSNNSIAIP TNFTISVTTE ILPVSMTKTS VDCTMYICGD STECSNLLLQ YGSFCTQLNR ALTGIAVEQD KNTQEVFAQV KQIYKTPPIK DFGGFNFSQI LPDPSKPSKR SPIEDLLFNK VTLADAGFIK QYGDCLGDIA ARDLICAQKF NGLTVLPPLL TDEMIAQYTS ALLAGTITSG WTFGAGPALQ IPFPMQMAYR FNGIGVTQNV LYENQKLIAN QFNSAIGKIQ DSLSSTPSAL GKLQDVVNQN AQALNTLVKQ LSSNFGAISS VLNDILSRLD PPEAEVQIDR LITGRLQSLQ TYVTQQLIRA AEIRASANLA ATKMSECVLG QSKRVDFCGK GYHLMSFPQS APHGVVFLHV TYVPAQEKNF TTAPAICHDG KAHFPREGVF VSNGTHWFVT QRNFYEPQII TTDNTFVSGN CDVVIGIVNN TVYDPLQPEL DSFKEELDKY FKNHTSPDVD LGDISGINAS VVNIQKEIDR LNEVAKNLNE SLIDLQELGK YEQGSGYIPE APRDGQAYVR KDGEWVLLST FLGRSLEVLF QGPGHHHHHH HHSAWSHPQF EKGGGSGGGG SGGSAWSHPQ FEK |
| 組換発現 | 生物種: Mammalia (両生類) |
-分子 #2: DH1338 Fab Heavy Chain
| 分子 | 名称: DH1338 Fab Heavy Chain / タイプ: other / ID: 2 / 分類: other |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MGWSCIILFL VATATGVHAQ LQLQESGPGL VKPSETLSLT CAVSGGSISR KNWSWIRQPP GKGLGWIGRI SGSGGSTDYN PSLKSRVTIS TDTSKNQFSL KLSSVTAADT AVYYCARVAY YEDDYGYYYT EGAFDFWGQG LRVTVSSAST KGPSVFPLAP SSRSTSESTA ...文字列: MGWSCIILFL VATATGVHAQ LQLQESGPGL VKPSETLSLT CAVSGGSISR KNWSWIRQPP GKGLGWIGRI SGSGGSTDYN PSLKSRVTIS TDTSKNQFSL KLSSVTAADT AVYYCARVAY YEDDYGYYYT EGAFDFWGQG LRVTVSSAST KGPSVFPLAP SSRSTSESTA ALGCLVKDYF PEPVTVSWNS GSLTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYVC NVNHKPSNTK VDKRVEI |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #3: DH1338 Fab Light Chain
| 分子 | 名称: DH1338 Fab Light Chain / タイプ: other / ID: 3 / 分類: other |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | 文字列: MGWSCIILFL VATATGVHAE IVMTQSPATL SLSPGERATL SCRTSQSVSS YVAWYQQKPE QTPRLLIYDA SSRATGIPDR FSGGGSGTDF TLTISSLEPE DFAVYYCQQY SNWPWTFGQG TKVEIKRAVA APSVFIFPPS EDQVKSGTVS VVCLLNNFYP REASVKWKVD ...文字列: MGWSCIILFL VATATGVHAE IVMTQSPATL SLSPGERATL SCRTSQSVSS YVAWYQQKPE QTPRLLIYDA SSRATGIPDR FSGGGSGTDF TLTISSLEPE DFAVYYCQQY SNWPWTFGQG TKVEIKRAVA APSVFIFPPS EDQVKSGTVS VVCLLNNFYP REASVKWKVD GVLKTGNSQE SVTEQDSKDN TYSLSSTLTL SSTDYQSHNV YACEVTHQGL SSPVTKSFNR GEC |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: LEICA EM GP |
| 詳細 | Fab to spike 6:1 ratio. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 7865 / 平均電子線量: 59.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X