+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 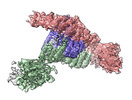 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of a HOPS core complex containing Vps33, Vps16, and Vps18 | ||||||||||||
 マップデータ マップデータ | Cryo-EM structure of a HOPS core complex containing Vps33, Vps16, and Vps18 | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | HOPS / membrane tethering / SNAREs / membrane fusion / endolysosomal trafficking / PROTEIN TRANSPORT | ||||||||||||
| 生物種 |  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.1 Å | ||||||||||||
 データ登録者 データ登録者 | Port SA / Farrell PD / Jeffrey PD / DiMaio F / Hughson FM | ||||||||||||
| 資金援助 |  ドイツ, ドイツ,  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Cryo-EM structure of the HOPS core complex and its implication for SNARE assembly 著者: Port SA / Farrell PD / Jeffrey PD / DiMaio F / Hughson FM | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27442.map.gz emd_27442.map.gz | 164.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27442-v30.xml emd-27442-v30.xml emd-27442.xml emd-27442.xml | 23.5 KB 23.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27442_fsc.xml emd_27442_fsc.xml | 16.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27442.png emd_27442.png | 114.6 KB | ||
| Filedesc metadata |  emd-27442.cif.gz emd-27442.cif.gz | 7.7 KB | ||
| その他 |  emd_27442_half_map_1.map.gz emd_27442_half_map_1.map.gz emd_27442_half_map_2.map.gz emd_27442_half_map_2.map.gz | 165 MB 165 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27442 http://ftp.pdbj.org/pub/emdb/structures/EMD-27442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27442 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27442 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27442_validation.pdf.gz emd_27442_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27442_full_validation.pdf.gz emd_27442_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_27442_validation.xml.gz emd_27442_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  emd_27442_validation.cif.gz emd_27442_validation.cif.gz | 26.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27442 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27442 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27442 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27442 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27442.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27442.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM structure of a HOPS core complex containing Vps33, Vps16, and Vps18 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.426 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map B
| ファイル | emd_27442_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_27442_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : HOPS core complex
| 全体 | 名称: HOPS core complex |
|---|---|
| 要素 |
|
-超分子 #1: HOPS core complex
| 超分子 | 名称: HOPS core complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
| 分子量 | 理論値: 171 KDa |
-超分子 #2: His-Vps33:Vps16
| 超分子 | 名称: His-Vps33:Vps16 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
-超分子 #3: Vps18
| 超分子 | 名称: Vps18 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
-分子 #1: Vacuolar protein sorting-associated protein 33
| 分子 | 名称: Vacuolar protein sorting-associated protein 33 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
| 分子量 | 理論値: 77.119617 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKHHHHHHHG AAGTSLYKKA GENLYFQGSM APRAGFDAEQ VRDKARKDLL HLLEGVRGKK NLVIEKDLAG PLGVIVKAST LRDYGVDNF FFLENKNTGT SQRNIVFIAR GESVRNAHAI AAQIKRIQRE SQTSHDFHIF WVPRRTLFSD KVLEEAGVLG D ANISELPL ...文字列: MKHHHHHHHG AAGTSLYKKA GENLYFQGSM APRAGFDAEQ VRDKARKDLL HLLEGVRGKK NLVIEKDLAG PLGVIVKAST LRDYGVDNF FFLENKNTGT SQRNIVFIAR GESVRNAHAI AAQIKRIQRE SQTSHDFHIF WVPRRTLFSD KVLEEAGVLG D ANISELPL YFFPLERDVL SLELNDSFRD LYLAKDPTPV FLLSRALMGI QKKHGLFPRI IGKGENAKRV ADLLSRMRQE LL AGEEAGE SDRAGLSPST TIESVIIIDR EVDFVTPLLT QLTYEGLIDE YFGIQNNQTD VDAVIVGAPA QSAASTSTAV PTN SSQSRK RKIQLDGSDS LYSQLRDANF AIVGSLLNTV ARRLKSDYES RHNTKTTAEL KEFVKKLPGY QAEQQSLKIH SNIA EEIIN YTRTEIFNKL LEVQQNLAAG ADPSSQFDSI EELVARDTPL PQVLRLLCLY SCISGGIKTK ELDHFRRLVL QGYGH QHLL TLHNLERLQM FLSKSSPLAS MITMSGSSGG PDQKTNYTYL RKQLRLIVDE VNEQDPNDIA YVYSGYAPLS IRLVQC VLQ KQYLLSITKG SGTVIAAGPV AGGGAQGWKG FEEIVKHARG PTFDEIQKGE DKAVKARALL SGSSGDKKTV FVVFVGG IT FTEIAALRFI AKQEEARRNI VICTTSIING NRMMNAAIET ATFEKTTVTT AAAQ |
-分子 #2: Vacuolar protein sorting-associated protein 16
| 分子 | 名称: Vacuolar protein sorting-associated protein 16 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
| 分子量 | 理論値: 94.204828 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDTAHPTASW EQLGERFYRK IQLYTQVFDQ DFDLDNYIVT GAPYGGAIAL YRDDEKLVAY QPSRSSRPTI DICSLSGKLL RRISWDQGP IKGVGWSEDE KLLIVMVDGT VRCYFDLQSE FTQFSLGHGA EEHGVKSCRF YSHGLVALLG NNALVSVSSY D EPRPKLLA ...文字列: MDTAHPTASW EQLGERFYRK IQLYTQVFDQ DFDLDNYIVT GAPYGGAIAL YRDDEKLVAY QPSRSSRPTI DICSLSGKLL RRISWDQGP IKGVGWSEDE KLLIVMVDGT VRCYFDLQSE FTQFSLGHGA EEHGVKSCRF YSHGLVALLG NNALVSVSSY D EPRPKLLA SPPEGRVYSW NIIPPAYSLS RSVEVLLSVN QTIYVCDASE CEDRFLDIGP FSHIAVSPNG RFCALYTTTG KV HVITSDF QSRLSEHDTK SKIAPNYFEW CGNDAVVIAW DDEVHLVGPS GSLARFFYDS GRIHLIPDFD GVRILANDRC DFL QKVPDV IEEVFGLGAD SPASILLDAV EQLEMKSPKA DDNIQLIRPH LVEAVDTCVS AAGQEFSIHW QKQLLKAASF GKSV LDIYN SDDFVDMCET LRVLNAVRFY EVGLPLSYEQ YQRLSPSGLI SRLLNRHEYL LAIRIADHLR LPTDKIHVHW ASAKV RLGS EDDDTICRKI VEKLSGKPGI SFEVIARTAY EEGRTRLATE LLNHEPRAGR QVPLLLSMEE DELALDKAIE SGDTDL IYF VIHQLRRKLP LASFFRVVSS RPTASAMVEA LARNSDGDGN EDTALLKDLY YQDDRRLDGA SVFIREALQQ PETRTAS DK LDLAANLLQG NQKEHVFELG ALKEAKMLLR MQETFERDLT DSFVGLSVNQ TMFKLIKLGY HGRAKKIQSE FKVPERVA W WIRLQALVAK RDWNEIEEIS RQRKSPIGWE PFFNQVLQAG NPRLAATFIP KCTNLEPGQT ITMYEKCGMR VKAAEEAVR LKDTEAWNRL LEAAGRNTAE GREIERLGAT VFKK |
-分子 #3: Vacuolar protein sorting-associated protein 18
| 分子 | 名称: Vacuolar protein sorting-associated protein 18 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) |
| 分子量 | 理論値: 108.52757 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MALDLSSGFA AADAIANLQL ADATLPIFEV LPVQLQFSVA ADFVAGQAAN NVLVIALSNG RILRIDLNKP EDIDDIDLPK KPSEVGVIR RMFLDPTASH LIICTSLGEN YYLHSQSRQP RPLARLRGVV IESIAWSPAL PTQSTREILI GAADGNIYEA Y IETSTEFY ...文字列: MALDLSSGFA AADAIANLQL ADATLPIFEV LPVQLQFSVA ADFVAGQAAN NVLVIALSNG RILRIDLNKP EDIDDIDLPK KPSEVGVIR RMFLDPTASH LIICTSLGEN YYLHSQSRQP RPLARLRGVV IESIAWSPAL PTQSTREILI GAADGNIYEA Y IETSTEFY RREDKYLKLV QKLPDGPITG LWADSLPGHK DTRRVLVATS SRLFHWVGKI GRGHDSGGGA SIYDKLFEAE QP TVHALSG ASAAAMSMLV VSPDAEQPSP RFREDEVPER AFAWLSSHGV YHGKLLLKGP LSELGAKVFA EAKLLPRAQL ANP EGASRR QLSTEYVDAV ALTQWHIVSL VAGRVVIANR LTGSIIYDQT ILNPGQKAVG LCVDQQKSTF WLFTPQEIFE IVPR DEDRD IWKIMLQLQQ FDAALQYAHT PAEKDAVAIA SGDHLVSKGQ FLEAAAVYGK SSKPFEEVAL TFIDNEQPDA LRKYL LTKL GTYKKSAVMQ RVMIATWLIE VFMAKLNSLD DTIITGAELS ETLNPNQTKE QLEAVRAEFQ DFINKHKGDL DRKTVY DVI GSHGREEELL YYANAINDYN YVLSYWVQRE RWTEALKVLK KQTDPEVFYR YSSVLMTHAA TELVEILMRQ SNLNPRN LI PAMLEYDRNY KGPLAQNQAV RYLLYVVNQL GSTDSAVHNT LVSIYASHPS KDESALLEYL ESQGEEPNYD PDFALRLC I QHRRVLSCAH IYTSMGQYGA AVDLALAHDE VELASIIADR PISNPQLRKK LWLKVAKKVI SQQSDGIKTA IDFLRRCDL LKIEDLIPFF PDFVVIDDFK EEICAALEDY SRNIDALRRE MDEASQTAAN IKVDIAALDK RYAIVEPGEK CYACGLPLLS RQFFVFPCQ HAFHSDCLAR RVLEQAPPAK ARRIKECQVQ ISKGLVNGEK REAMIAELDA LIASACDYAI RRINEPFIKD D DDKDEWAL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.08 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 詳細: PELCO Easiglow, 10 mA/ 10 s discharge/ 30 s hold | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV 詳細: Blotting parameters: 30 seconds waiting time, blotting force 1, blot total 1. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-32 / 撮影したグリッド数: 1 / 実像数: 3590 / 平均露光時間: 5.6 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.03 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 2.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X