+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of CasLambda (Cas12l) bound to crRNA and DNA | |||||||||
 マップデータ マップデータ | LocSpiral map from cryoSPARC half maps | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR / RNA Binding Protein / DNA binding protein / phage / viral protein / enzyme / ribonucleoprotein / RNA BINDING PROTEIN-RNA-DNA complex | |||||||||
| 生物種 |  uncultured virus (環境試料) uncultured virus (環境試料) | |||||||||
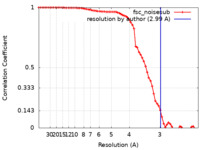
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.99 Å | |||||||||
 データ登録者 データ登録者 | Al-Shayeb B / Skopintsev P / Soczek K / Doudna J | |||||||||
| 資金援助 |  米国, 米国,  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: Diverse virus-encoded CRISPR-Cas systems include streamlined genome editors. 著者: Basem Al-Shayeb / Petr Skopintsev / Katarzyna M Soczek / Elizabeth C Stahl / Zheng Li / Evan Groover / Dylan Smock / Amy R Eggers / Patrick Pausch / Brady F Cress / Carolyn J Huang / Brian ...著者: Basem Al-Shayeb / Petr Skopintsev / Katarzyna M Soczek / Elizabeth C Stahl / Zheng Li / Evan Groover / Dylan Smock / Amy R Eggers / Patrick Pausch / Brady F Cress / Carolyn J Huang / Brian Staskawicz / David F Savage / Steven E Jacobsen / Jillian F Banfield / Jennifer A Doudna /   要旨: CRISPR-Cas systems are host-encoded pathways that protect microbes from viral infection using an adaptive RNA-guided mechanism. Using genome-resolved metagenomics, we find that CRISPR systems are ...CRISPR-Cas systems are host-encoded pathways that protect microbes from viral infection using an adaptive RNA-guided mechanism. Using genome-resolved metagenomics, we find that CRISPR systems are also encoded in diverse bacteriophages, where they occur as divergent and hypercompact anti-viral systems. Bacteriophage-encoded CRISPR systems belong to all six known CRISPR-Cas types, though some lack crucial components, suggesting alternate functional roles or host complementation. We describe multiple new Cas9-like proteins and 44 families related to type V CRISPR-Cas systems, including the Casλ RNA-guided nuclease family. Among the most divergent of the new enzymes identified, Casλ recognizes double-stranded DNA using a uniquely structured CRISPR RNA (crRNA). The Casλ-RNA-DNA structure determined by cryoelectron microscopy reveals a compact bilobed architecture capable of inducing genome editing in mammalian, Arabidopsis, and hexaploid wheat cells. These findings reveal a new source of CRISPR-Cas enzymes in phages and highlight their value as genome editors in plant and human cells. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27320.map.gz emd_27320.map.gz | 1.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27320-v30.xml emd-27320-v30.xml emd-27320.xml emd-27320.xml | 23.3 KB 23.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27320_fsc.xml emd_27320_fsc.xml | 7.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27320.png emd_27320.png | 84.4 KB | ||
| マスクデータ |  emd_27320_msk_1.map emd_27320_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-27320.cif.gz emd-27320.cif.gz | 6.6 KB | ||
| その他 |  emd_27320_additional_1.map.gz emd_27320_additional_1.map.gz emd_27320_additional_2.map.gz emd_27320_additional_2.map.gz emd_27320_half_map_1.map.gz emd_27320_half_map_1.map.gz emd_27320_half_map_2.map.gz emd_27320_half_map_2.map.gz | 28.8 MB 15.4 MB 28.4 MB 28.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27320 http://ftp.pdbj.org/pub/emdb/structures/EMD-27320 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27320 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27320 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27320_validation.pdf.gz emd_27320_validation.pdf.gz | 919.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27320_full_validation.pdf.gz emd_27320_full_validation.pdf.gz | 918.8 KB | 表示 | |
| XML形式データ |  emd_27320_validation.xml.gz emd_27320_validation.xml.gz | 14.2 KB | 表示 | |
| CIF形式データ |  emd_27320_validation.cif.gz emd_27320_validation.cif.gz | 18 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27320 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27320 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27320 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27320 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27320.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27320.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | LocSpiral map from cryoSPARC half maps | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.115 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27320_msk_1.map emd_27320_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
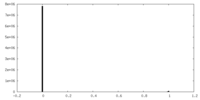
| 投影像・断面図 |
| ||||||||||||
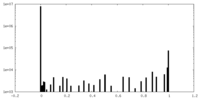
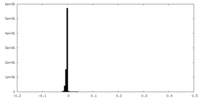
| 密度ヒストグラム |
-追加マップ: cryoSPARC sharp map
| ファイル | emd_27320_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoSPARC sharp map | ||||||||||||
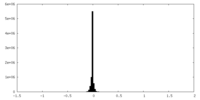
| 投影像・断面図 |
| ||||||||||||
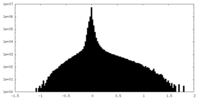
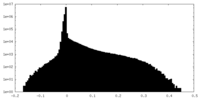
| 密度ヒストグラム |
-追加マップ: cryoSPARC map
| ファイル | emd_27320_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | cryoSPARC map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27320_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
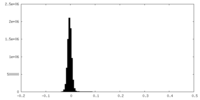
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27320_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
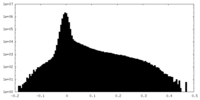
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CasLambda-crRNA-dsDNA ternary structure
| 全体 | 名称: CasLambda-crRNA-dsDNA ternary structure |
|---|---|
| 要素 |
|
-超分子 #1: CasLambda-crRNA-dsDNA ternary structure
| 超分子 | 名称: CasLambda-crRNA-dsDNA ternary structure / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  uncultured virus (環境試料) uncultured virus (環境試料) |
-分子 #1: CasLambda
| 分子 | 名称: CasLambda / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  uncultured virus (環境試料) uncultured virus (環境試料) |
| 分子量 | 理論値: 87.337969 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASHKKTESN QIIKTFSFKI KNANGLSLDV LNDAITEYQN YYNICSDWIK DHLTMKISEL YKYIPNEKKN SGYALTLISD EWKDKPMYM MFKKGYPANN RDNAIYETLN TCNTEHYTGN ILNFSDTYYR RFGYVASAIS NYVTKISKMS TGSRSKNISN D SDVDTIME ...文字列: MASHKKTESN QIIKTFSFKI KNANGLSLDV LNDAITEYQN YYNICSDWIK DHLTMKISEL YKYIPNEKKN SGYALTLISD EWKDKPMYM MFKKGYPANN RDNAIYETLN TCNTEHYTGN ILNFSDTYYR RFGYVASAIS NYVTKISKMS TGSRSKNISN D SDVDTIME QVIYEMEHNG WTSVKDWENQ MEYLESKTDS NPNFVYRMTT LYEFYKSHID EVNSKMETMS IDSLIKFGGC RR KDSKKSM YIMGGSNTPF DITQIGGNSL NIKFSKNLNV DVFGRYDVIK DNTLLVDIIN GHGASFVLKI INDEIYIDIN VSV PFDKKI ATTNKVVGID VNIKHMLLAT NILDDGNVKG YVNIYKEVIN DSDFKKVCNS TVMQYFTDFS KFVTFCPLEF DFLF SRVCN QKGIYNDNSA MEKSFSDVLN KLKWNFIETG DNTKRIYIEN VMKLRSQMKA YAIVKNAYYK QQSEYDFGKS EEFIQ EHPF SNTDKGIEIL NKLDNISKKI LGCRNNIIQY SYNLFEINGY DMVSLEKLTS SQFKKKPFPT VNSLLKYHKI LGCTQE EME KKDIYSVIKK GYYDIIFDND VVTDAKLSAK GELSKFKDDF FNLMIKSIHF ADIKDYFITL SNNGTAGVSL VPSYFTS QM DSIDHKIYFV QDNKSGKLKL ANKHKVRSSQ EKHINGLNAD YNAARNIAYI MENTDCRNMF MKQSRTDKSL YNKPSYET F IKTQGSAVAK LKKEGFVKIL DEASVGSSGH HHHHH |
-分子 #2: RNA (51-MER)
| 分子 | 名称: RNA (51-MER) / タイプ: rna / ID: 2 / コピー数: 1 |
|---|---|
| 由来(天然) | 生物種:  uncultured virus (環境試料) uncultured virus (環境試料) |
| 分子量 | 理論値: 16.483719 KDa |
| 配列 | 文字列: AUUGUUGUAA CUCUUAUUUU GUAUGGAGUA AACAACUAGC AUCACCUUCA CC |
-分子 #3: DNA TS
| 分子 | 名称: DNA TS / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  uncultured virus (環境試料) uncultured virus (環境試料) |
| 分子量 | 理論値: 14.296228 KDa |
| 配列 | 文字列: (DC)(DA)(DT)(DT)(DA)(DA)(DC)(DA)(DT)(DT) (DA)(DC)(DT)(DA)(DA)(DG)(DA)(DG)(DG)(DG) (DT)(DG)(DA)(DA)(DG)(DG)(DT)(DG)(DA) (DT)(DG)(DC)(DT)(DA)(DC)(DA)(DA)(DA)(DC) (DG) (DG)(DT)(DC)(DA)(DA)(DG) |
-分子 #4: DNA NTS
| 分子 | 名称: DNA NTS / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  uncultured virus (環境試料) uncultured virus (環境試料) |
| 分子量 | 理論値: 14.371228 KDa |
| 配列 | 文字列: (DC)(DT)(DT)(DG)(DA)(DC)(DC)(DG)(DT)(DT) (DT)(DG)(DA)(DT)(DC)(DG)(DT)(DA)(DG)(DT) (DG)(DG)(DA)(DA)(DG)(DT)(DG)(DG)(DG) (DA)(DG)(DA)(DT)(DA)(DG)(DT)(DA)(DA)(DT) (DG) (DT)(DT)(DA)(DA)(DT)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 281 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-8dc2: |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X