+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cereblon bound to CC-92480, Focused Refinement | |||||||||
 マップデータ マップデータ | focused refinement of CRBN~DDB1 with Mezigdomide | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Watson ER / Lander GC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2022 ジャーナル: Science / 年: 2022タイトル: Molecular glue CELMoD compounds are regulators of cereblon conformation. 著者: Edmond R Watson / Scott Novick / Mary E Matyskiela / Philip P Chamberlain / Andres H de la Peña / Jinyi Zhu / Eileen Tran / Patrick R Griffin / Ingrid E Wertz / Gabriel C Lander /  要旨: Cereblon (CRBN) is a ubiquitin ligase (E3) substrate receptor protein co-opted by CRBN E3 ligase modulatory drug (CELMoD) agents that target therapeutically relevant proteins for degradation. Prior ...Cereblon (CRBN) is a ubiquitin ligase (E3) substrate receptor protein co-opted by CRBN E3 ligase modulatory drug (CELMoD) agents that target therapeutically relevant proteins for degradation. Prior crystallographic studies defined the drug-binding site within CRBN's thalidomide-binding domain (TBD), but the allostery of drug-induced neosubstrate binding remains unclear. We performed cryo-electron microscopy analyses of the DNA damage-binding protein 1 (DDB1)-CRBN apo complex and compared these structures with DDB1-CRBN in the presence of CELMoD compounds alone and complexed with neosubstrates. Association of CELMoD compounds to the TBD is necessary and sufficient for triggering CRBN allosteric rearrangement from an open conformation to the canonical closed conformation. The neosubstrate Ikaros only stably associates with the closed CRBN conformation, illustrating the importance of allostery for CELMoD compound efficacy and informing structure-guided design strategies to improve therapeutic efficacy. #1:  ジャーナル: Biorxiv / 年: 2022 ジャーナル: Biorxiv / 年: 2022タイトル: Molecular glue CELMoD compounds are allosteric regulators of cereblon conformation 著者: Watson ER / Novick SJ / Matyskiela ME / Chamberlain PP / de la Pena AH / Zhu JY / Tran E / Griffin PR / Wertz IE / Lander GC | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27239.map.gz emd_27239.map.gz | 40.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27239-v30.xml emd-27239-v30.xml emd-27239.xml emd-27239.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27239_fsc.xml emd_27239_fsc.xml | 7.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27239.png emd_27239.png | 112.4 KB | ||
| マスクデータ |  emd_27239_msk_1.map emd_27239_msk_1.map | 42.9 MB |  マスクマップ マスクマップ | |
| その他 |  emd_27239_additional_1.map.gz emd_27239_additional_1.map.gz emd_27239_additional_2.map.gz emd_27239_additional_2.map.gz emd_27239_half_map_1.map.gz emd_27239_half_map_1.map.gz emd_27239_half_map_2.map.gz emd_27239_half_map_2.map.gz | 21.7 MB 38 MB 39.8 MB 39.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27239 http://ftp.pdbj.org/pub/emdb/structures/EMD-27239 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27239 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27239 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27239_validation.pdf.gz emd_27239_validation.pdf.gz | 714.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27239_full_validation.pdf.gz emd_27239_full_validation.pdf.gz | 714.1 KB | 表示 | |
| XML形式データ |  emd_27239_validation.xml.gz emd_27239_validation.xml.gz | 14.9 KB | 表示 | |
| CIF形式データ |  emd_27239_validation.cif.gz emd_27239_validation.cif.gz | 19.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27239 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27239 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27239 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27239 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27239.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27239.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | focused refinement of CRBN~DDB1 with Mezigdomide | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||
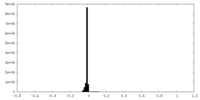
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27239_msk_1.map emd_27239_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
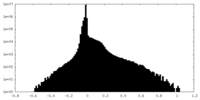
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_27239_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
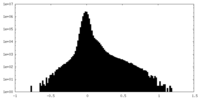
| 密度ヒストグラム |
-追加マップ: Postprocessed with DeepEMhancer
| ファイル | emd_27239_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Postprocessed with DeepEMhancer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_27239_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27239_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cereblon bound to CC-92480
| 全体 | 名称: Cereblon bound to CC-92480 |
|---|---|
| 要素 |
|
-超分子 #1: Cereblon bound to CC-92480
| 超分子 | 名称: Cereblon bound to CC-92480 / タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Cereblon
| 分子 | 名称: Cereblon / タイプ: protein_or_peptide / ID: 1 / 詳細: bound to CC-92480 mezigdomide ligand / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGEGDQQDA AHNMGNHLPL LPAESEEEDE MEVEDQDSKE AKKPNIINFD TSLPTSHTYL GADMEEFHGR TLHDDDSCQV IPVLPQVMMI LIPGQTLPLQ LFHPQEVSMV RNLIQKDRTF AVLAYSNVQE REAQFGTTAE IYAYREEQDF GIEIVKVKAI GRQRFKVLEL ...文字列: MAGEGDQQDA AHNMGNHLPL LPAESEEEDE MEVEDQDSKE AKKPNIINFD TSLPTSHTYL GADMEEFHGR TLHDDDSCQV IPVLPQVMMI LIPGQTLPLQ LFHPQEVSMV RNLIQKDRTF AVLAYSNVQE REAQFGTTAE IYAYREEQDF GIEIVKVKAI GRQRFKVLEL RTQSDGIQQA KVQILPECVL PSTMSAVQLE SLNKCQIFPS KPVSREDQCS YKWWQKYQKR KFHCANLTSW PRWLYSLYDA ETLMDRIKKQ LREWDENLKD DSLPSNPIDF SYRVAACLPI DDVLRIQLLK IGSAIQRLRC ELDIMNKCTS LCCKQCQETE ITTKNEIFSL SLCGPMAAYV NPHGYVHETL TVYKACNLNL IGRPSTEHSW FPGYAWTVAQ CKICASHIGW KFTATKKDMS PQKFWGLTRS ALLPTIPDTE DEISPDKVIL CL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 20 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 62.5 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z
Z Y
Y X
X