+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 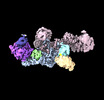 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EM map of the human UBR5 HECT-type E3 ubiquitin ligase in a dimeric form | |||||||||
 マップデータ マップデータ | Primary map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HECT / E3 ligase / Dimer / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoplasm protein quality control / heterochromatin boundary formation / cytoplasm protein quality control by the ubiquitin-proteasome system / protein K29-linked ubiquitination / nuclear protein quality control by the ubiquitin-proteasome system / HECT-type E3 ubiquitin transferase / protein K11-linked ubiquitination / DNA repair-dependent chromatin remodeling / ubiquitin-ubiquitin ligase activity / progesterone receptor signaling pathway ...cytoplasm protein quality control / heterochromatin boundary formation / cytoplasm protein quality control by the ubiquitin-proteasome system / protein K29-linked ubiquitination / nuclear protein quality control by the ubiquitin-proteasome system / HECT-type E3 ubiquitin transferase / protein K11-linked ubiquitination / DNA repair-dependent chromatin remodeling / ubiquitin-ubiquitin ligase activity / progesterone receptor signaling pathway / protein K48-linked ubiquitination / negative regulation of smoothened signaling pathway / ubiquitin binding / positive regulation of protein import into nucleus / protein polyubiquitination / positive regulation of canonical Wnt signaling pathway / ubiquitin protein ligase activity / proteasome-mediated ubiquitin-dependent protein catabolic process / DNA repair / DNA damage response / positive regulation of gene expression / chromatin / perinuclear region of cytoplasm / protein-containing complex / RNA binding / zinc ion binding / nucleoplasm / membrane / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
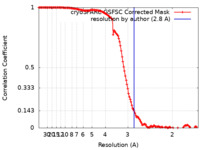
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Wang F / He Q / Lin G / Li H | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: Structure of the human UBR5 E3 ubiquitin ligase. 著者: Feng Wang / Qing He / Wenhu Zhan / Ziqi Yu / Efrat Finkin-Groner / Xiaojing Ma / Gang Lin / Huilin Li /  要旨: The human UBR5 is a single polypeptide chain homology to E6AP C terminus (HECT)-type E3 ubiquitin ligase essential for embryonic development in mammals. Dysregulated UBR5 functions like an ...The human UBR5 is a single polypeptide chain homology to E6AP C terminus (HECT)-type E3 ubiquitin ligase essential for embryonic development in mammals. Dysregulated UBR5 functions like an oncoprotein to promote cancer growth and metastasis. Here, we report that UBR5 assembles into a dimer and a tetramer. Our cryoelectron microscopy (cryo-EM) structures reveal that two crescent-shaped UBR5 monomers assemble head to tail to form the dimer, and two dimers bind face to face to form the cage-like tetramer with all four catalytic HECT domains facing the central cavity. Importantly, the N-terminal region of one subunit and the HECT of the other form an "intermolecular jaw" in the dimer. We show the jaw-lining residues are important for function, suggesting that the intermolecular jaw functions to recruit ubiquitin-loaded E2 to UBR5. Further work is needed to understand how oligomerization regulates UBR5 ligase activity. This work provides a framework for structure-based anticancer drug development and contributes to a growing appreciation of E3 ligase diversity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27201.map.gz emd_27201.map.gz | 216.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27201-v30.xml emd-27201-v30.xml emd-27201.xml emd-27201.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27201_fsc.xml emd_27201_fsc.xml | 13.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27201.png emd_27201.png | 76.7 KB | ||
| Filedesc metadata |  emd-27201.cif.gz emd-27201.cif.gz | 7.8 KB | ||
| その他 |  emd_27201_additional_1.map.gz emd_27201_additional_1.map.gz emd_27201_half_map_1.map.gz emd_27201_half_map_1.map.gz emd_27201_half_map_2.map.gz emd_27201_half_map_2.map.gz | 119.9 MB 226.3 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27201 http://ftp.pdbj.org/pub/emdb/structures/EMD-27201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27201 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27201.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27201.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Primary map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.828 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: RAW map
| ファイル | emd_27201_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
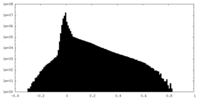
| 注釈 | RAW map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_27201_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
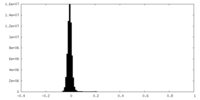
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_27201_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
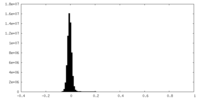
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Homodimer of human E3 ligase UBR5
| 全体 | 名称: Homodimer of human E3 ligase UBR5 |
|---|---|
| 要素 |
|
-超分子 #1: Homodimer of human E3 ligase UBR5
| 超分子 | 名称: Homodimer of human E3 ligase UBR5 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: UBR5 expressed in insect cell |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 610 KDa |
-分子 #1: E3 ubiquitin-protein ligase UBR5
| 分子 | 名称: E3 ubiquitin-protein ligase UBR5 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: HECT-type E3 ubiquitin transferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 310.604625 KDa |
| 組換発現 | 生物種: Insect expression vector pBlueBachsGCA1 (その他) |
| 配列 | 文字列: DYKDDDKMTS IHFVVHPLPG TEDQLNDRLR EVSEKLNKYN LNSHPPLNVL EQATIKQCVV GPNHAAFLLE DGRVCRIGFS VQPDRLELG KPDNNDGSKL NSNSGAGRTS RPGRTSDSPW FLSGSETLGR LAGNTLGSRW SSGVGGSGGG SSGRSSAGAR D SRRQTRVI ...文字列: DYKDDDKMTS IHFVVHPLPG TEDQLNDRLR EVSEKLNKYN LNSHPPLNVL EQATIKQCVV GPNHAAFLLE DGRVCRIGFS VQPDRLELG KPDNNDGSKL NSNSGAGRTS RPGRTSDSPW FLSGSETLGR LAGNTLGSRW SSGVGGSGGG SSGRSSAGAR D SRRQTRVI RTGRDRGSGL LGSQPQPVIP ASVIPEELIS QAQVVLQGKS RSVIIRELQR TNLDVNLAVN NLLSRDDEDG DD GDDTASE SYLPGEDLMS LLDADIHSAH PSVIIDADAM FSEDISYFGY PSFRRSSLSR LGSSRVLLLP LERDSELLRE RES VLRLRE RRWLDGASFD NERGSTSKEG EPNLDKKNTP VQSPVSLGED LQWWPDKDGT KFICIGALYS ELLAVSSKGE LYQW KWSES EPYRNAQNPS LHHPRATFLG LTNEKIVLLS ANSIRATVAT ENNKVATWVD ETLSSVASKL EHTAQTYSEL QGERI VSLH CCALYTCAQL ENSLYWWGVV PFSQRKKMLE KARAKNKKPK SSAGISSMPN ITVGTQVCLR NNPLYHAGAV AFSISA GIP KVGVLMESVW NMNDSCRFQL RSPESLKNME KASKTTEAKP ESKQEPVKTE MGPPPSPAST CSDASSIASS ASMPYKR RR STPAPKEEEK VNEEQWSLRE VVFVEDVKNV PVGKVLKVDG AYVAVKFPGT SSNTNCQNSS GPDADPSSLL QDCRLLRI D ELQVVKTGGT PKVPDCFQRT PKKLCIPEKT EILAVNVDSK GVHAVLKTGN WVRYCIFDLA TGKAEQENNF PTSSIAFLG QNERNVAIFT AGQESPIILR DGNGTIYPMA KDCMGGIRDP DWLDLPPISS LGMGVHSLIN LPANSTIKKK AAVIIMAVEK QTLMQHILR CDYEACRQYL MNLEQAVVLE QNLQMLQTFI SHRCDGNRNI LHACVSVCFP TSNKETKEEE EAERSERNTF A ERLSAVEA IANAISVVSS NGPGNRAGSS SSRSLRLREM MRRSLRAAGL GRHEAGASSS DHQDPVSPPI APPSWVPDPP AM DPDGDID FILAPAVGSL TTAATGTGQG PSTSTIPGPS TEPSVVESKD RKANAHFILK LLCDSVVLQP YLRELLSAKD ARG MTPFMS AVSGRAYPAA ITILETAQKI AKAEISSSEK EEDVFMGMVC PSGTNPDDSP LYVLCCNDTC SFTWTGAEHI NQDI FECRT CGLLESLCCC TECARVCHKG HDCKLKRTSP TAYCDCWEKC KCKTLIAGQK SARLDLLYRL LTATNLVTLP NSRGE HLLL FLVQTVARQT VEHCQYRPPR IREDRNRKTA SPEDSDMPDH DLEPPRFAQL ALERVLQDWN ALKSMIMFGS QENKDP LSA SSRIGHLLPE EQVYLNQQSG TIRLDCFTHC LIVKCTADIL LLDTLLGTLV KELQNKYTPG RREEAIAVTM RFLRSVA RV FVILSVEMAS SKKKNNFIPQ PIGKCKRVFQ ALLPYAVEEL CNVAESLIVP VRMGIARPTA PFTLASTSID AMQGSEEL F SVEPLPPRPS SDQSSSSSQS QSSYIIRNPQ QRRISQSQPV RGRDEEQDDI VSADVEEVEV VEGVAGEEDH HDEQEEHGE ENAEAEGQHD EHDEDGSDME LDLLAAAETE SDSESNHSNQ DNASGRRSVV TAATAGSEAG ASSVPAFFSE DDSQSNDSSD SDSSSSQSD DIEQETFMLD EPLERTTNSS HANGAAQAPR SMQWAVRNTQ HQRAASTAPS STSTPAASSA GLIYIDPSNL R RSGTISTS AAAAAAALEA SNASSYLTSA SSLARAYSIV IRQISDLMGL IPKYNHLVYS QIPAAVKLTY QDAVNLQNYV EE KLIPTWN WMVSIMDSTE AQLRYGSALA SAGDPGHPNH PLHASQNSAR RERMTAREEA SLRTLEGRRR ATLLSARQGM MSA RGDFLN YALSLMRSHN DEHSDVLPVL DVCSLKHVAY VFQALIYWIK AMNQQTTLDT PQLERKRTRE LLELGIDNED SEHE NDDDT NQSATLNDKD DDSLPAETGQ NHPFFRRSDS MTFLGCIPPN PFEVPLAEAI PLADQPHLLQ PNARKEDLFG RPSQG LYSS SASSGKCLME VTVDRNCLEV LPTKMSYAAN LKNVMNMQNR QKKEGEEQPV LPEETESSKP GPSAHDLAAQ LKSSLL AEI GLTESEGPPL TSFRPQCSFM GMVISHDMLL GRWRLSLELF GRVFMEDVGA EPGSILTELG GFEVKESKFR REMEKLR NQ QSRDLSLEVD RDRDLLIQQT MRQLNNHFGR RCATTPMAVH RVKVTFKDEP GEGSGVARSF YTAIAQAFLS NEKLPNLE C IQNANKGTHT SLMQRLRNRG ERDRERERER EMRRSSGLRA GSRRDRDRDF RRQLSIDTRP FRPASEGNPS DDPEPLPAH RQALGERLYP RVQAMQPAFA SKITGMLLEL SPAQLLLLLA SEDSLRARVD EAMELIIAHG RENGADSILD LGLVDSSEKV QQENRKRHG SSRSVVDMDL DDTDDGDDNA PLFYQPGKRG FYTPRPGKNT EARLNCFRNI GRILGLCLLQ NELCPITLNR H VIKVLLGR KVNWHDFAFF DPVMYESLRQ LILASQSSDA DAVFSAMDLA FAIDLCKEEG GGQVELIPNG VNIPVTPQNV YE YVRKYAE HRMLVVAEQP LHAMRKGLLD VLPKNSLEDL TAEDFRLLVN GCGEVNVQML ISFTSFNDES GENAEKLLQF KRW FWSIVE KMSMTERQDL VYFWTSSPSL PASEEGFQPM PSITIRPPDD QHLPTANTCI SRLYVPLYSS KQILKQKLLL AIKT KNFGF V UniProtKB: E3 ubiquitin-protein ligase UBR5 |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 6 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 279 K / 装置: FEI VITROBOT MARK IV / 詳細: blot 2S, blot forth 2. | ||||||||||||
| 詳細 | freshly purified UBR5 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 193.0 K / 最高: 193.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 65.0 e/Å2 詳細: Images were collected in movie-mode at 75 frames per second |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 90.94 / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-8d4x: |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X