+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2676 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of bovine ComplexI | |||||||||
 マップデータ マップデータ | Reconstruction of bovine ComplexI | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NADH dehydrogenase / respiratory complex | |||||||||
| 生物種 |  | |||||||||
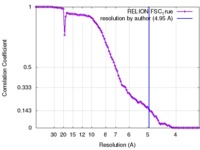
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.95 Å | |||||||||
 データ登録者 データ登録者 | Vinothkumar KR / Zhu J / Hirst J | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2014 ジャーナル: Nature / 年: 2014タイトル: Architecture of mammalian respiratory complex I. 著者: Kutti R Vinothkumar / Jiapeng Zhu / Judy Hirst /  要旨: Complex I (NADH:ubiquinone oxidoreductase) is essential for oxidative phosphorylation in mammalian mitochondria. It couples electron transfer from NADH to ubiquinone with proton translocation across ...Complex I (NADH:ubiquinone oxidoreductase) is essential for oxidative phosphorylation in mammalian mitochondria. It couples electron transfer from NADH to ubiquinone with proton translocation across the energy-transducing inner membrane, providing electrons for respiration and driving ATP synthesis. Mammalian complex I contains 44 different nuclear- and mitochondrial-encoded subunits, with a combined mass of 1 MDa. The 14 conserved 'core' subunits have been structurally defined in the minimal, bacterial complex, but the structures and arrangement of the 30 'supernumerary' subunits are unknown. Here we describe a 5 Å resolution structure of complex I from Bos taurus heart mitochondria, a close relative of the human enzyme, determined by single-particle electron cryo-microscopy. We present the structures of the mammalian core subunits that contain eight iron-sulphur clusters and 60 transmembrane helices, identify 18 supernumerary transmembrane helices, and assign and model 14 supernumerary subunits. Thus, we considerably advance knowledge of the structure of mammalian complex I and the architecture of its supernumerary ensemble around the core domains. Our structure provides insights into the roles of the supernumerary subunits in regulation, assembly and homeostasis, and a basis for understanding the effects of mutations that cause a diverse range of human diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2676.map.gz emd_2676.map.gz | 78.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2676-v30.xml emd-2676-v30.xml emd-2676.xml emd-2676.xml | 10.2 KB 10.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_2676_fsc.xml emd_2676_fsc.xml | 8.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  ComplexI_image_forEMDB.tif ComplexI_image_forEMDB.tif | 377.5 KB | ||
| その他 |  emd_2676_half_map_1.map.gz emd_2676_half_map_1.map.gz emd_2676_half_map_2.map.gz emd_2676_half_map_2.map.gz | 65.5 MB 65.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2676 http://ftp.pdbj.org/pub/emdb/structures/EMD-2676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2676 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2676 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2676_validation.pdf.gz emd_2676_validation.pdf.gz | 329.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2676_full_validation.pdf.gz emd_2676_full_validation.pdf.gz | 328.6 KB | 表示 | |
| XML形式データ |  emd_2676_validation.xml.gz emd_2676_validation.xml.gz | 11 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2676 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2676 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2676 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2676.map.gz / 形式: CCP4 / 大きさ: 81.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2676.map.gz / 形式: CCP4 / 大きさ: 81.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of bovine ComplexI | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.717 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 2676 half map 1.map
| ファイル | emd_2676_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 2676 half map 2.map
| ファイル | emd_2676_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : NADH:ubiquinone oxidoreductase
| 全体 | 名称: NADH:ubiquinone oxidoreductase |
|---|---|
| 要素 |
|
-超分子 #1000: NADH:ubiquinone oxidoreductase
| 超分子 | 名称: NADH:ubiquinone oxidoreductase / タイプ: sample / ID: 1000 詳細: The enzyme was isolated from bovine heart mitochondria in detergent micelles and imaged on ice as single particles 集合状態: 1 / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1 MDa / 理論値: 1 MDa |
-分子 #1: NADH:ubiquinone oxidoreductase
| 分子 | 名称: NADH:ubiquinone oxidoreductase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Complex I / コピー数: 1 / 集合状態: 1 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 1 MDa / 理論値: 1 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 20mM Tris-HCl, 150 mM NaCl, 0.03% Cymal-7 |
| グリッド | 詳細: 300 mesh holey-carbon Quantifoil gold grid R 0.6/1 glow discharged in air |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 100 K / 装置: OTHER 手法: The specimen was vitrified in an environmental plunge-freeze apparatus (Bellare et al, J.Electr. Micros. Tech., 1988, 10, 87-111). Blot for 15-18 seconds after the diameter of the blotted ...手法: The specimen was vitrified in an environmental plunge-freeze apparatus (Bellare et al, J.Electr. Micros. Tech., 1988, 10, 87-111). Blot for 15-18 seconds after the diameter of the blotted meniscus ceases to expand and plunged. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80 K / 最高: 90 K / 平均: 85 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 120,000 times magnification once at the start of data collection |
| 詳細 | Exposure intensity set to give 50 electron/pixel/second at the detector, which translates to ~17 electrons/square_Angstrom/second at the specimen. Each image was exposed for 4 seconds. |
| 日付 | 2013年10月21日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) デジタル化 - サンプリング間隔: 14 µm / 実像数: 1154 / 平均電子線量: 64 e/Å2 詳細: An in-house built system was used to intercept the frames from the detector at a rate of 18 frames per second. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 81495 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 47000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)