+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram averaged map of hACE2 dimers on the surface of extracellular vesicles | ||||||||||||
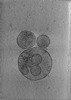
 マップデータ マップデータ | Final subtomogram averaged map of hACE2 on the surface of extracellular vesicles. | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
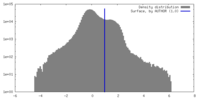
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 19.0 Å | ||||||||||||
 データ登録者 データ登録者 | Marcink TC / Kicmal T / Armbruster E / Zhang Z / Zipursky G / Idris M / Khao J / McGill G / Gallagher T / Porotto M ...Marcink TC / Kicmal T / Armbruster E / Zhang Z / Zipursky G / Idris M / Khao J / McGill G / Gallagher T / Porotto M / des Georges A / Moscona A | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Intermediates in SARS-CoV-2 spike-mediated cell entry. 著者: Tara C Marcink / Thomas Kicmal / Emily Armbruster / Zhening Zhang / Gillian Zipursky / Kate L Golub / Mohab Idris / Jonathan Khao / Jennifer Drew-Bear / Gael McGill / Tom Gallagher / Matteo ...著者: Tara C Marcink / Thomas Kicmal / Emily Armbruster / Zhening Zhang / Gillian Zipursky / Kate L Golub / Mohab Idris / Jonathan Khao / Jennifer Drew-Bear / Gael McGill / Tom Gallagher / Matteo Porotto / Amédée des Georges / Anne Moscona /   要旨: SARS-CoV-2 cell entry is completed after viral spike (S) protein-mediated membrane fusion between viral and host cell membranes. Stable prefusion and postfusion S structures have been resolved by ...SARS-CoV-2 cell entry is completed after viral spike (S) protein-mediated membrane fusion between viral and host cell membranes. Stable prefusion and postfusion S structures have been resolved by cryo-electron microscopy and cryo-electron tomography, but the refolding intermediates on the fusion pathway are transient and have not been examined. We used an antiviral lipopeptide entry inhibitor to arrest S protein refolding and thereby capture intermediates as S proteins interact with hACE2 and fusion-activating proteases on cell-derived target membranes. Cryo-electron tomography imaged both extended and partially folded intermediate states of S2, as well as a novel late-stage conformation on the pathway to membrane fusion. The intermediates now identified in this dynamic S protein-directed fusion provide mechanistic insights that may guide the design of CoV entry inhibitors. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26679.map.gz emd_26679.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26679-v30.xml emd-26679-v30.xml emd-26679.xml emd-26679.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26679.png emd_26679.png | 39.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26679 http://ftp.pdbj.org/pub/emdb/structures/EMD-26679 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26679 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26679 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26679_validation.pdf.gz emd_26679_validation.pdf.gz | 380.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26679_full_validation.pdf.gz emd_26679_full_validation.pdf.gz | 380 KB | 表示 | |
| XML形式データ |  emd_26679_validation.xml.gz emd_26679_validation.xml.gz | 5.5 KB | 表示 | |
| CIF形式データ |  emd_26679_validation.cif.gz emd_26679_validation.cif.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26679 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26679 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26679 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26679 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26679.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26679.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
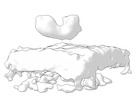
| 注釈 | Final subtomogram averaged map of hACE2 on the surface of extracellular vesicles. | ||||||||||||||||||||||||||||||||||||
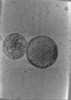
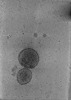
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.24 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Dimeric complex consisting of full-length membrane-bound human AC...
| 全体 | 名称: Dimeric complex consisting of full-length membrane-bound human ACE2 with a LgBiT tag. |
|---|---|
| 要素 |
|
-超分子 #1: Dimeric complex consisting of full-length membrane-bound human AC...
| 超分子 | 名称: Dimeric complex consisting of full-length membrane-bound human ACE2 with a LgBiT tag. タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293T / 組換プラスミド: pcDNA3.1 Homo sapiens (ヒト) / 組換細胞: HEK293T / 組換プラスミド: pcDNA3.1 |
-分子 #1: hACE2-LgBiT
| 分子 | 名称: hACE2-LgBiT / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSSSSWLLLS LVAVTAAQST IEEQAKTFLD KFNHEAEDLF YQSSLASWNY NTNITEENVQ NMNNAGDKWS AFLKEQSTLA QMYPLQEIQN LTVKLQLQAL QQNGSSVLSE DKSKRLNTIL NTMSTIYSTG KVCNPDNPQE CLLLEPGLNE IMANSLDYNE RLWAWESWRS ...文字列: MSSSSWLLLS LVAVTAAQST IEEQAKTFLD KFNHEAEDLF YQSSLASWNY NTNITEENVQ NMNNAGDKWS AFLKEQSTLA QMYPLQEIQN LTVKLQLQAL QQNGSSVLSE DKSKRLNTIL NTMSTIYSTG KVCNPDNPQE CLLLEPGLNE IMANSLDYNE RLWAWESWRS EVGKQLRPLY EEYVVLKNEM ARANHYEDYG DYWRGDYEVN GVDGYDYSRG QLIEDVEHTF EEIKPLYEHL HAYVRAKLMN AYPSYISPIG CLPAHLLGDM WGRFWTNLYS LTVPFGQKPN IDVTDAMVDQ AWDAQRIFKE AEKFFVSVGL PNMTQGFWEN SMLTDPGNVQ KAVCHPTAWD LGKGDFRILM CTKVTMDDFL TAHHEMGHIQ YDMAYAAQPF LLRNGANEGF HEAVGEIMSL SAATPKHLKS IGLLSPDFQE DNETEINFLL KQALTIVGTL PFTYMLEKWR WMVFKGEIPK DQWMKKWWEM KREIVGVVEP VPHDETYCDP ASLFHVSNDY SFIRYYTRTL YQFQFQEALC QAAKHEGPLH KCDISNSTEA GQKLFNMLRL GKSEPWTLAL ENVVGAKNMN VRPLLNYFEP LFTWLKDQNK NSFVGWSTDW SPYADQSIKV RISLKSALGD KAYEWNDNEM YLFRSSVAYA MRQYFLKVKN QMILFGEEDV RVANLKPRIS FNFFVTAPKN VSDIIPRTEV EKAIRMSRSR INDAFRLNDN SLEFLGIQPT LGPPNQPPVS IWLIVFGVVM GVIVVGIVIL IFTGIRDRKK KNKARSGENP YASIDISKGE NNPGFQNTDD VQTSF |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 3.0 nm |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum SE / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 2.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 4.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 19.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: Dynamo (ver. 1.1.514) / 使用したサブトモグラム数: 315 |
|---|---|
| 抽出 | トモグラム数: 1 / 使用した粒子像数: 1 |
| CTF補正 | ソフトウェア - 名称: Warp (ver. 1.0.9) |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)