+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
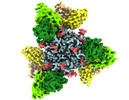
| タイトル | Native Lassa glycoprotein in complex with neutralizing antibodies 12.1F and 37.2D | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / receptor-mediated endocytosis of virus by host cell / host cell endoplasmic reticulum membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / membrane / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Lassa virus (ウイルス) Lassa virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.75 Å | |||||||||
 データ登録者 データ登録者 | Li H / Saphire EO | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Transl Med / 年: 2022 ジャーナル: Sci Transl Med / 年: 2022タイトル: A cocktail of protective antibodies subverts the dense glycan shield of Lassa virus. 著者: Haoyang Li / Tierra Buck / Michelle Zandonatti / Jieyun Yin / Alex Moon-Walker / Jingru Fang / Anatoliy Koval / Megan L Heinrich / Megan M Rowland / Ruben Diaz Avalos / Sharon L Schendel / ...著者: Haoyang Li / Tierra Buck / Michelle Zandonatti / Jieyun Yin / Alex Moon-Walker / Jingru Fang / Anatoliy Koval / Megan L Heinrich / Megan M Rowland / Ruben Diaz Avalos / Sharon L Schendel / Diptiben Parekh / Dawid Zyla / Adrian Enriquez / Stephanie Harkins / Brian Sullivan / Victoria Smith / Onyeka Chukwudozie / Reika Watanabe / James E Robinson / Robert F Garry / Luis M Branco / Kathryn M Hastie / Erica Ollmann Saphire /  要旨: Developing potent therapeutics and effective vaccines are the ultimate goals in controlling infectious diseases. Lassa virus (LASV), the causative pathogen of Lassa fever (LF), infects hundreds of ...Developing potent therapeutics and effective vaccines are the ultimate goals in controlling infectious diseases. Lassa virus (LASV), the causative pathogen of Lassa fever (LF), infects hundreds of thousands annually, but effective antivirals or vaccines against LASV infection are still lacking. Furthermore, neutralizing antibodies against LASV are rare. Here, we describe biochemical analyses and high-resolution cryo-electron microscopy structures of a therapeutic cocktail of three broadly protective antibodies that target the LASV glycoprotein complex (GPC), previously identified from survivors of multiple LASV infections. Structural and mechanistic analyses reveal compatible neutralizing epitopes and complementary neutralization mechanisms that offer high potency, broad range, and resistance to escape. These antibodies either circumvent or exploit specific glycans comprising the extensive glycan shield of GPC. Further, they require mammalian glycosylation, native GPC cleavage, and proper GPC trimerization. These findings guided engineering of a next-generation GPC antigen suitable for future neutralizing antibody and vaccine discovery. Together, these results explain protective mechanisms of rare, broad, and potent antibodies and identify a strategy for the rational design of therapeutic modalities against LF and related infectious diseases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26655.map.gz emd_26655.map.gz | 254.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26655-v30.xml emd-26655-v30.xml emd-26655.xml emd-26655.xml | 20.7 KB 20.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26655.png emd_26655.png | 110.6 KB | ||
| その他 |  emd_26655_half_map_1.map.gz emd_26655_half_map_1.map.gz emd_26655_half_map_2.map.gz emd_26655_half_map_2.map.gz | 475.3 MB 475.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26655 http://ftp.pdbj.org/pub/emdb/structures/EMD-26655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26655 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26655_validation.pdf.gz emd_26655_validation.pdf.gz | 969.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26655_full_validation.pdf.gz emd_26655_full_validation.pdf.gz | 969.4 KB | 表示 | |
| XML形式データ |  emd_26655_validation.xml.gz emd_26655_validation.xml.gz | 18.8 KB | 表示 | |
| CIF形式データ |  emd_26655_validation.cif.gz emd_26655_validation.cif.gz | 22.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26655 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26655 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7uovMC  7uotC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26655.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26655.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.66 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_26655_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_26655_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Native Lassa glycoprotein in complex with 12.1F-scFv and 37.2D-scFv
| 全体 | 名称: Native Lassa glycoprotein in complex with 12.1F-scFv and 37.2D-scFv |
|---|---|
| 要素 |
|
-超分子 #1: Native Lassa glycoprotein in complex with 12.1F-scFv and 37.2D-scFv
| 超分子 | 名称: Native Lassa glycoprotein in complex with 12.1F-scFv and 37.2D-scFv タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|
-分子 #1: 12.1F heavy chain (variable domain)
| 分子 | 名称: 12.1F heavy chain (variable domain) / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 12.731153 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQESGAG LLKPSETLSL SCTVDGESFN GFFWTWIRQP PGKGLEWIGE INHLASTGYN PSLKSRVTIS VDTSKNQFSL KLTSVTAAD TAVYYCARGY SYGFAWPNYH YLDVW |
-分子 #2: 12.1F light chain (variable domain)
| 分子 | 名称: 12.1F light chain (variable domain) / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.602884 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EIVLTQSPAT LSLSPGERAT LSCRASQSVS SYLAWYQHKP GQAPRLLIYG ASKRATGIPS RFSGSGSGTD FSLTISSLEP EDFAVYYCQ HRSDWRTTFG QGTRLEI |
-分子 #3: Glycoprotein G1
| 分子 | 名称: Glycoprotein G1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa virus (ウイルス) / 株: Mouse/Sierra Leone/Josiah/1976 Lassa virus (ウイルス) / 株: Mouse/Sierra Leone/Josiah/1976 |
| 分子量 | 理論値: 29.064402 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGQIVTFFQE VPHVIEEVMN IVLIALSVLA VLKGLYNFAT CGLVGLVTFL LLCGRSCTTS LYKGVYELQT LELNMETLNM TMPLSCTKN NSHHYIMVGN ETGLELTLTN TSIINHKFCN LSDAHKKNLY DHALMSIIST FHLSIPNFNQ YEAMSCDFNG G KISVQYNL ...文字列: MGQIVTFFQE VPHVIEEVMN IVLIALSVLA VLKGLYNFAT CGLVGLVTFL LLCGRSCTTS LYKGVYELQT LELNMETLNM TMPLSCTKN NSHHYIMVGN ETGLELTLTN TSIINHKFCN LSDAHKKNLY DHALMSIIST FHLSIPNFNQ YEAMSCDFNG G KISVQYNL SHSYAGDAAN HCGTVANGVL QTFMRMAWGG SYIALDSGRG NWDCIMTSYQ YLIIQNTTWE DHCQFSRPSP IG YLGLLSQ RTRDIYISRR LL |
-分子 #4: Glycoprotein G2
| 分子 | 名称: Glycoprotein G2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lassa virus (ウイルス) / 株: Mouse/Sierra Leone/Josiah/1976 Lassa virus (ウイルス) / 株: Mouse/Sierra Leone/Josiah/1976 |
| 分子量 | 理論値: 26.797973 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GTFTWTLSDS EGKDTPGGYC LTRWMLIEAE LKCFGNTAVA KCNEKHDEEF CDMLRLFDFN KQAIQRLKAE AQTSIQLINK AVNALINDQ LIMKNHLRDI MGIPYCNYSK YWYLNHTTTG RTSLPKCWLV SNGSYLNETH FSDDIEQQAD NMITEMLQKE Y MERQGKTP ...文字列: GTFTWTLSDS EGKDTPGGYC LTRWMLIEAE LKCFGNTAVA KCNEKHDEEF CDMLRLFDFN KQAIQRLKAE AQTSIQLINK AVNALINDQ LIMKNHLRDI MGIPYCNYSK YWYLNHTTTG RTSLPKCWLV SNGSYLNETH FSDDIEQQAD NMITEMLQKE Y MERQGKTP LGLVDLFVFS TSFYLISIFL HLVKIPTHRH IVGKSCPKPH RLNHMGICSC GLYKQPGVPV KWKR |
-分子 #5: 37.2D light chain (variable domain)
| 分子 | 名称: 37.2D light chain (variable domain) / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.412506 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ETTLTQSPAT LSVSPGETAT LSCRASQNVI NNLAWYQQKP GQAPRLLIYG ASTRATGIPA RFSGSGSGTE FTLTISSMQS EDFAVYYCQ QYNDWPRSFG QGTRLD |
-分子 #6: 37.2D heavy chain (variable domain)
| 分子 | 名称: 37.2D heavy chain (variable domain) / タイプ: protein_or_peptide / ID: 6 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.946536 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVQSGAE VKKPGASVKV SCKASGYTFT KYGISWVRQA PGQGLEWMGW ISAFNGYTRY GQRFQGKVTM TTDTSTNTAS LEVRTLTSN DTAVYYCARQ YPDQYSSSGW PRLFAMDVWG QGTTVTV |
-分子 #13: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 13 / コピー数: 1 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.75 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 286170 |
|---|---|
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X