+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2648 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | stem cell factor-induced intact KIT dimers | |||||||||
 マップデータ マップデータ | Reconstruction of intact, kinase-dead KIT-SCF complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | KIT / c-Kit / SCF / stem cell factor / transmembrane / receptor tyrosine kinase / RTK | |||||||||
| 機能・相同性 | :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
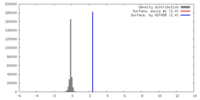
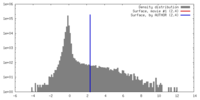
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 28.8 Å | |||||||||
 データ登録者 データ登録者 | Opatowsky Y / Bleichert F / Unger VM / Schlessinger J | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2014 ジャーナル: Proc Natl Acad Sci U S A / 年: 2014タイトル: Structure, domain organization, and different conformational states of stem cell factor-induced intact KIT dimers. 著者: Yarden Opatowsky / Irit Lax / Francisco Tomé / Franziska Bleichert / Vinzenz M Unger / Joseph Schlessinger /  要旨: Using electron microscopy and fitting of crystal structures, we present the 3D reconstruction of ligand-induced dimers of intact receptor tyrosine kinase, KIT. We observe that KIT protomers form ...Using electron microscopy and fitting of crystal structures, we present the 3D reconstruction of ligand-induced dimers of intact receptor tyrosine kinase, KIT. We observe that KIT protomers form close contacts throughout the entire structure of ligand-bound receptor dimers, and that the dimeric receptors adopt multiple, defined conformational states. Interestingly, the homotypic interactions in the membrane proximal Ig-like domain of the extracellular region differ from those observed in the crystal structure of the unconstrained extracellular regions. We observe two prevalent conformations in which the tyrosine kinase domains interact asymmetrically. The asymmetric arrangement of the cytoplasmic regions may represent snapshots of molecular interactions occurring during trans autophosphorylation. Moreover, the asymmetric arrangements may facilitate specific intermolecular interactions necessary for trans phosphorylation of different KIT autophosphorylation sites that are required for stimulation of kinase activity and recruitment of signaling proteins by activated KIT. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2648.map.gz emd_2648.map.gz | 971.5 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2648-v30.xml emd-2648-v30.xml emd-2648.xml emd-2648.xml | 11 KB 11 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2648.png emd_2648.png | 501.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2648 http://ftp.pdbj.org/pub/emdb/structures/EMD-2648 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2648 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2648 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2648_validation.pdf.gz emd_2648_validation.pdf.gz | 198.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2648_full_validation.pdf.gz emd_2648_full_validation.pdf.gz | 197.8 KB | 表示 | |
| XML形式データ |  emd_2648_validation.xml.gz emd_2648_validation.xml.gz | 4.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2648 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2648 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2648 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2648 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2648.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2648.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of intact, kinase-dead KIT-SCF complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Intact human KIT dimer in complex with stem cell factor (SCF)dimer
| 全体 | 名称: Intact human KIT dimer in complex with stem cell factor (SCF)dimer |
|---|---|
| 要素 |
|
-超分子 #1000: Intact human KIT dimer in complex with stem cell factor (SCF)dimer
| 超分子 | 名称: Intact human KIT dimer in complex with stem cell factor (SCF)dimer タイプ: sample / ID: 1000 / 集合状態: one dimer of SCF binds to KIT dimer / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 290 KDa / 手法: SDS-PAGE |
-分子 #1: stem cell growth factor
| 分子 | 名称: stem cell growth factor / タイプ: protein_or_peptide / ID: 1 / Name.synonym: SCF / コピー数: 2 / 集合状態: dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane |
| 分子量 | 理論値: 15 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: UNIPROTKB: P21583 |
-分子 #2: KIT
| 分子 | 名称: KIT / タイプ: protein_or_peptide / ID: 2 / 集合状態: dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Plasma membrane |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 200mM NaCl, 20mM HEPES,15% glycerol, 0.05% DDM |
| 染色 | タイプ: NEGATIVE 詳細: grids with adsorbed protein were floated on 3 drops of 1% uranyl formate for 30 seconds each |
| グリッド | 詳細: 400 mesh copper grid with continuous thin carbon support over holy carbon film |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 日付 | 2008年11月27日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: OTHER / 実像数: 400 / 平均電子線量: 20 e/Å2 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt angle max: 55 |
- 画像解析
画像解析
| 詳細 | Particles of tilt pairs (at 0 and 55 degrees)were manually selected using WEB. Initial reference-free alignment and classification of the untilted views was performed using IMAGIC-5. A representative class average was used for reference-based alignment, followed by cycles of multireference alignment and classification. Three out of 8 classes were used in random conical tilt reconstruction using SPIDER. |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 28.8 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER / 使用した粒子像数: 1900 |
| 最終 2次元分類 | クラス数: 3 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The domains were separately fitted by manual docking using Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The domains were separately fitted by manual docking using Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)