+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Photosynthetic assembly of Chlorobaculum tepidum (RC-FMO2) | |||||||||
 マップデータ マップデータ | RC-FMO2 map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | FMO / reaction center / single-particle cryo-EM / bacteriochlorophyll / photosynthesis / electron transport chain / energy transfer | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thylakoid / bacteriochlorophyll binding / iron-sulfur cluster binding / photosynthesis / electron transfer activity / heme binding / membrane / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chlorobaculum tepidum TLS (バクテリア) Chlorobaculum tepidum TLS (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.08 Å | |||||||||
 データ登録者 データ登録者 | Puskar R / Truong CD / Swain K / Li S / Cheng K-W / Wang TY / Poh Y-P / Liu H / Chou T-F / Nannenga B / Chiu P-L | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Molecular asymmetry of a photosynthetic supercomplex from green sulfur bacteria. 著者: Ryan Puskar / Chloe Du Truong / Kyle Swain / Saborni Chowdhury / Ka-Yi Chan / Shan Li / Kai-Wen Cheng / Ting Yu Wang / Yu-Ping Poh / Yuval Mazor / Haijun Liu / Tsui-Fen Chou / Brent L Nannenga / Po-Lin Chiu /  要旨: The photochemical reaction center (RC) features a dimeric architecture for charge separation across the membrane. In green sulfur bacteria (GSB), the trimeric Fenna-Matthews-Olson (FMO) complex ...The photochemical reaction center (RC) features a dimeric architecture for charge separation across the membrane. In green sulfur bacteria (GSB), the trimeric Fenna-Matthews-Olson (FMO) complex mediates the transfer of light energy from the chlorosome antenna complex to the RC. Here we determine the structure of the photosynthetic supercomplex from the GSB Chlorobaculum tepidum using single-particle cryogenic electron microscopy (cryo-EM) and identify the cytochrome c subunit (PscC), two accessory protein subunits (PscE and PscF), a second FMO trimeric complex, and a linker pigment between FMO and the RC core. The protein subunits that are assembled with the symmetric RC core generate an asymmetric photosynthetic supercomplex. One linker bacteriochlorophyll (BChl) is located in one of the two FMO-PscA interfaces, leading to differential efficiencies of the two energy transfer branches. The two FMO trimeric complexes establish two different binding interfaces with the RC cytoplasmic surface, driven by the associated accessory subunits. This structure of the GSB photosynthetic supercomplex provides mechanistic insight into the light excitation energy transfer routes and a possible evolutionary transition intermediate of the bacterial photosynthetic supercomplex from the primitive homodimeric RC. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_26471.map.gz emd_26471.map.gz | 168.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-26471-v30.xml emd-26471-v30.xml emd-26471.xml emd-26471.xml | 28.9 KB 28.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_26471.png emd_26471.png | 68.1 KB | ||
| Filedesc metadata |  emd-26471.cif.gz emd-26471.cif.gz | 7.9 KB | ||
| その他 |  emd_26471_half_map_1.map.gz emd_26471_half_map_1.map.gz emd_26471_half_map_2.map.gz emd_26471_half_map_2.map.gz | 165.3 MB 165.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-26471 http://ftp.pdbj.org/pub/emdb/structures/EMD-26471 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26471 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-26471 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_26471_validation.pdf.gz emd_26471_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_26471_full_validation.pdf.gz emd_26471_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_26471_validation.xml.gz emd_26471_validation.xml.gz | 14.7 KB | 表示 | |
| CIF形式データ |  emd_26471_validation.cif.gz emd_26471_validation.cif.gz | 17.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26471 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26471 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26471 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-26471 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7uebMC  7ueaC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_26471.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_26471.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RC-FMO2 map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: RC-FMO2 half map 1
| ファイル | emd_26471_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RC-FMO2 half map 1 | ||||||||||||
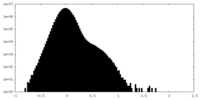
| 投影像・断面図 |
| ||||||||||||
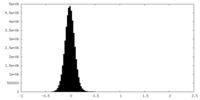
| 密度ヒストグラム |
-ハーフマップ: RC-FMO2 half map 2
| ファイル | emd_26471_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RC-FMO2 half map 2 | ||||||||||||
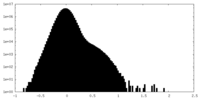
| 投影像・断面図 |
| ||||||||||||
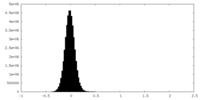
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Photosynthetic assembly (RC-FMO2)
+超分子 #1: Photosynthetic assembly (RC-FMO2)
+分子 #1: Photosystem P840 reaction center, large subunit
+分子 #2: Photosystem P840 reaction center iron-sulfur protein
+分子 #3: Cytochrome c
+分子 #4: P840 reaction center 17 kDa protein
+分子 #5: PscE
+分子 #6: PscF
+分子 #7: Bacteriochlorophyll a protein
+分子 #8: Bacteriochlorophyll A isomer
+分子 #9: Chlorophyll A ester
+分子 #10: BACTERIOCHLOROPHYLL A
+分子 #11: [(2R,3S,4S,5R,6R)-6-[(10E,12E,14E)-2,6,10,14,19,23-hexamethyl-25-...
+分子 #12: 2-[(1E,3E,5E,7E,9E,11E,13E,15E,17E,19E)-3,7,12,16,20,24-hexamethy...
+分子 #13: 1,2-DIPALMITOYL-PHOSPHATIDYL-GLYCEROLE
+分子 #14: 1,2-DISTEAROYL-MONOGALACTOSYL-DIGLYCERIDE
+分子 #15: IRON/SULFUR CLUSTER
+分子 #16: CALCIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-2/1 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 32898 / 平均露光時間: 6.0 sec. / 平均電子線量: 45.4 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 倍率(補正後): 47259 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X