+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2641 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-electron tomogram of Chlamydia trachomatis in contact with a eukaryotic host cell (U2OS). | |||||||||
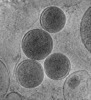
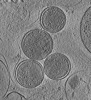
 マップデータ マップデータ | Chlamydia trachomatis elementary body in contact with a eukaryotic U2OS cell | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Chlamydia / type III secretion system / injectisome | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 | |||||||||
 データ登録者 データ登録者 | Nans A / Saibil HR / Hayward RD | |||||||||
 引用 引用 |  ジャーナル: Cell Microbiol / 年: 2014 ジャーナル: Cell Microbiol / 年: 2014タイトル: Pathogen-host reorganization during Chlamydia invasion revealed by cryo-electron tomography. 著者: Andrea Nans / Helen R Saibil / Richard D Hayward /  要旨: Invasion of host cells is a key early event during bacterial infection, but the underlying pathogen-host interactions are yet to be fully visualized in three-dimensional detail. We have captured ...Invasion of host cells is a key early event during bacterial infection, but the underlying pathogen-host interactions are yet to be fully visualized in three-dimensional detail. We have captured snapshots of the early stages of bacterial-mediated endocytosis in situ by exploiting the small size of chlamydial elementary bodies (EBs) for whole-cell cryo-electron tomography. Chlamydiae are obligate intracellular bacteria that infect eukaryotic cells and cause sexually transmitted infections and trachoma, the leading cause of preventable blindness. We demonstrate that Chlamydia trachomatis LGV2 EBs are intrinsically polarized. One pole is characterized by a tubular inner membrane invagination, while the other exhibits asymmetric periplasmic expansion to accommodate an array of type III secretion systems (T3SSs). Strikingly, EBs orient with their T3SS-containing pole facing target cells, enabling the T3SSs to directly contact the cellular plasma membrane. This contact induces enveloping macropinosomes, actin-rich filopodia and phagocytic cups to zipper tightly around the internalizing bacteria. Once encapsulated into tight early vacuoles, EB polarity and the T3SSs are lost. Our findings reveal previously undescribed structural transitions in both pathogen and host during the initial steps of chlamydial invasion. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2641.map.gz emd_2641.map.gz | 1.6 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2641-v30.xml emd-2641-v30.xml emd-2641.xml emd-2641.xml | 8 KB 8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2641.tif emd_2641.tif emd_2641_1.tif emd_2641_1.tif emd_2641_1_1.tif emd_2641_1_1.tif emd_2641_2.tif emd_2641_2.tif | 758.8 KB 763.3 KB 763.3 KB 758.8 KB | ||
| その他 |  emd_2641_additional_1.map.gz emd_2641_additional_1.map.gz | 922.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2641 http://ftp.pdbj.org/pub/emdb/structures/EMD-2641 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2641 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2641 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2641_validation.pdf.gz emd_2641_validation.pdf.gz | 175.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2641_full_validation.pdf.gz emd_2641_full_validation.pdf.gz | 174.5 KB | 表示 | |
| XML形式データ |  emd_2641_validation.xml.gz emd_2641_validation.xml.gz | 5.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2641 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2641 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2641 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2641 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2641.map.gz / 形式: CCP4 / 大きさ: 3.6 GB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) ダウンロード / ファイル: emd_2641.map.gz / 形式: CCP4 / 大きさ: 3.6 GB / タイプ: IMAGE STORED AS SIGNED INTEGER (2 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Chlamydia trachomatis elementary body in contact with a eukaryotic U2OS cell | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 2641 additional 1.map
| ファイル | emd_2641_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Chlamydia trachomatis elementary bodies in contact with eukaryoti...
| 全体 | 名称: Chlamydia trachomatis elementary bodies in contact with eukaryotic U2OS cell |
|---|---|
| 要素 |
|
-超分子 #1000: Chlamydia trachomatis elementary bodies in contact with eukaryoti...
| 超分子 | 名称: Chlamydia trachomatis elementary bodies in contact with eukaryotic U2OS cell タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|
-超分子 #1: Chlamydia trachomatis elementary bodies and a U2OS cell
| 超分子 | 名称: Chlamydia trachomatis elementary bodies and a U2OS cell タイプ: organelle_or_cellular_component / ID: 1 / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  株: LGV 2 / 細胞: U2OS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| グリッド | 詳細: Quantifoil 200 mesh gold (R 3.5/1) grids |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK III / 手法: Blot for 10 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 日付 | 2013年4月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 49 / 平均電子線量: 150 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 12.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -52 ° / Tilt series - Axis1 - Max angle: 42 ° / Tilt series - Axis1 - Angle increment: 2 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | IMOD for alignment and reconstruction. Spider for anisotropic diffusion. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / ソフトウェア - 名称:  IMOD / 詳細: Anisotropic diffusion was done in Spider. / 使用した粒子像数: 49 IMOD / 詳細: Anisotropic diffusion was done in Spider. / 使用した粒子像数: 49 |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)