+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the YejM/LapB complex | |||||||||
 マップデータ マップデータ | sharpened map with a B factor of -216 A2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | YejM / YciM / regulation / lipopolysaccharide synthesis / LpxC / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / lipopolysaccharide metabolic process / regulation of lipid biosynthetic process / membrane => GO:0016020 / iron ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
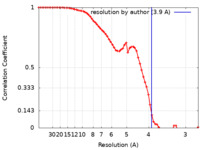
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Mi W / Shu S | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Regulatory mechanisms of lipopolysaccharide synthesis in Escherichia coli. 著者: Sheng Shu / Wei Mi /  要旨: Lipopolysaccharide (LPS) is an essential glycolipid and forms a protective permeability barrier for most Gram-negative bacteria. In E. coli, LPS levels are under feedback control, achieved by FtsH- ...Lipopolysaccharide (LPS) is an essential glycolipid and forms a protective permeability barrier for most Gram-negative bacteria. In E. coli, LPS levels are under feedback control, achieved by FtsH-mediated degradation of LpxC, which catalyzes the first committed step in LPS synthesis. FtsH is a membrane-bound AAA+ protease, and its protease activity toward LpxC is regulated by essential membrane proteins LapB and YejM. However, the regulatory mechanisms are elusive. We establish an in vitro assay to analyze the kinetics of LpxC degradation and demonstrate that LapB is an adaptor protein that utilizes its transmembrane helix to interact with FtsH and its cytoplasmic domains to recruit LpxC. Our YejM/LapB complex structure reveals that YejM is an anti-adaptor protein, competing with FtsH for LapB to inhibit LpxC degradation. Structural analysis unravels that LapB and LPS have overlapping binding sites in YejM. Thus, LPS levels control formation of the YejM/LapB complex to determine LpxC protein levels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25713.map.gz emd_25713.map.gz | 25.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25713-v30.xml emd-25713-v30.xml emd-25713.xml emd-25713.xml | 20.6 KB 20.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25713_fsc.xml emd_25713_fsc.xml | 6.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25713.png emd_25713.png | 77.8 KB | ||
| マスクデータ |  emd_25713_msk_1.map emd_25713_msk_1.map | 27 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-25713.cif.gz emd-25713.cif.gz | 11.1 KB | ||
| その他 |  emd_25713_additional_1.map.gz emd_25713_additional_1.map.gz emd_25713_half_map_1.map.gz emd_25713_half_map_1.map.gz emd_25713_half_map_2.map.gz emd_25713_half_map_2.map.gz | 13.4 MB 25.1 MB 25.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25713 http://ftp.pdbj.org/pub/emdb/structures/EMD-25713 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25713 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25713 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25713_validation.pdf.gz emd_25713_validation.pdf.gz | 735.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25713_full_validation.pdf.gz emd_25713_full_validation.pdf.gz | 734.7 KB | 表示 | |
| XML形式データ |  emd_25713_validation.xml.gz emd_25713_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_25713_validation.cif.gz emd_25713_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25713 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25713 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25713 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25713 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7t6dMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25713.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25713.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map with a B factor of -216 A2 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.346 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_25713_msk_1.map emd_25713_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
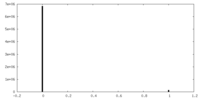
| 密度ヒストグラム |
-追加マップ: original 3D reconstruction without sharpening
| ファイル | emd_25713_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | original 3D reconstruction without sharpening | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
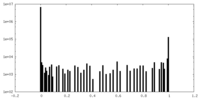
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_25713_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
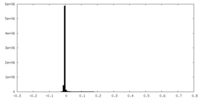
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_25713_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
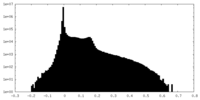
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : complex of YejM and YciM
| 全体 | 名称: complex of YejM and YciM |
|---|---|
| 要素 |
|
-超分子 #1: complex of YejM and YciM
| 超分子 | 名称: complex of YejM and YciM / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 225.556 kDa/nm |
-分子 #1: Lipopolysaccharide assembly protein B
| 分子 | 名称: Lipopolysaccharide assembly protein B / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 45.546953 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLELLFLLLP VAAAYGWYMG RRSAQQNKQD EANRLSRDYV AGVNFLLSNQ QDKAVDLFLD MLKEDTGTVE AHLTLGNLFR SRGEVDRAI RIHQTLMESA SLTYEQRLLA IQQLGRDYMA AGLYDRAEDM FNQLTDETDF RIGALQQLLQ IYQATSEWQK A IDVAERLV ...文字列: MLELLFLLLP VAAAYGWYMG RRSAQQNKQD EANRLSRDYV AGVNFLLSNQ QDKAVDLFLD MLKEDTGTVE AHLTLGNLFR SRGEVDRAI RIHQTLMESA SLTYEQRLLA IQQLGRDYMA AGLYDRAEDM FNQLTDETDF RIGALQQLLQ IYQATSEWQK A IDVAERLV KLGKDKQRVE IAHFYCELAL QHMASDDLDR AMTLLKKGAA ADKNSARVSI MMGRVFMAKG EYAKAVESLQ RV ISQDREL VSETLEMLQT CYQQLGKTAE WAEFLQRAVE ENTGADAELM LADIIEARDG SEAAQVYITR QLQRHPTMRV FHK LMDYHL NEAEEGRAKE SLMVLRDMVG EKVRSKPRYR CQKCGFTAYT LYWHCPSCRA WSTIKPIRGL DGLEHHHHHH UniProtKB: Lipopolysaccharide assembly protein B |
-分子 #2: Inner membrane protein YejM
| 分子 | 名称: Inner membrane protein YejM / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 67.36468 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVTHRQRYRE KVSQMVSWGH WFALFNILLS LVIGSRYLFI ADWPTTLAGR IYSYVSIIGH FSFLVFATYL LILFPLTFIV GSQRLMRFL SVILATAGMT LLLIDSEVFT RFHLHLNPIV WQLVINPDEN EMARDWQLMF ISVPVILLLE LVFATWSWQK L RSLTRRRR ...文字列: MVTHRQRYRE KVSQMVSWGH WFALFNILLS LVIGSRYLFI ADWPTTLAGR IYSYVSIIGH FSFLVFATYL LILFPLTFIV GSQRLMRFL SVILATAGMT LLLIDSEVFT RFHLHLNPIV WQLVINPDEN EMARDWQLMF ISVPVILLLE LVFATWSWQK L RSLTRRRR FARPLAAFLF IAFIASHVVY IWADANFYRP ITMQRANLPL SYPMTARRFL EKHGLLDAQE YQRRLIEQGN PD AVSVQYP LSELRYRDMG TGQNVLLITV DGLNYSRFEK QMPALAGFAE QNISFTRHMS SGNTTDNGIF GLFYGISPSY MDG ILSTRT PAALITALNQ QGYQLGLFSS DGFTSPLYRQ ALLSDFSMPS VRTQSDEQTA TQWINWLGRY AQEDNRWFSW VSFN GTNID DSNQQAFARK YSRAAGNVDD QINRVLNALR DSGKLDNTVV IITAGRGIPL SEEEETFDWS HGHLQVPLVI HWPGT PAQR INALTDHTDL MTTLMQRLLH VSTPASEYSQ GQDLFNPQRR HYWVTAADND TLAITTPKKT LVLNNNGKYR TYNLRG ERV KDEKPQLSLL LQVLTDEKRF IAN UniProtKB: Inner membrane protein YejM |
-分子 #3: 2-(HEXADECANOYLOXY)-1-[(PHOSPHONOOXY)METHYL]ETHYL HEXADECANOATE
| 分子 | 名称: 2-(HEXADECANOYLOXY)-1-[(PHOSPHONOOXY)METHYL]ETHYL HEXADECANOATE タイプ: ligand / ID: 3 / コピー数: 1 / 式: LPP |
|---|---|
| 分子量 | 理論値: 648.891 Da |
| Chemical component information |  ChemComp-LPP: |
-分子 #4: (1R)-2-{[{[(2S)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
| 分子 | 名称: (1R)-2-{[{[(2S)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-1-[(PALMITOYLOXY)METHYL]ETHYL (11E)-OCTADEC-11-ENOATE タイプ: ligand / ID: 4 / コピー数: 1 / 式: PGV |
|---|---|
| 分子量 | 理論値: 749.007 Da |
| Chemical component information |  ChemComp-PGV: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 6 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 平均露光時間: 8.84 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 64000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7t6d: |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)