+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
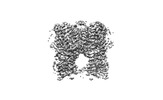
| タイトル | Cryo-EM structure of the full-length TRPV1 with RTx at 4 degrees Celsius, in an open state, class III | |||||||||
 マップデータ マップデータ | 4C_RTX_Open | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報temperature-gated ion channel activity / response to capsazepine / negative regulation of establishment of blood-brain barrier / sensory perception of mechanical stimulus / peptide secretion / urinary bladder smooth muscle contraction / detection of chemical stimulus involved in sensory perception of pain / cellular response to temperature stimulus / smooth muscle contraction involved in micturition / TRP channels ...temperature-gated ion channel activity / response to capsazepine / negative regulation of establishment of blood-brain barrier / sensory perception of mechanical stimulus / peptide secretion / urinary bladder smooth muscle contraction / detection of chemical stimulus involved in sensory perception of pain / cellular response to temperature stimulus / smooth muscle contraction involved in micturition / TRP channels / cellular response to acidic pH / excitatory extracellular ligand-gated monoatomic ion channel activity / thermoception / fever generation / detection of temperature stimulus involved in thermoception / glutamate secretion / negative regulation of systemic arterial blood pressure / chloride channel regulator activity / dendritic spine membrane / response to pH / monoatomic cation transmembrane transporter activity / extracellular ligand-gated monoatomic ion channel activity / cellular response to ATP / negative regulation of heart rate / temperature homeostasis / response to pain / cellular response to alkaloid / behavioral response to pain / diet induced thermogenesis / intracellularly gated calcium channel activity / cellular response to cytokine stimulus / detection of temperature stimulus involved in sensory perception of pain / negative regulation of mitochondrial membrane potential / ligand-gated monoatomic ion channel activity / calcium ion import across plasma membrane / monoatomic cation channel activity / monoatomic ion transmembrane transport / sensory perception of pain / : / phosphatidylinositol binding / cellular response to nerve growth factor stimulus / calcium ion transmembrane transport / phosphoprotein binding / microglial cell activation / calcium channel activity / lipid metabolic process / cellular response to growth factor stimulus / response to peptide hormone / transmembrane signaling receptor activity / calcium ion transport / positive regulation of nitric oxide biosynthetic process / cellular response to tumor necrosis factor / cellular response to heat / positive regulation of cytosolic calcium ion concentration / response to heat / postsynaptic membrane / protein homotetramerization / calmodulin binding / neuron projection / positive regulation of apoptotic process / external side of plasma membrane / neuronal cell body / dendrite / negative regulation of transcription by RNA polymerase II / ATP binding / identical protein binding / membrane / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.11 Å | |||||||||
 データ登録者 データ登録者 | Kwon DH / Suo Y / Lee S-Y | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Vanilloid-dependent TRPV1 opening trajectory from cryoEM ensemble analysis. 著者: Do Hoon Kwon / Feng Zhang / Justin G Fedor / Yang Suo / Seok-Yong Lee /  要旨: Single particle cryo-EM often yields multiple protein conformations within a single dataset, but experimentally deducing the temporal relationship of these conformers within a conformational ...Single particle cryo-EM often yields multiple protein conformations within a single dataset, but experimentally deducing the temporal relationship of these conformers within a conformational trajectory is not trivial. Here, we use thermal titration methods and cryo-EM in an attempt to obtain temporal resolution of the conformational trajectory of the vanilloid receptor TRPV1 with resiniferatoxin (RTx) bound. Based on our cryo-EM ensemble analysis, RTx binding to TRPV1 appears to induce intracellular gate opening first, followed by selectivity filter dilation, then pore loop rearrangement to reach the final open state. This apparent conformational wave likely arises from the concerted, stepwise, additive structural changes of TRPV1 over many subdomains. Greater understanding of the RTx-mediated long-range allostery of TRPV1 could help further the therapeutic potential of RTx, which is a promising drug candidate for pain relief associated with advanced cancer or knee arthritis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24638.map.gz emd_24638.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24638-v30.xml emd-24638-v30.xml emd-24638.xml emd-24638.xml | 16 KB 16 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24638.png emd_24638.png | 65.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24638 http://ftp.pdbj.org/pub/emdb/structures/EMD-24638 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24638 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24638 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24638_validation.pdf.gz emd_24638_validation.pdf.gz | 431.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24638_full_validation.pdf.gz emd_24638_full_validation.pdf.gz | 430.9 KB | 表示 | |
| XML形式データ |  emd_24638_validation.xml.gz emd_24638_validation.xml.gz | 6.2 KB | 表示 | |
| CIF形式データ |  emd_24638_validation.cif.gz emd_24638_validation.cif.gz | 7.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24638 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24638 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24638 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24638 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24638.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24638.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 4C_RTX_Open | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Transient receptor potential cation channel subfamily V member 1
| 全体 | 名称: Transient receptor potential cation channel subfamily V member 1 |
|---|---|
| 要素 |
|
-超分子 #1: Transient receptor potential cation channel subfamily V member 1
| 超分子 | 名称: Transient receptor potential cation channel subfamily V member 1 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 400 KDa |
-分子 #1: Transient receptor potential cation channel subfamily V member 1
| 分子 | 名称: Transient receptor potential cation channel subfamily V member 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 98.719758 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MEQRASLDSE ESESPPQENS CLDPPDRDPN CKPPPVKPHI FTTRSRTRLF GKGDSEEASP LDCPYEEGGL ASCPIITVSS VLTIQRPGD GPASVRPSSQ DSVSAGEKPP RLYDRRSIFD AVAQSNCQEL ESLLPFLQRS KKRLTDSEFK DPETGKTCLL K AMLNLHNG ...文字列: MEQRASLDSE ESESPPQENS CLDPPDRDPN CKPPPVKPHI FTTRSRTRLF GKGDSEEASP LDCPYEEGGL ASCPIITVSS VLTIQRPGD GPASVRPSSQ DSVSAGEKPP RLYDRRSIFD AVAQSNCQEL ESLLPFLQRS KKRLTDSEFK DPETGKTCLL K AMLNLHNG QNDTIALLLD VARKTDSLKQ FVNASYTDSY YKGQTALHIA IERRNMTLVT LLVENGADVQ AAANGDFFKK TK GRPGFYF GELPLSLAAC TNQLAIVKFL LQNSWQPADI SARDSVGNTV LHALVEVADN TVDNTKFVTS MYNEILILGA KLH PTLKLE EITNRKGLTP LALAASSGKI GVLAYILQRE IHEPECRHLS RKFTEWAYGP VHSSLYDLSC IDTCEKNSVL EVIA YSSSE TPNRHDMLLV EPLNRLLQDK WDRFVKRIFY FNFFVYCLYM IIFTAAAYYR PVEGLPPYKL KNTVGDYFRV TGEIL SVSG GVYFFFRGIQ YFLQRRPSLK SLFVDSYSEI LFFVQSLFML VSVVLYFSQR KEYVASMVFS LAMGWTNMLY YTRGFQ QMG IYAVMIEKMI LRDLCRFMFV YLVFLFGFST AVVTLIEDGK NNSLPMESTP HKCRGSACKP GNSYNSLYST CLELFKF TI GMGDLEFTEN YDFKAVFIIL LLAYVILTYI LLLNMLIALM GETVNKIAQE SKNIWKLQRA ITILDTEKSF LKCMRKAF R SGKLLQVGFT PDGKDDYRWC FRVDEVNWTT WNTNVGIINE DPGNCEGVKR TLSFSLRSGR VSGRNWKNFA LVPLLRDAS TRDRHATQQE EVQLKHYTGS LKPEDAEVFK DSMVPGEKEN SLEVLFQGPD YKDDDDKAHH HHHHHHHH |
-分子 #2: resiniferatoxin
| 分子 | 名称: resiniferatoxin / タイプ: ligand / ID: 2 / コピー数: 4 / 式: 6EU |
|---|---|
| 分子量 | 理論値: 628.708 Da |
| Chemical component information | 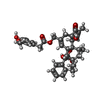 ChemComp-6EU: |
-分子 #3: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIOLEOYL-SN-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 3 / コピー数: 4 / 式: PCW |
|---|---|
| 分子量 | 理論値: 787.121 Da |
| Chemical component information |  ChemComp-PCW: |
-分子 #4: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 4 / コピー数: 4 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-分子 #5: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 5 / コピー数: 8 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 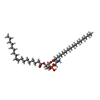 ChemComp-6OU: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.9 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY | ||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 2427 / 平均露光時間: 4.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー