+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
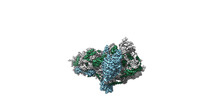
| タイトル | Ytm1 associated nascent 60S in fkbp39 deletion cells class 1A | |||||||||
 マップデータ マップデータ | Ytm1 associated nascent 60S in fkbp39 deletion cells class 1A | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Major pathway of rRNA processing in the nucleolus and cytosol / L13a-mediated translational silencing of Ceruloplasmin expression / Formation of a pool of free 40S subunits / GTP hydrolysis and joining of the 60S ribosomal subunit / SRP-dependent cotranslational protein targeting to membrane / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / PeBoW complex / rRNA primary transcript binding / sno(s)RNA-containing ribonucleoprotein complex ...Major pathway of rRNA processing in the nucleolus and cytosol / L13a-mediated translational silencing of Ceruloplasmin expression / Formation of a pool of free 40S subunits / GTP hydrolysis and joining of the 60S ribosomal subunit / SRP-dependent cotranslational protein targeting to membrane / Nonsense Mediated Decay (NMD) independent of the Exon Junction Complex (EJC) / Nonsense Mediated Decay (NMD) enhanced by the Exon Junction Complex (EJC) / PeBoW complex / rRNA primary transcript binding / sno(s)RNA-containing ribonucleoprotein complex / maturation of 5.8S rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / preribosome, small subunit precursor / maturation of 5.8S rRNA / cell division site / ribosomal large subunit binding / preribosome, large subunit precursor / ribonucleoprotein complex binding / ribosomal subunit export from nucleus / translation initiation factor activity / maturation of LSU-rRNA from tricistronic rRNA transcript (SSU-rRNA, 5.8S rRNA, LSU-rRNA) / 90S preribosome / nuclear periphery / assembly of large subunit precursor of preribosome / maturation of LSU-rRNA / ribosomal large subunit biogenesis / cytosolic ribosome assembly / maturation of SSU-rRNA / mitotic spindle / rRNA processing / large ribosomal subunit rRNA binding / cytosolic large ribosomal subunit / cytoplasmic translation / RNA helicase activity / rRNA binding / negative regulation of translation / ribosome / RNA helicase / structural constituent of ribosome / translation / cell cycle / GTPase activity / mRNA binding / nucleolus / GTP binding / ATP hydrolysis activity / RNA binding / zinc ion binding / nucleoplasm / ATP binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Bilokapic S / Halic M | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: Chromatin localization of nucleophosmin organizes ribosome biogenesis. 著者: Ilaria Ugolini / Silvija Bilokapic / Mylene Ferrolino / Josiah Teague / Yinxia Yan / Xuelin Zhou / Ashish Deshmukh / Michael White / Richard W Kriwacki / Mario Halic /   要旨: Ribosome biogenesis takes place in the nucleolus, a nuclear membrane-less organelle. Although well studied, it remains unknown how nascent ribosomal subunits separate from the central chromatin ...Ribosome biogenesis takes place in the nucleolus, a nuclear membrane-less organelle. Although well studied, it remains unknown how nascent ribosomal subunits separate from the central chromatin compartment and move to the outer granular component, where maturation occurs. We find that the Schizosaccharomyces pombe nucleophosmin-like protein Fkbp39 localizes to rDNA sites encoding the 60S subunit rRNA, and this localization contributes to its specific association with nascent 60S subunits. Fkbp39 dissociates from chromatin to bind nascent 60S subunits, causing the latter to partition away from chromatin and from nascent 40S subunits through liquid-liquid phase separation. In vivo, Fkbp39 binding directs the translocation of nascent 60S subunits toward the nucleophosmin-rich granular component. This process increases the efficiency of 60S subunit assembly, facilitating the incorporation of 60S RNA domain III. Thus, chromatin localization determines the specificity of nucleophosmin in sorting nascent ribosomal subunits and coordinates their movement into specialized assembly compartments within the nucleolus. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
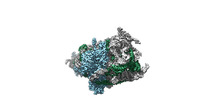
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24420.map.gz emd_24420.map.gz | 280.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24420-v30.xml emd-24420-v30.xml emd-24420.xml emd-24420.xml | 7.8 KB 7.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24420.png emd_24420.png | 23.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24420 http://ftp.pdbj.org/pub/emdb/structures/EMD-24420 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24420 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24420 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24420_validation.pdf.gz emd_24420_validation.pdf.gz | 348.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24420_full_validation.pdf.gz emd_24420_full_validation.pdf.gz | 347.6 KB | 表示 | |
| XML形式データ |  emd_24420_validation.xml.gz emd_24420_validation.xml.gz | 8 KB | 表示 | |
| CIF形式データ |  emd_24420_validation.cif.gz emd_24420_validation.cif.gz | 9.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24420 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24420 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24420 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24420 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8euyMC  8esqC  8esrC  8etcC  8etgC  8ethC  8etiC  8etjC  8eugC  8euiC  8eupC  8ev3C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24420.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24420.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Ytm1 associated nascent 60S in fkbp39 deletion cells class 1A | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : nascent 60S
| 全体 | 名称: nascent 60S |
|---|---|
| 要素 |
|
-超分子 #1: nascent 60S
| 超分子 | 名称: nascent 60S / タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 80.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 22000 |
|---|---|
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー