+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23817 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Glutamate synthase, glutamate dehydrogenase counter-enzyme complex | |||||||||
 マップデータ マップデータ | Sharpened map used in model refinement. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報glutamate synthase activity / oxidoreductase activity, acting on the CH-NH2 group of donors, NAD or NADP as acceptor / glutamate biosynthetic process / glutamate dehydrogenase (NAD+) activity / glutamate catabolic process / 3 iron, 4 sulfur cluster binding / glutamine metabolic process / iron-sulfur cluster binding / nucleotide binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.42 Å | |||||||||
 データ登録者 データ登録者 | Jayaraman V / Lee DJ / Elad N / Fraser JS / Tawfik DS | |||||||||
| 資金援助 |  イスラエル, イスラエル,  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2022 ジャーナル: Nat Chem Biol / 年: 2022タイトル: A counter-enzyme complex regulates glutamate metabolism in Bacillus subtilis. 著者: Vijay Jayaraman / D John Lee / Nadav Elad / Shay Vimer / Michal Sharon / James S Fraser / Dan S Tawfik /   要旨: Multi-enzyme assemblies composed of metabolic enzymes catalyzing sequential reactions are being increasingly studied. Here, we report the discovery of a 1.6 megadalton multi-enzyme complex from ...Multi-enzyme assemblies composed of metabolic enzymes catalyzing sequential reactions are being increasingly studied. Here, we report the discovery of a 1.6 megadalton multi-enzyme complex from Bacillus subtilis composed of two enzymes catalyzing opposite ('counter-enzymes') rather than sequential reactions: glutamate synthase (GltAB) and glutamate dehydrogenase (GudB), which make and break glutamate, respectively. In vivo and in vitro studies show that the primary role of complex formation is to inhibit the activity of GudB. Using cryo-electron microscopy, we elucidated the structure of the complex and the molecular basis of inhibition of GudB by GltAB. The complex exhibits unusual oscillatory progress curves and is necessary for both planktonic growth, in glutamate-limiting conditions, and for biofilm growth, in glutamate-rich media. The regulation of a key metabolic enzyme by complexing with its counter enzyme may thus enable cell growth under fluctuating glutamate concentrations. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23817.map.gz emd_23817.map.gz | 475.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23817-v30.xml emd-23817-v30.xml emd-23817.xml emd-23817.xml | 20.8 KB 20.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23817.png emd_23817.png | 140.1 KB | ||
| その他 |  emd_23817_additional_1.map.gz emd_23817_additional_1.map.gz | 474.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23817 http://ftp.pdbj.org/pub/emdb/structures/EMD-23817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23817 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23817_validation.pdf.gz emd_23817_validation.pdf.gz | 513.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23817_full_validation.pdf.gz emd_23817_full_validation.pdf.gz | 513 KB | 表示 | |
| XML形式データ |  emd_23817_validation.xml.gz emd_23817_validation.xml.gz | 8.3 KB | 表示 | |
| CIF形式データ |  emd_23817_validation.cif.gz emd_23817_validation.cif.gz | 9.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23817 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23817 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23817 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23817.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23817.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map used in model refinement. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.824 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Original map (not sharpened).
| ファイル | emd_23817_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Original map (not sharpened). | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
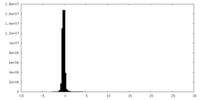
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : GudB6-GltA2-GltB2
| 全体 | 名称: GudB6-GltA2-GltB2 |
|---|---|
| 要素 |
|
-超分子 #1: GudB6-GltA2-GltB2
| 超分子 | 名称: GudB6-GltA2-GltB2 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 / 詳細: GudB hexamer binding two GltAB heterodimers. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 740 KDa |
-分子 #1: Glutamate dehydrogenase
| 分子 | 名称: Glutamate dehydrogenase / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 47.100715 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAADRNTGHT EEDKLDVLKS TQTVIHKALE KLGYPEEVYE LLKEPMRLLT VKIPVRMDDG SVKIFTGYRA QHNDSVGPTK GGIRFHPNV TEKEVKALSI WMSLKCGIID LPYGGGKGGI VCDPRDMSFR ELERLSRGYV RAISQIVGPT KDVPAPDVFT N SQIMAWMM ...文字列: MAADRNTGHT EEDKLDVLKS TQTVIHKALE KLGYPEEVYE LLKEPMRLLT VKIPVRMDDG SVKIFTGYRA QHNDSVGPTK GGIRFHPNV TEKEVKALSI WMSLKCGIID LPYGGGKGGI VCDPRDMSFR ELERLSRGYV RAISQIVGPT KDVPAPDVFT N SQIMAWMM DEYSRIDEFN SPGFITGKPL VLGGSHGRES ATAKGVTICI KEAAKKRGID IKGARVVVQG FGNAGSYLAK FM HDAGAKV VGISDAYGGL YDPEGLDIDY LLDRRDSFGT VTKLFNDTIT NQELLELDCD ILVPAAIENQ ITEENAHNIR AKI VVEAAN GPTTLEGTKI LSDRDILLVP DVLASAGGVT VSYFEWVQNN QGFYWSEEEV EEKLEKMMVK SFNNIYEMAN NRRI DMRLA AYMVGVRKMA EASRFRGWI |
-分子 #2: Glutamate synthase (NADPH) large chain
| 分子 | 名称: Glutamate synthase (NADPH) large chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 169.006375 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTYNQMPKAQ GLYRPEFEHD ACGIGLYAHL KGKQTHDIVK QGLKMLCQLD HRGGQGSDPD TGDGAGLLVQ IPDAFFRKEC KNINLPEKE RYGVGMVFFS QKEDERKKIE KQINALIEQE GQVVLGWRTV PVNVGKIGTV AQKSCPFVRQ VFIGASSDLK D NLSFERKL ...文字列: MTYNQMPKAQ GLYRPEFEHD ACGIGLYAHL KGKQTHDIVK QGLKMLCQLD HRGGQGSDPD TGDGAGLLVQ IPDAFFRKEC KNINLPEKE RYGVGMVFFS QKEDERKKIE KQINALIEQE GQVVLGWRTV PVNVGKIGTV AQKSCPFVRQ VFIGASSDLK D NLSFERKL YVIRKQAENW GVTEGLDFYF ASLSSQTIVY KGLLTPEQVD AFYSDLQDEA FVSAFALVHS RFSTNTFPTW ER AHPNRYL VHNGEINTLR GNINWMRARE QQFVSESFGE DLNKILPILN ADGSDSSILD NAFEFFVMAG RKPAHTAMML IPE PWTENT HMSKEKRAFY EYHSSLMEPW DGPTAISFTD GKQIGAILDR NGLRPARYYV TKDDYIIFSS EVGVIEVEQE NVLY KNRLE PGKMLLIDLE EGRIISDEEV KTQIATEYPY QKWLEEELVQ VNPDPESREE EQFSDLLTRQ KAFGYTYEDI QKYLI PVIK EGKDPLGSMG NDAPLAVLSD RAQSLFNYFK QLFAQVTNPP IDAIREQLVT STMTWLGAEG DLLHPSERNV RRIKLY TPV LSNEQFYALK TIVHPDLKSQ KIDVLFSEDL ERGLKDMFTQ AEKAISQGVS LLILSDKKMN ERLTPIPPLL AVSALHQ HL IRKGLRTKVS IIVESGEARE VHHFAALIGY GADAINPYLA YATYKQEIDE GRLDISYEEA VSKYGKSITE GVVKVMSK M GISTVQSYRG AQIFEAVGIS RDVIDRYFSG TASQLGGIDL QTIAEEAQRR HREAYQDDYS KTLEPGSDFQ WRNGGEHHA FNPKTIHTLQ WACRRNDYNL FKQYTKAADE ERIGFLRNLF AFDGNRKPLK LEEVESAESI VKRFKTGAMS FGSLSKEAHE ALAIAMNRL GGKSNSGEGG EDPKRFVPDE NGDDRRSAIK QIASGRFGVK SHYLVNADEL QIKMAQGAKP GEGGQLPGNK V YPWVADVR GSTPGVGLIS PPPHHDIYSI EDLAQLIHDL KNANRDARIS VKLVSKAGVG TIAAGVAKAT ADVIVISGYD GG TGASPKT SIKHTGLPWE LGLAEAHQTL MLNGLRDRVV LETDGKLMTG RDVVMAALLG AEEFGFATAP LVVLGCVMMR ACH LDTCPV GVATQNPELR KKFMGDPDHI VNYMLFIAEE VREYMAALGF KTFDEMIGRT DVLHVSERAK EHWKASQLDL STLL YQPEG VRTFQSPQNH KIDQSLDITT ILPAVQEAIE SGKEADISIE INNTNRVAGT ITGSEISKRY GEEGLPEDTI KLHFT GSAG QSFGAFVPKG MTLYLDGDSN DYVGKGLSGG KIIVKSSEGF NSASDDNVII GNVAFYGATS GEAYINGRAG ERFAVR NSG VNVVVEGIGD HGCEYMTGGS VVVLGDVGKN FAAGMSGGIA YVLTEDVKAF KRKCNLEMIL FESLEDEKEI QQIKAML ER HTAYTNSQKA EDLLDQWEDS VKKFVKVIPK NYKQMLASIE EQKAAGLSDE EAIMFAFEAN TKPKQNTAAS GQKQAVVQ |
-分子 #3: Glutamate synthase (NADPH) small chain
| 分子 | 名称: Glutamate synthase (NADPH) small chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 58.010598 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGKPTGFMEI KREKPAERDP LTRLKDWKEY SAPFSEEASK RQGARCMDCG TPFCQIGADI NGFTSGCPIY NLIPEWNGLV YRGRWKEAL ERLLKTNNFP EFTGRVCPAP CEGSCTLAIS DPAVSIKNIE RTIIDKGFEN GWIQPRIPKK RTGKKVAIVG S GPAGLASA ...文字列: MGKPTGFMEI KREKPAERDP LTRLKDWKEY SAPFSEEASK RQGARCMDCG TPFCQIGADI NGFTSGCPIY NLIPEWNGLV YRGRWKEAL ERLLKTNNFP EFTGRVCPAP CEGSCTLAIS DPAVSIKNIE RTIIDKGFEN GWIQPRIPKK RTGKKVAIVG S GPAGLASA DQLNQAGHSV TVFERADRAG GLLTYGIPNM KLEKGIVERR IKLLTQEGID FVTNTEIGVD ITADELKEQF DA VILCTGA QKQRDLLIEG RDSKGVHYAM DYLTLATKSY LDSNFKDKQF IDAKGKDVIV IGGGDTGADC VATALRQKAK SVH QFGKHP KLPPARTNDN MWPEQPHVFT LEYAYEEAEA KFGRDPREYS IQTTKMVADK NGKLKELHTI QMEKVKNEHG KYEF RELPG TEKVWPAQLV FIAIGFEGTE QPLLKQFGVN SVNNKISAAY GDYQTNIDGV FAAGDARRGQ SLIVWAINEG REVAR EVDR YLMGSSVLPG SWSHPQFEKG GGSGGGSGGS AWSHPQFENK |
-分子 #4: FLAVIN MONONUCLEOTIDE
| 分子 | 名称: FLAVIN MONONUCLEOTIDE / タイプ: ligand / ID: 4 / コピー数: 2 / 式: FMN |
|---|---|
| 分子量 | 理論値: 456.344 Da |
| Chemical component information |  ChemComp-FMN: |
-分子 #5: FE3-S4 CLUSTER
| 分子 | 名称: FE3-S4 CLUSTER / タイプ: ligand / ID: 5 / コピー数: 2 / 式: F3S |
|---|---|
| 分子量 | 理論値: 295.795 Da |
| Chemical component information |  ChemComp-F3S: |
-分子 #6: FLAVIN-ADENINE DINUCLEOTIDE
| 分子 | 名称: FLAVIN-ADENINE DINUCLEOTIDE / タイプ: ligand / ID: 6 / コピー数: 2 / 式: FAD |
|---|---|
| 分子量 | 理論値: 785.55 Da |
| Chemical component information |  ChemComp-FAD: |
-分子 #7: IRON/SULFUR CLUSTER
| 分子 | 名称: IRON/SULFUR CLUSTER / タイプ: ligand / ID: 7 / コピー数: 4 / 式: SF4 |
|---|---|
| 分子量 | 理論値: 351.64 Da |
| Chemical component information |  ChemComp-FS1: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.9 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 47.7 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | Real-space refinement involved iterative rounds of ISOLDE, Coot and PHENIX refinement. 1OFD and 6S6T were used as templates for homology modeling in SWISS-MODEL. | ||||||||
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT | ||||||||
| 得られたモデル |  PDB-7mfm: |
 ムービー
ムービー コントローラー
コントローラー




















 Z
Z Y
Y X
X