登録情報 データベース : EMDB / ID : EMD-23420タイトル CIII2CIV2 yeast supercomplex with cytochrome c. Non-uniform refinement. CIII2CIV2 yeast supercomplex with cytochrome c. Non-uniform refinement. 複合体 : yeast III-IV supercomplex with cytochrome c機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae (パン酵母)手法 / / 解像度 : 4.0 Å Moe A / Di Trani J / Rubinstein J / Brzezinski P 資金援助 Organization Grant number 国 Canadian Institutes of Health Research (CIHR) PJT162186 Knut and Alice Wallenberg Foundation 2019.0043
ジャーナル : Proc Natl Acad Sci U S A / 年 : 2021タイトル : Cryo-EM structure and kinetics reveal electron transfer by 2D diffusion of cytochrome in the yeast III-IV respiratory supercomplex.著者 : Agnes Moe / Justin Di Trani / John L Rubinstein / Peter Brzezinski / 要旨 : Energy conversion in aerobic organisms involves an electron current from low-potential donors, such as NADH and succinate, to dioxygen through the membrane-bound respiratory chain. Electron transfer ... Energy conversion in aerobic organisms involves an electron current from low-potential donors, such as NADH and succinate, to dioxygen through the membrane-bound respiratory chain. Electron transfer is coupled to transmembrane proton transport, which maintains the electrochemical proton gradient used to produce ATP and drive other cellular processes. Electrons are transferred from respiratory complexes III to IV (CIII and CIV) by water-soluble cytochrome (cyt.) In and some other organisms, these complexes assemble into larger CIIICIV supercomplexes, the functional significance of which has remained enigmatic. In this work, we measured the kinetics of the supercomplex cyt. -mediated QH:O oxidoreductase activity under various conditions. The data indicate that the electronic link between CIII and CIV is confined to the surface of the supercomplex. Single-particle electron cryomicroscopy (cryo-EM) structures of the supercomplex with cyt. show the positively charged cyt. bound to either CIII or CIV or along a continuum of intermediate positions. Collectively, the structural and kinetic data indicate that cyt. travels along a negatively charged patch on the supercomplex surface. Thus, rather than enhancing electron transfer rates by decreasing the distance that cyt. must diffuse in three dimensions, formation of the CIIICIV supercomplex facilitates electron transfer by two-dimensional (2D) diffusion of cyt. This mechanism enables the CIIICIV supercomplex to increase QH:O oxidoreductase activity and suggests a possible regulatory role for supercomplex formation in the respiratory chain. 履歴 登録 2021年2月4日 - ヘッダ(付随情報) 公開 2021年4月14日 - マップ公開 2021年4月14日 - 更新 2021年4月21日 - 現状 2021年4月21日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 カナダ,
カナダ,  スウェーデン, 2件
スウェーデン, 2件  引用
引用 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021
ジャーナル: Proc Natl Acad Sci U S A / 年: 2021

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_23420.map.gz
emd_23420.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-23420-v30.xml
emd-23420-v30.xml emd-23420.xml
emd-23420.xml EMDBヘッダ
EMDBヘッダ emd_23420_fsc.xml
emd_23420_fsc.xml FSCデータファイル
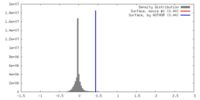
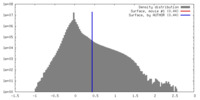
FSCデータファイル emd_23420.png
emd_23420.png http://ftp.pdbj.org/pub/emdb/structures/EMD-23420
http://ftp.pdbj.org/pub/emdb/structures/EMD-23420 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23420
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23420 emd_23420_validation.pdf.gz
emd_23420_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_23420_full_validation.pdf.gz
emd_23420_full_validation.pdf.gz emd_23420_validation.xml.gz
emd_23420_validation.xml.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23420
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23420 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23420
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23420 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_23420.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_23420.map.gz / 形式: CCP4 / 大きさ: 209.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素
 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)