+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23366 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Polyclonal Immune complex of Fab binding to BG505 SOSIP.664 from serum of rabbit 5724 at wk 26 | |||||||||
 マップデータ マップデータ | Polyclonal Immune complex of Fab binding to BG505 SOSIP.664 from serum of rabbit 5724 at wk 26 | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |    Human immunodeficiency virus (ヒト免疫不全ウイルス) Human immunodeficiency virus (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Pratap PP / Ward AB | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2021 ジャーナル: Elife / 年: 2021タイトル: High-resolution mapping of the neutralizing and binding specificities of polyclonal sera post-HIV Env trimer vaccination. 著者: Adam S Dingens / Payal Pratap / Keara Malone / Sarah K Hilton / Thomas Ketas / Christopher A Cottrell / Julie Overbaugh / John P Moore / P J Klasse / Andrew B Ward / Jesse D Bloom /  要旨: Mapping polyclonal serum responses is critical to rational vaccine design. However, most high-resolution mapping approaches involve isolating and characterizing individual antibodies, which ...Mapping polyclonal serum responses is critical to rational vaccine design. However, most high-resolution mapping approaches involve isolating and characterizing individual antibodies, which incompletely defines the polyclonal response. Here we use two complementary approaches to directly map the specificities of the neutralizing and binding antibodies of polyclonal anti-HIV-1 sera from rabbits immunized with BG505 Env SOSIP trimers. We used mutational antigenic profiling to determine how all mutations in Env affected viral neutralization and electron microscopy polyclonal epitope mapping (EMPEM) to directly visualize serum Fabs bound to Env trimers. The dominant neutralizing specificities were generally only a subset of the more diverse binding specificities. Additional differences between binding and neutralization reflected antigenicity differences between virus and soluble Env trimer. Furthermore, we refined residue-level epitope specificity directly from sera, revealing subtle differences across sera. Together, mutational antigenic profiling and EMPEM yield a holistic view of the binding and neutralizing specificity of polyclonal sera. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23366.map.gz emd_23366.map.gz | 20.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23366-v30.xml emd-23366-v30.xml emd-23366.xml emd-23366.xml | 8.8 KB 8.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23366.png emd_23366.png | 50.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23366 http://ftp.pdbj.org/pub/emdb/structures/EMD-23366 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23366 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23366 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23366_validation.pdf.gz emd_23366_validation.pdf.gz | 309.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23366_full_validation.pdf.gz emd_23366_full_validation.pdf.gz | 309.3 KB | 表示 | |
| XML形式データ |  emd_23366_validation.xml.gz emd_23366_validation.xml.gz | 5.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23366 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23366 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23366 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23366 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23366.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23366.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
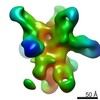
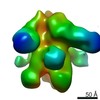
| 注釈 | Polyclonal Immune complex of Fab binding to BG505 SOSIP.664 from serum of rabbit 5724 at wk 26 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.98 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
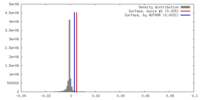
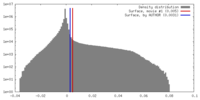
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Polyclonal Immune complex of Fab binding to BG505 SOSIP.664 from ...
| 全体 | 名称: Polyclonal Immune complex of Fab binding to BG505 SOSIP.664 from serum of rabbit 5724 at wk 26 |
|---|---|
| 要素 |
|
-超分子 #1: Polyclonal Immune complex of Fab binding to BG505 SOSIP.664 from ...
| 超分子 | 名称: Polyclonal Immune complex of Fab binding to BG505 SOSIP.664 from serum of rabbit 5724 at wk 26 タイプ: complex / ID: 1 / 親要素: 0 |
|---|
-超分子 #2: Polyclonal Fabs
| 超分子 | 名称: Polyclonal Fabs / タイプ: complex / ID: 2 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: BG505 SOSIP.664
| 超分子 | 名称: BG505 SOSIP.664 / タイプ: complex / ID: 3 / 親要素: 1 |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus (ヒト免疫不全ウイルス) Human immunodeficiency virus (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: 2% w/v uranyl formate |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS TALOS F200C |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DARK FIELD |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)