+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23343 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of KdpFABC in E2Pi state with AlF4 | |||||||||
 マップデータ マップデータ | Sharpened map from Non-Uniform Refinement in cryoSPARC | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報P-type K+ transporter / P-type potassium transmembrane transporter activity / potassium:proton antiporter complex / potassium ion-transporting ATPase complex / monoatomic cation transmembrane transport / potassium ion binding / potassium ion transmembrane transport / potassium ion transport / magnesium ion binding / ATP hydrolysis activity ...P-type K+ transporter / P-type potassium transmembrane transporter activity / potassium:proton antiporter complex / potassium ion-transporting ATPase complex / monoatomic cation transmembrane transport / potassium ion binding / potassium ion transmembrane transport / potassium ion transport / magnesium ion binding / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
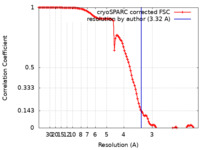
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.32 Å | |||||||||
 データ登録者 データ登録者 | Sweet ME / Larsen C / Pedersen BP / Stokes DL | |||||||||
| 資金援助 |  米国, 米国,  デンマーク, 2件 デンマーク, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: Structural basis for potassium transport in prokaryotes by KdpFABC. 著者: Marie E Sweet / Casper Larsen / Xihui Zhang / Michael Schlame / Bjørn P Pedersen / David L Stokes /   要旨: KdpFABC is an oligomeric K transport complex in prokaryotes that maintains ionic homeostasis under stress conditions. The complex comprises a channel-like subunit (KdpA) from the superfamily of K ...KdpFABC is an oligomeric K transport complex in prokaryotes that maintains ionic homeostasis under stress conditions. The complex comprises a channel-like subunit (KdpA) from the superfamily of K transporters and a pump-like subunit (KdpB) from the superfamily of P-type ATPases. Recent structural work has defined the architecture and generated contradictory hypotheses for the transport mechanism. Here, we use substrate analogs to stabilize four key intermediates in the reaction cycle and determine the corresponding structures by cryogenic electron microscopy. We find that KdpB undergoes conformational changes consistent with other representatives from the P-type superfamily, whereas KdpA, KdpC, and KdpF remain static. We observe a series of spherical densities that we assign as K or water and which define a pathway for K transport. This pathway runs through an intramembrane tunnel in KdpA and delivers ions to sites in the membrane domain of KdpB. Our structures suggest a mechanism where ATP hydrolysis is coupled to K transfer between alternative sites in KdpB, ultimately reaching a low-affinity site where a water-filled pathway allows release of K to the cytoplasm. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23343.map.gz emd_23343.map.gz | 118 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23343-v30.xml emd-23343-v30.xml emd-23343.xml emd-23343.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23343_fsc.xml emd_23343_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23343.png emd_23343.png | 98.9 KB | ||
| その他 |  emd_23343_half_map_1.map.gz emd_23343_half_map_1.map.gz emd_23343_half_map_2.map.gz emd_23343_half_map_2.map.gz | 116 MB 116 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23343 http://ftp.pdbj.org/pub/emdb/structures/EMD-23343 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23343 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23343 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23343_validation.pdf.gz emd_23343_validation.pdf.gz | 539.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23343_full_validation.pdf.gz emd_23343_full_validation.pdf.gz | 539.1 KB | 表示 | |
| XML形式データ |  emd_23343_validation.xml.gz emd_23343_validation.xml.gz | 18.8 KB | 表示 | |
| CIF形式データ |  emd_23343_validation.cif.gz emd_23343_validation.cif.gz | 24.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23343 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23343 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23343 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23343 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23343.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23343.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map from Non-Uniform Refinement in cryoSPARC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.059 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Un-sharpened half-map A from Non-Uniform Refinement in cryoSPARC
| ファイル | emd_23343_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Un-sharpened half-map A from Non-Uniform Refinement in cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Un-sharpened half-map B from Non-Uniform Refinement in cryoSPARC
| ファイル | emd_23343_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Un-sharpened half-map B from Non-Uniform Refinement in cryoSPARC | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : KdpFABC in E2Pi state with AlF4 in sample buffer
| 全体 | 名称: KdpFABC in E2Pi state with AlF4 in sample buffer |
|---|---|
| 要素 |
|
-超分子 #1: KdpFABC in E2Pi state with AlF4 in sample buffer
| 超分子 | 名称: KdpFABC in E2Pi state with AlF4 in sample buffer / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: KdpFABC complex solublized in decyl maltopyranoside (DM) detergent. |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 160 KDa |
-分子 #1: Potassium-transporting ATPase potassium-binding subunit
| 分子 | 名称: Potassium-transporting ATPase potassium-binding subunit タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAAQGFLLIA TFLLVLMVLA RPLGSGLARL INDIPLPGTT GVERVLFRAL GVSDREMNWK QYLCAILGLN MLGLAVLFFM LLGQHYLPL NPQQLPGLSW DLALNTAVSF VTNTNWRSYS GETTLSYFSQ MAGLTVQNFL SAASGIAVIF ALIRAFTRQS M STLGNAWV ...文字列: MAAQGFLLIA TFLLVLMVLA RPLGSGLARL INDIPLPGTT GVERVLFRAL GVSDREMNWK QYLCAILGLN MLGLAVLFFM LLGQHYLPL NPQQLPGLSW DLALNTAVSF VTNTNWRSYS GETTLSYFSQ MAGLTVQNFL SAASGIAVIF ALIRAFTRQS M STLGNAWV DLLRITLWVL VPVALLIALF FIQQGALQNF LPYQAVNTVE GAQQLLPMGP VASQEAIKML GTNGGGFFNA NS SHPFENP TALTNFVQML AIFLIPTALC FAFGEVMGDR RQGRMLLWAM SVIFVICVGV VMWAEVQGNP HLLALGTDSS INM EGKESR FGVLVSSLFA VVTTAASCGA VIAMHDSFTA LGGMVPMWLM QIGEVVFGGV GSGLYGMMLF VLLAVFIAGL MIGR TPEYL GKKIDVREMK LTALAILVTP TLVLMGAALA MMTDAGRSAM LNPGPHGFSE VLYAVSSAAN NNGSAFAGLS ANSPF WNCL LAFCMFVGRF GVIIPVMAIA GSLVSKKSQA ASSGTLPTHG PLFVGLLIGT VLLVGALTFI PALALGPVAE YLS |
-分子 #2: Potassium-transporting ATPase ATP-binding subunit
| 分子 | 名称: Potassium-transporting ATPase ATP-binding subunit / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO / EC番号: P-type K+ transporter |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSRKQLALFE PTLVVQALKE AVKKLNPQAQ WRNPVMFIVW IGSLLTTCIS IAMASGAMPG NALFSAAISG WLWITVLFAN FAEALAEGR SKAQANSLKG VKKTAFARKL REPKYGAAAD KVPADQLRKG DIVLVEAGDI IPCDGEVIEG GASVDESAIT G EAAPVIRE ...文字列: MSRKQLALFE PTLVVQALKE AVKKLNPQAQ WRNPVMFIVW IGSLLTTCIS IAMASGAMPG NALFSAAISG WLWITVLFAN FAEALAEGR SKAQANSLKG VKKTAFARKL REPKYGAAAD KVPADQLRKG DIVLVEAGDI IPCDGEVIEG GASVDESAIT G EAAPVIRE SGGDFASVTG GTRILSDWLV IECSVNPGET FLDRMIAMVE GAQRRKTPNE IALTILLIAL TIVFLLATAT LW PFSAWGG NAVSVTVLVA LLVCLIPTTI GGLLSAIGVA GMSRMLGANV IATSGRAVEA AGDVDVLLLD KTGTITLGNR QAS EFIPAQ GVDEKTLADA AQLASLADET PEGRSIVILA KQRFNLRERD VQSLHATFVP FTAQSRMSGI NIDNRMIRKG SVDA IRRHV EANGGHFPTD VDQKVDQVAR QGATPLVVVE GSRVLGVIAL KDIVKGGIKE RFAQLRKMGI KTVMITGDNR LTAAA IAAE AGVDDFLAEA TPEAKLALIR QYQAEGRLVA MTGDGTNDAP ALAQADVAVA MNSGTQAAKE AGNMVDLDSN PTKLIE VVH IGKQMLMTRG SLTTFSIAND VAKYFAIIPA AFAATYPQLN ALNIMCLHSP DSAILSAVIF NALIIVFLIP LALKGVS YK PLTASAMLRR NLWIYGLGGL LVPFIGIKVI DLLLTVCGLV |
-分子 #3: Potassium-transporting ATPase KdpC subunit
| 分子 | 名称: Potassium-transporting ATPase KdpC subunit / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGLRPALST FIFLLLITGG VYPLLTTVLG QWWFPWQANG SLIREGDTVR GSALIGQNFT GNGYFHGRPS ATAEMPYNPQ ASGGSNLAV SNPELDKLIA ARVAALRAAN PDASASVPVE LVTASASGLD NNITPQAAAW QIPRVAKARN LSVEQLTQLI A KYSQQPLV ...文字列: MSGLRPALST FIFLLLITGG VYPLLTTVLG QWWFPWQANG SLIREGDTVR GSALIGQNFT GNGYFHGRPS ATAEMPYNPQ ASGGSNLAV SNPELDKLIA ARVAALRAAN PDASASVPVE LVTASASGLD NNITPQAAAW QIPRVAKARN LSVEQLTQLI A KYSQQPLV KYIGQPVVNI VELNLALDKL DEGTGLVPRG SSHHHHHHHH |
-分子 #4: Potassium-transporting ATPase KdpF subunit
| 分子 | 名称: Potassium-transporting ATPase KdpF subunit / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAGVITGVL LVFLLLGYLV YALINAEAF |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.5 mg/mL | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: AlF4 was added to the protein after size-exclusion chromatography and incubated 1 hr prior to freezing | |||||||||||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY / 支持フィルム - Film thickness: 50.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR 詳細: PELCO easiGlow Glow Discharge Cleaning System used for glow discharge with default settings. | |||||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 3 uL of protein mixture applied to grid. Blot time 4 seconds, blot force 0, no wait before plunging.. | |||||||||||||||||||||
| 詳細 | DM-solublized KdpFABC complex purified through SEC the same day as grid freezing. Peak fraction diluted to 4.5 mg/mL and complexed with AlF4 for 1 hr at room temperature prior to application to grid. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 77.0 K / 最高: 77.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 検出モード: COUNTING / デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-50 / 撮影したグリッド数: 1 / 実像数: 2636 / 平均露光時間: 2.7 sec. / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 当てはまり具合の基準: Cross-correlation |
|---|
 ムービー
ムービー コントローラー
コントローラー

































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)