+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22931 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The capsid of Myoviridae Phage XM1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Myoviridae / Vibrio phage / Capsid / HK97 fold / VIRUS | |||||||||
| 生物種 |  Vibrio phage XM1 (ファージ) Vibrio phage XM1 (ファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Wang Z / Klose T | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021タイトル: Structure of Vibrio phage XM1, a simple contractile DNA injection machine 著者: Wang Z / Fokine A / Guo X / Jiang W / Rossmann MG / Kuhn RJ / Luo ZH / Klose T | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22931.map.gz emd_22931.map.gz | 1.5 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22931-v30.xml emd-22931-v30.xml emd-22931.xml emd-22931.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
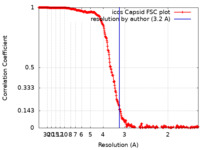
| FSC (解像度算出) |  emd_22931_fsc.xml emd_22931_fsc.xml | 33.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22931.png emd_22931.png | 166.1 KB | ||
| Filedesc metadata |  emd-22931.cif.gz emd-22931.cif.gz | 5.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22931 http://ftp.pdbj.org/pub/emdb/structures/EMD-22931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22931 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22931_validation.pdf.gz emd_22931_validation.pdf.gz | 773.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22931_full_validation.pdf.gz emd_22931_full_validation.pdf.gz | 773 KB | 表示 | |
| XML形式データ |  emd_22931_validation.xml.gz emd_22931_validation.xml.gz | 25.3 KB | 表示 | |
| CIF形式データ |  emd_22931_validation.cif.gz emd_22931_validation.cif.gz | 35.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22931 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22931 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22931 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22931 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22931.map.gz / 形式: CCP4 / 大きさ: 3.3 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22931.map.gz / 形式: CCP4 / 大きさ: 3.3 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.81 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Vibrio phage XM1
| 全体 | 名称:  Vibrio phage XM1 (ファージ) Vibrio phage XM1 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Vibrio phage XM1
| 超分子 | 名称: Vibrio phage XM1 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 2748688 / 生物種: Vibrio phage XM1 / ウイルスタイプ: VIRION / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Vibrio rotiferianus (バクテリア) Vibrio rotiferianus (バクテリア) |
| ウイルス殻 | Shell ID: 1 / 名称: capsid / 直径: 640.0 Å / T番号(三角分割数): 7 |
-分子 #1: Minor capsid protein
| 分子 | 名称: Minor capsid protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vibrio phage XM1 (ファージ) Vibrio phage XM1 (ファージ) |
| 分子量 | 理論値: 16.823863 KDa |
| 配列 | 文字列: MAFNNAVLQE VSDLPAGEVI KASPHNVSAF EVFQNGLIEG RFVKFDAGSI DILDASATPT IAGIAKRKVT GEIGPGVYST SGIEIDQVA EVINFGFATV TVQDAAAPSK YDPVYAINLD SAEAGKATEN SGATGALAVA DCVFWEQKAA NVWLVRMNKF L |
-分子 #2: Major capsid protein
| 分子 | 名称: Major capsid protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vibrio phage XM1 (ファージ) Vibrio phage XM1 (ファージ) |
| 分子量 | 理論値: 35.512773 KDa |
| 配列 | 文字列: MKKDLKFIKS VYDLKSFSDK AAFAKKTFKD EGGIILARNL EHVSSEIFTQ EFAGLTFLQG GIVVNNEGGY ATSVTKLKLK AEGGFRESG NDTNTTGKIT LSGESDSIPV FTLEGESDWS EIELKQAELQ NVNLPSRYFE AHAELYNRKI DELGYLGQTR T DGTQKTLG ...文字列: MKKDLKFIKS VYDLKSFSDK AAFAKKTFKD EGGIILARNL EHVSSEIFTQ EFAGLTFLQG GIVVNNEGGY ATSVTKLKLK AEGGFRESG NDTNTTGKIT LSGESDSIPV FTLEGESDWS EIELKQAELQ NVNLPSRYFE AHAELYNRKI DELGYLGQTR T DGTQKTLG LLNYGFVASG AGDTAANLSG DNLYQAIADL ITDQWAGVFN VETYKADRVV MPDTVYNICA KKILNSNGSE MS VLRALMT NFPTVTFGLT TKARDVGGTS RTTAYSSNRR AMQMRIPTPL NVSSVDQRGF KYYVESYFGV AGLDVIEDTA GRH LTGL |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 50 mM Tris, pH 7.5, 100 mM NaCl, 8 mM MgSO4 |
|---|---|
| グリッド | モデル: PELCO Ultrathin Carbon with Lacey Carbon / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: LACEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 298 K / 装置: GATAN CRYOPLUNGE 3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: DIRECT ELECTRON DE-16 (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7kmx: |
 ムービー
ムービー コントローラー
コントローラー