+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2226 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle analysis of recombinant human anaphase promoting complex (APC/C) | |||||||||
 マップデータ マップデータ | reconstruction of recombinant human APC/C | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Anaphase promoting complex / cyclosome / cell cycle / recombinant expression / single particle / 3-dimensional structure / ubiquitination | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Zhang Z / Yang J / Kong EH / Chao WCH / Morris EP / da Fonseca PCA / Barford D | |||||||||
 引用 引用 |  ジャーナル: Biochem J / 年: 2013 ジャーナル: Biochem J / 年: 2013タイトル: Recombinant expression, reconstitution and structure of human anaphase-promoting complex (APC/C). 著者: Ziguo Zhang / Jing Yang / Eric H Kong / William C H Chao / Edward P Morris / Paula C A da Fonseca / David Barford /  要旨: Mechanistic and structural studies of large multi-subunit assemblies are greatly facilitated by their reconstitution in heterologous recombinant systems. In the present paper, we describe the ...Mechanistic and structural studies of large multi-subunit assemblies are greatly facilitated by their reconstitution in heterologous recombinant systems. In the present paper, we describe the generation of recombinant human APC/C (anaphase-promoting complex/cyclosome), an E3 ubiquitin ligase that regulates cell-cycle progression. Human APC/C is composed of 14 distinct proteins that assemble into a complex of at least 19 subunits with a combined molecular mass of ~1.2 MDa. We show that recombinant human APC/C is correctly assembled, as judged by its capacity to ubiquitinate the budding yeast APC/C substrate Hsl1 (histone synthetic lethal 1) dependent on the APC/C co-activator Cdh1 [Cdc (cell division cycle) 20 homologue 1], and its three-dimensional reconstruction by electron microscopy and single-particle analysis. Successful reconstitution validates the subunit composition of human APC/C. The structure of human APC/C is compatible with the Saccharomyces cerevisiae APC/C homology model, and in contrast with endogenous human APC/C, no evidence for conformational flexibility of the TPR (tetratricopeptide repeat) lobe is observed. Additional density present in the human APC/C structure, proximal to Apc3/Cdc27 of the TPR lobe, is assigned to the TPR subunit Apc7, a subunit specific to vertebrate APC/C. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2226.map.gz emd_2226.map.gz | 10 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2226-v30.xml emd-2226-v30.xml emd-2226.xml emd-2226.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2226.tif emd_2226.tif | 137.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2226 http://ftp.pdbj.org/pub/emdb/structures/EMD-2226 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2226 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2226 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2226_validation.pdf.gz emd_2226_validation.pdf.gz | 197.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2226_full_validation.pdf.gz emd_2226_full_validation.pdf.gz | 196.2 KB | 表示 | |
| XML形式データ |  emd_2226_validation.xml.gz emd_2226_validation.xml.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2226 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2226 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2226 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2226 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2226.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2226.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | reconstruction of recombinant human APC/C | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.4725 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
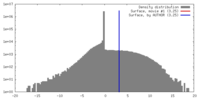
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : recombinant human APC/C
+超分子 #1000: recombinant human APC/C
+分子 #1: Apc1
+分子 #2: Apc5
+分子 #3: Apc15
+分子 #4: Apc10
+分子 #5: Apc8
+分子 #6: Apc13
+分子 #7: Apc2
+分子 #8: Apc4
+分子 #9: Apc11
+分子 #10: Apc3
+分子 #11: Apc16
+分子 #12: Cdc26
+分子 #13: Apc7
+分子 #14: Apc6
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 20 mM HEPES NaOH pH 8.0, 200 mM NaCl, 2 mM DTT |
|---|---|
| 染色 | タイプ: NEGATIVE / 詳細: grids stained with 2% (w/v) uranyl acetate |
| グリッド | 詳細: Quantifoil R1.2/1.3 grids coated with a thin layer of carbon |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 日付 | 2012年1月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F415 (4k x 4k) 平均電子線量: 100 e/Å2 詳細: on recording each adjacent boxes of 2x2 pixels were averaged |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Particles were manually selected using the EMAN boxer software. The images were analysed using a combination of IMAGIC and Spider software. |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Spider, Imagic / 使用した粒子像数: 5639 |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)



















 Trichoplusia ni (イラクサキンウワバ)
Trichoplusia ni (イラクサキンウワバ)