+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21247 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A 2.8 Angstrom Cryo-EM Structure of a Glycoprotein B-Neutralizing Antibody Complex Reveals a Critical Domain for Herpesvirus Fusion Initiation | |||||||||||||||
 マップデータ マップデータ | Varicella zoster virus human neutralizing antibody, 93k, in complex with native gB | |||||||||||||||
 試料 試料 |
| |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / host cell endosome membrane / membrane => GO:0016020 / symbiont entry into host cell / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane 類似検索 - 分子機能 | |||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Human herpesvirus 3 (水痘・帯状疱疹ウイルス) / HHV-3, Varicella zoster virus, Human alphaherpesvirus 3 Human herpesvirus 3 (水痘・帯状疱疹ウイルス) / HHV-3, Varicella zoster virus, Human alphaherpesvirus 3 | |||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||||||||
 データ登録者 データ登録者 | Oliver SL | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2020 ジャーナル: Nat Commun / 年: 2020タイトル: A glycoprotein B-neutralizing antibody structure at 2.8 Å uncovers a critical domain for herpesvirus fusion initiation. 著者: Stefan L Oliver / Yi Xing / Dong-Hua Chen / Soung Hun Roh / Grigore D Pintilie / David A Bushnell / Marvin H Sommer / Edward Yang / Andrea Carfi / Wah Chiu / Ann M Arvin /   要旨: Members of the Herpesviridae, including the medically important alphaherpesvirus varicella-zoster virus (VZV), induce fusion of the virion envelope with cell membranes during entry, and between cells ...Members of the Herpesviridae, including the medically important alphaherpesvirus varicella-zoster virus (VZV), induce fusion of the virion envelope with cell membranes during entry, and between cells to form polykaryocytes in infected tissues. The conserved glycoproteins, gB, gH and gL, are the core functional proteins of the herpesvirus fusion complex. gB serves as the primary fusogen via its fusion loops, but functions for the remaining gB domains remain unexplained. As a pathway for biological discovery of domain function, our approach used structure-based analysis of the viral fusogen together with a neutralizing antibody. We report here a 2.8 Å cryogenic-electron microscopy structure of native gB recovered from VZV-infected cells, in complex with a human monoclonal antibody, 93k. This high-resolution structure guided targeted mutagenesis at the gB-93k interface, providing compelling evidence that a domain spatially distant from the gB fusion loops is critical for herpesvirus fusion, revealing a potential new target for antiviral therapies. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21247.map.gz emd_21247.map.gz | 202.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21247-v30.xml emd-21247-v30.xml emd-21247.xml emd-21247.xml | 20.5 KB 20.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
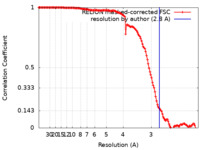
| FSC (解像度算出) |  emd_21247_fsc.xml emd_21247_fsc.xml | 13.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_21247.png emd_21247.png | 170.1 KB | ||
| その他 |  emd_21247_half_map_1.map.gz emd_21247_half_map_1.map.gz emd_21247_half_map_2.map.gz emd_21247_half_map_2.map.gz | 171.1 MB 171.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21247 http://ftp.pdbj.org/pub/emdb/structures/EMD-21247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21247 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21247 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21247_validation.pdf.gz emd_21247_validation.pdf.gz | 649 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21247_full_validation.pdf.gz emd_21247_full_validation.pdf.gz | 648.6 KB | 表示 | |
| XML形式データ |  emd_21247_validation.xml.gz emd_21247_validation.xml.gz | 21 KB | 表示 | |
| CIF形式データ |  emd_21247_validation.cif.gz emd_21247_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21247 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21247 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21247 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21247 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21247.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21247.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Varicella zoster virus human neutralizing antibody, 93k, in complex with native gB | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: half map 1
| ファイル | emd_21247_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 1 | ||||||||||||
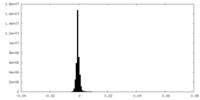
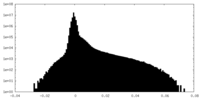
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map 2
| ファイル | emd_21247_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map 2 | ||||||||||||
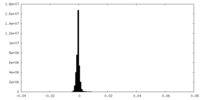
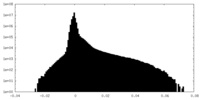
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Varicella zoster virus glycoprotein B in complex with Fab fragmen...
| 全体 | 名称: Varicella zoster virus glycoprotein B in complex with Fab fragments derived from human monoclonal antibody 93k |
|---|---|
| 要素 |
|
-超分子 #1: Varicella zoster virus glycoprotein B in complex with Fab fragmen...
| 超分子 | 名称: Varicella zoster virus glycoprotein B in complex with Fab fragments derived from human monoclonal antibody 93k タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 実験値: 750 KDa |
-超分子 #2: human monoclonal antibody 93k Fab
| 超分子 | 名称: human monoclonal antibody 93k Fab / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Varicella zoster virus glycoprotein B
| 超分子 | 名称: Varicella zoster virus glycoprotein B / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Human herpesvirus 3 (水痘・帯状疱疹ウイルス) Human herpesvirus 3 (水痘・帯状疱疹ウイルス) |
-分子 #1: Human monoclonal antibody 93k variable light chain
| 分子 | 名称: Human monoclonal antibody 93k variable light chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.826245 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPST LSASVGDRVT ITCRASQTIS TWLAWYQQTP RKAPKLMIYK ASILENGVPS RFSGSGSGTE FTLTISSLQP EDFATYYCQ QYKSYPWTFG QGTKVEI |
-分子 #2: Human monoclonal antibody 93k variable heavy chain
| 分子 | 名称: Human monoclonal antibody 93k variable heavy chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 13.658357 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLVQSGAE VKKPGSSVKV SCKASGGTFS NFAISWVRQA PGQGLEWMGR IMPLFVTSTY AQKFQGRVTI SADASTSTAY MELSSLRSD DTAMYYCARD ITAPGAAPTP LNFYGMDVWG QGTTVTVSS |
-分子 #3: Envelope glycoprotein B
| 分子 | 名称: Envelope glycoprotein B / タイプ: protein_or_peptide / ID: 3 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: HHV-3, Varicella zoster virus, Human alphaherpesvirus 3 |
| 分子量 | 理論値: 105.50232 KDa |
| 配列 | 文字列: MSPCGYYSKW RNRDRPEYRR NLRFRRFFSS IHPNAAAGSG FNGPGVFITS VTGVWLCFLC IFSMFVTAVV SVSPSSFYES LQVEPTQSE DITRSAHLGD GDEIREAIHK SQDAETKPTF YVCPPPTGST IVRLEPPRTC PDYHLGKNFT EGIAVVYKEN I AAYKFKAT ...文字列: MSPCGYYSKW RNRDRPEYRR NLRFRRFFSS IHPNAAAGSG FNGPGVFITS VTGVWLCFLC IFSMFVTAVV SVSPSSFYES LQVEPTQSE DITRSAHLGD GDEIREAIHK SQDAETKPTF YVCPPPTGST IVRLEPPRTC PDYHLGKNFT EGIAVVYKEN I AAYKFKAT VYYKDVIVST AWAGSSYTQI TNRYADRVPI PVSEITDTID KFGKCSSKAT YVRNNHKVEA FNEDKNPQDM PL IASKYNS VGSKAWHTTN DTYMVAGTPG TYRTGTSVNC IIEEVEARSI FPYDSFGLST GDIIYMSPFF GLRDGAYREH SNY AMDRFH QFEGYRQRDL DTRALLEPAA RNFLVTPHLT VGWNWKPKRT EVCSLVKWRE VEDVVRDEYA HNFRFTMKTL STTF ISETN EFNLNQIHLS QCVKEEARAI INRIYTTRYN SSHVRTGDIQ TYLARGGFVV VFQPLLSNSL ARLYLQELVR ENTNH SPQK HPTRNTRSRR SVPVELRANR TITTTSSVEF AMLQFTYDHI QEHVNEMLAR ISSSWCQLQN RERALWSGLF PINPSA LAS TILDQRVKAR ILGDVISVSN CPELGSDTRI ILQNSMRVSG STTRCYSRPL ISIVSLNGSG TVEGQLGTDN ELIMSRD LL EPCVANHKRY FLFGHHYVYY EDYRYVREIA VHDVGMISTY VDLNLTLLKD REFMPLRVYT RDELRDTGLL DYSEIQRR N QMHSLRFYDI DKVVQYDSGT AIMQGMAQFF QGLGTAGQAV GHVVLGATGA LLSTVHGFTT FLSNPFGALA VGLLVLAGL VAAFFAYRYV LKLKTSPMKA LYPLTTKGLK QLPEGMDPFA EKPNATDTPI EEIGDSQNTE PSVNSGFDPD KFREAQEMIK YMTLVSAAE RQESKARKKN KTSALLTSRL TGLALRNRRG YSRVRTENVT GV |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| グリッド | 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 98 % / チャンバー内温度: 298 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 4 / 実像数: 11283 / 平均露光時間: 12.0 sec. / 平均電子線量: 7.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z
Z Y
Y X
X