+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-21021 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | MicroED structure of an oxindole product from modified trpB at 0.9A resolution | |||||||||
 マップデータ マップデータ | MicroED 2Fo-Fc map for an oxindole product from modified trpB at 0.9A resolution | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線結晶学 / クライオ電子顕微鏡法 / 解像度: 0.9 Å | |||||||||
 データ登録者 データ登録者 | Dick M / Sarai NS / Martynowycz MW / Gonen T / Arnold FH | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Am Chem Soc / 年: 2019 ジャーナル: J Am Chem Soc / 年: 2019タイトル: Tailoring Tryptophan Synthase TrpB for Selective Quaternary Carbon Bond Formation. 著者: Markus Dick / Nicholas S Sarai / Michael W Martynowycz / Tamir Gonen / Frances H Arnold /  要旨: We previously engineered the β-subunit of tryptophan synthase (TrpB), which catalyzes the condensation of l-serine and indole to l-tryptophan, to synthesize a range of noncanonical amino acids from ...We previously engineered the β-subunit of tryptophan synthase (TrpB), which catalyzes the condensation of l-serine and indole to l-tryptophan, to synthesize a range of noncanonical amino acids from l-serine and indole derivatives or other nucleophiles. Here we employ directed evolution to engineer TrpB to accept 3-substituted oxindoles and form C-C bonds leading to new quaternary stereocenters. Initially, the variants that could use 3-substituted oxindoles preferentially formed N-C bonds on N of the substrate. Protecting N encouraged evolution toward C-alkylation, which persisted when protection was removed. Six generations of directed evolution resulted in TrpB with a 400-fold improvement in activity for alkylation of 3-substituted oxindoles and the ability to selectively form a new, all-carbon quaternary stereocenter at the γ-position of the amino acid products. The enzyme can also alkylate and form all-carbon quaternary stereocenters on structurally similar lactones and ketones, where it exhibits excellent regioselectivity for the tertiary carbon. The configurations of the γ-stereocenters of two of the products were determined via microcrystal electron diffraction (MicroED), and we report the MicroED structure of a small molecule obtained using the Falcon III direct electron detector. Highly thermostable and expressed at >500 mg/L culture, TrpB offers an efficient, sustainable, and selective platform for the construction of diverse noncanonical amino acids bearing all-carbon quaternary stereocenters. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_21021.map.gz emd_21021.map.gz | 558 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-21021-v30.xml emd-21021-v30.xml emd-21021.xml emd-21021.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_21021.png emd_21021.png | 43.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-21021 http://ftp.pdbj.org/pub/emdb/structures/EMD-21021 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21021 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-21021 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_21021_validation.pdf.gz emd_21021_validation.pdf.gz | 79.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_21021_full_validation.pdf.gz emd_21021_full_validation.pdf.gz | 78.8 KB | 表示 | |
| XML形式データ |  emd_21021_validation.xml.gz emd_21021_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21021 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21021 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21021 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-21021 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_21021.map.gz / 形式: CCP4 / 大きさ: 4.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_21021.map.gz / 形式: CCP4 / 大きさ: 4.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | MicroED 2Fo-Fc map for an oxindole product from modified trpB at 0.9A resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 0.28929 Å / Y: 0.28063 Å / Z: 0.28556 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
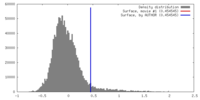
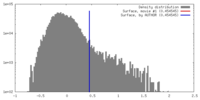
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : (S)-2-amino-3-((S)-3-methyl-2-oxoindolin-3-yl)propanoic acid
| 全体 | 名称: (S)-2-amino-3-((S)-3-methyl-2-oxoindolin-3-yl)propanoic acid |
|---|---|
| 要素 |
|
-超分子 #1: (S)-2-amino-3-((S)-3-methyl-2-oxoindolin-3-yl)propanoic acid
| 超分子 | 名称: (S)-2-amino-3-((S)-3-methyl-2-oxoindolin-3-yl)propanoic acid タイプ: complex / ID: 1 / 親要素: 0 / 詳細: Oxindole product from TrpB |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 215 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線結晶学 |
| 試料の集合状態 | 3D array |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI CETA (4k x 4k) / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 28.0 µm / 撮影したグリッド数: 2 / 実像数: 120 / 回折像の数: 120 / 平均露光時間: 1.0 sec. / 平均電子線量: 0.01 e/Å2 詳細: Frames were recorded every 0.5 degrees for ~60 degrees with a 1 second integration time. |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / カメラ長: 1053 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN / 傾斜角度: 0, 60 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 0.9 Å / 解像度の算出法: DIFFRACTION PATTERN/LAYERLINES |
|---|---|
| 結晶パラメータ | 単位格子 - A: 12.15 Å / 単位格子 - B: 8.98 Å / 単位格子 - C: 10.28 Å / 単位格子 - γ: 90 ° / 単位格子 - α: 90 ° / 単位格子 - β: 99.41 ° / 空間群: 14 |
| Crystallography statistics | Number intensities measured: 2524 / Number structure factors: 1028 / Fourier space coverage: 65.1 / R sym: 12.8 / R merge: 10.1 / Overall phase error: 20 / Overall phase residual: 20 / Phase error rejection criteria: 0 / High resolution: 0.9 Å / 殻 - Shell ID: 1 / 殻 - High resolution: 10.0 Å / 殻 - Low resolution: 0.9 Å / 殻 - Number structure factors: 1024 / 殻 - Phase residual: 20 / 殻 - Fourier space coverage: 65.1 / 殻 - Multiplicity: 2.4 |
-原子モデル構築 1
| 精密化 | 空間: RECIPROCAL / プロトコル: AB INITIO MODEL / 温度因子: 6 / 当てはまり具合の基準: Least Squares |
|---|
 ムービー
ムービー コントローラー
コントローラー





 Z (Sec.)
Z (Sec.) X (Row.)
X (Row.) Y (Col.)
Y (Col.)