+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20729 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the mitochondrial TOM complex from yeast (tetramer) | |||||||||
 マップデータ マップデータ | Filtered, sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Membrane protein / mitochondrial protein import / mitochondrial outer membrane / protein translocation / TRANSLOCASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial outer membrane translocase complex assembly / mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / protein targeting to mitochondrion / protein insertion into mitochondrial outer membrane / porin activity / pore complex / protein transmembrane transporter activity / monoatomic ion transport / mitochondrial intermembrane space ...mitochondrial outer membrane translocase complex assembly / mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / protein targeting to mitochondrion / protein insertion into mitochondrial outer membrane / porin activity / pore complex / protein transmembrane transporter activity / monoatomic ion transport / mitochondrial intermembrane space / mitochondrial outer membrane / mitochondrion / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
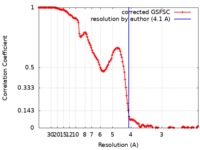
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Park E / Tucker K | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2019 ジャーナル: Nat Struct Mol Biol / 年: 2019タイトル: Cryo-EM structure of the mitochondrial protein-import channel TOM complex at near-atomic resolution. 著者: Kyle Tucker / Eunyong Park /  要旨: Nearly all mitochondrial proteins are encoded by the nuclear genome and imported into mitochondria after synthesis on cytosolic ribosomes. These precursor proteins are translocated into mitochondria ...Nearly all mitochondrial proteins are encoded by the nuclear genome and imported into mitochondria after synthesis on cytosolic ribosomes. These precursor proteins are translocated into mitochondria by the TOM complex, a protein-conducting channel in the mitochondrial outer membrane. We have determined high-resolution cryo-EM structures of the core TOM complex from Saccharomyces cerevisiae in dimeric and tetrameric forms. Dimeric TOM consists of two copies each of five proteins arranged in two-fold symmetry: pore-forming β-barrel protein Tom40 and four auxiliary α-helical transmembrane proteins. The pore of each Tom40 has an overall negatively charged inner surface attributed to multiple functionally important acidic patches. The tetrameric complex is essentially a dimer of dimeric TOM, which may be capable of forming higher-order oligomers. Our study reveals the detailed molecular organization of the TOM complex and provides new insights about the mechanism of protein translocation into mitochondria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20729.map.gz emd_20729.map.gz | 229.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20729-v30.xml emd-20729-v30.xml emd-20729.xml emd-20729.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_20729_fsc.xml emd_20729_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_20729.png emd_20729.png | 181.6 KB | ||
| Filedesc metadata |  emd-20729.cif.gz emd-20729.cif.gz | 6 KB | ||
| その他 |  emd_20729_additional.map.gz emd_20729_additional.map.gz | 226.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20729 http://ftp.pdbj.org/pub/emdb/structures/EMD-20729 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20729 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20729 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_20729_validation.pdf.gz emd_20729_validation.pdf.gz | 515.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_20729_full_validation.pdf.gz emd_20729_full_validation.pdf.gz | 515 KB | 表示 | |
| XML形式データ |  emd_20729_validation.xml.gz emd_20729_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_20729_validation.cif.gz emd_20729_validation.cif.gz | 18.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20729 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20729 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20729 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-20729 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20729.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20729.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Filtered, sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Raw combined map
| ファイル | emd_20729_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw combined map | ||||||||||||
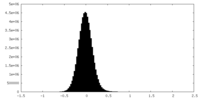
| 投影像・断面図 |
| ||||||||||||
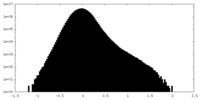
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Tetrameric TOM complex from yeast
| 全体 | 名称: Tetrameric TOM complex from yeast |
|---|---|
| 要素 |
|
-超分子 #1: Tetrameric TOM complex from yeast
| 超分子 | 名称: Tetrameric TOM complex from yeast / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
-分子 #1: Mitochondrial import receptor subunit TOM40
| 分子 | 名称: Mitochondrial import receptor subunit TOM40 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 43.227387 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAPTPLAEA SQIPTIPALS PLTAKQSKGN FFSSNPISSF VVDTYKQLHS HRQSLELVNP GTVENLNKEV SRDVFLSQYF FTGLRADLN KAFSMNPAFQ TSHTFSIGSQ ALPKYAFSAL FANDNLFAQG NIDNDLSVSG RLNYGWDKKN ISKVNLQISD G QPTMCQLE ...文字列: MSAPTPLAEA SQIPTIPALS PLTAKQSKGN FFSSNPISSF VVDTYKQLHS HRQSLELVNP GTVENLNKEV SRDVFLSQYF FTGLRADLN KAFSMNPAFQ TSHTFSIGSQ ALPKYAFSAL FANDNLFAQG NIDNDLSVSG RLNYGWDKKN ISKVNLQISD G QPTMCQLE QDYQASDFSV NVKTLNPSFS EKGEFTGVAV ASFLQSVTPQ LALGLETLYS RTDGSAPGDA GVSYLTRYVS KK QDWIFSG QLQANGALIA SLWRKVAQNV EAGIETTLQA GMVPITDPLM GTPIGIQPTV EGSTTIGAKY EYRQSVYRGT LDS NGKVAC FLERKVLPTL SVLFCGEIDH FKNDTKIGCG LQFETAGNQE LLMLQQGLDA DGNPLQALPQ LGGWSHPQFE K UniProtKB: Mitochondrial import receptor subunit TOM40 |
-分子 #2: Mitochondrial import receptor subunit TOM22
| 分子 | 名称: Mitochondrial import receptor subunit TOM22 / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 18.02065 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MVELTEIKDD VVQLDEPQFS RNQAIVEEKA SATNNDVVDD EDDSDSDFED EFDENETLLD RIVALKDIVP PGKRQTISNF FGFTSSFVR NAFTKSGNLA WTLTTTALLL GVPLSLSILA EQQLIEMEKT FDLQSDANNI LAQGEKDAAA TANGGHHHHH H HH UniProtKB: Mitochondrial import receptor subunit TOM22 |
-分子 #3: Mitochondrial import receptor subunit TOM5
| 分子 | 名称: Mitochondrial import receptor subunit TOM5 / タイプ: protein_or_peptide / ID: 3 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 5.993924 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MFGLPQQEVS EEEKRAHQEQ TEKTLKQAAY VAAFLWVSPM IWHLVKKQWK UniProtKB: Mitochondrial import receptor subunit TOM5 |
-分子 #4: Mitochondrial import receptor subunit TOM6
| 分子 | 名称: Mitochondrial import receptor subunit TOM6 / タイプ: protein_or_peptide / ID: 4 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 6.41046 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MDGMFAMPGA AAGAASPQQP KSRFQAFKES PLYTIALNGA FFVAGVAFIQ SPLMDMLAPQ L UniProtKB: Mitochondrial import receptor subunit TOM6 |
-分子 #5: Mitochondrial import receptor subunit TOM7
| 分子 | 名称: Mitochondrial import receptor subunit TOM7 / タイプ: protein_or_peptide / ID: 5 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  株: ATCC 204508 / S288c |
| 分子量 | 理論値: 6.876955 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSFLPSFILS DESKERISKI LTLTHNVAHY GWIPFVLYLG WAHTSNRPNF LNLLSPLPSV UniProtKB: Mitochondrial import receptor subunit TOM7 |
-分子 #6: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE
| 分子 | 名称: 1,2-DIMYRISTOYL-RAC-GLYCERO-3-PHOSPHOCHOLINE / タイプ: ligand / ID: 6 / コピー数: 2 / 式: MC3 |
|---|---|
| 分子量 | 理論値: 677.933 Da |
| Chemical component information |  ChemComp-MC3: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 43.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X