+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1993 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain reconstruction of recombinantly expressed yeast proteasome lid | |||||||||
 マップデータ マップデータ | Map of recombinantly expressed Saccharomyces cerevisiae proteasome lid | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | 26S / 19S / proteasome / yeast lid / regulatory particle / ubiquitin recognition / deubiquitination / AAA-ATPase | |||||||||
| 機能・相同性 | Proteasome/cyclosome repeat / proteasome regulatory particle, lid subcomplex 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Lander GC / Estrin E / Matyskiela M / Bashore C / Nogales E / Martin A | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2012 ジャーナル: Nature / 年: 2012タイトル: Complete subunit architecture of the proteasome regulatory particle. 著者: Gabriel C Lander / Eric Estrin / Mary E Matyskiela / Charlene Bashore / Eva Nogales / Andreas Martin /  要旨: The proteasome is the major ATP-dependent protease in eukaryotic cells, but limited structural information restricts a mechanistic understanding of its activities. The proteasome regulatory particle, ...The proteasome is the major ATP-dependent protease in eukaryotic cells, but limited structural information restricts a mechanistic understanding of its activities. The proteasome regulatory particle, consisting of the lid and base subcomplexes, recognizes and processes polyubiquitinated substrates. Here we used electron microscopy and a new heterologous expression system for the lid to delineate the complete subunit architecture of the regulatory particle from yeast. Our studies reveal the spatial arrangement of ubiquitin receptors, deubiquitinating enzymes and the protein unfolding machinery at subnanometre resolution, outlining the substrate's path to degradation. Unexpectedly, the ATPase subunits within the base unfoldase are arranged in a spiral staircase, providing insight into potential mechanisms for substrate translocation through the central pore. Large conformational rearrangements of the lid upon holoenzyme formation suggest allosteric regulation of deubiquitination. We provide a structural basis for the ability of the proteasome to degrade a diverse set of substrates and thus regulate vital cellular processes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1993.map.gz emd_1993.map.gz | 398.1 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1993-v30.xml emd-1993-v30.xml emd-1993.xml emd-1993.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  em1993.png em1993.png | 237.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1993 http://ftp.pdbj.org/pub/emdb/structures/EMD-1993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1993 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1993 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_1993_validation.pdf.gz emd_1993_validation.pdf.gz | 187 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_1993_full_validation.pdf.gz emd_1993_full_validation.pdf.gz | 186.2 KB | 表示 | |
| XML形式データ |  emd_1993_validation.xml.gz emd_1993_validation.xml.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1993 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1993 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1993 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-1993 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1993.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1993.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of recombinantly expressed Saccharomyces cerevisiae proteasome lid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.76 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
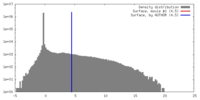
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : recombinantly expressed yeast lid
| 全体 | 名称: recombinantly expressed yeast lid |
|---|---|
| 要素 |
|
-超分子 #1000: recombinantly expressed yeast lid
| 超分子 | 名称: recombinantly expressed yeast lid / タイプ: sample / ID: 1000 / 詳細: monodisperse / 集合状態: 8 subunit complex / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 361 KDa / 理論値: 361 KDa / 手法: Mass Spectrometry |
-分子 #1: lid
| 分子 | 名称: lid / タイプ: protein_or_peptide / ID: 1 / Name.synonym: lid / 詳細: all subunits were recombinantly expressed / コピー数: 1 / 集合状態: monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 361 KDa / 理論値: 361 KDa |
| 組換発現 | 生物種: Escherichia coli BL21-star (DE3) / 組換プラスミド: pAM001,pAM002,pAM003 |
| 配列 | GO: proteasome regulatory particle, lid subcomplex / InterPro: Proteasome/cyclosome repeat |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.009 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 25mM HEPES, 100mM NaCl, 100mM KCl, 5% glycerol |
| 染色 | タイプ: NEGATIVE 詳細: Protein adsorbed to grid for 1 minute, then passed over four 50uL drops of 2% w/v uranyl formate, 5 seconds on each drop |
| グリッド | 詳細: 200 mesh Cu grid |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 温度 | 最低: 78 K / 最高: 78 K / 平均: 78 K |
| アライメント法 | Legacy - 非点収差: objective lens astigmatism was corrected at 210,000 times magnification Legacy - Electron beam tilt params: 0 |
| 詳細 | Data acquired using Leginon |
| 日付 | 2011年8月22日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GENERIC GATAN (4k x 4k) 実像数: 303 / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 80000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダー: Room temp single tilt / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 画像解析
画像解析
| 詳細 | Image processing performed in the Appion processing environment. 3D reconstruction performed using EMAN2 and SPARX libraries |
|---|---|
| CTF補正 | 詳細: whole micrograph |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN2 SPARX / 使用した粒子像数: 29345 |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)