+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RUVBL1/2 in complex with ATP | |||||||||
 マップデータ マップデータ | CryoEM map (Refine3D) for RUVBL1/2-ATP complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RUVBL1 / RUVBL2 / CB-6644 / Inhibitor / Chaperone | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報promoter-enhancer loop anchoring activity / regulation of DNA strand elongation / positive regulation of telomere maintenance in response to DNA damage / establishment of protein localization to chromatin / R2TP complex / dynein axonemal particle / RPAP3/R2TP/prefoldin-like complex / Swr1 complex / positive regulation of telomerase RNA localization to Cajal body / regulation of double-strand break repair ...promoter-enhancer loop anchoring activity / regulation of DNA strand elongation / positive regulation of telomere maintenance in response to DNA damage / establishment of protein localization to chromatin / R2TP complex / dynein axonemal particle / RPAP3/R2TP/prefoldin-like complex / Swr1 complex / positive regulation of telomerase RNA localization to Cajal body / regulation of double-strand break repair / Ino80 complex / protein folding chaperone complex / box C/D snoRNP assembly / regulation of chromosome organization / NuA4 histone acetyltransferase complex / regulation of DNA replication / TFIID-class transcription factor complex binding / regulation of embryonic development / MLL1 complex / Telomere Extension By Telomerase / positive regulation of double-strand break repair via homologous recombination / RNA polymerase II core promoter sequence-specific DNA binding / regulation of DNA repair / DNA helicase activity / Deposition of new CENPA-containing nucleosomes at the centromere / TBP-class protein binding / positive regulation of DNA repair / telomere maintenance / cellular response to estradiol stimulus / Formation of the beta-catenin:TCF transactivating complex / DNA Damage Recognition in GG-NER / negative regulation of canonical Wnt signaling pathway / euchromatin / chromatin DNA binding / ADP binding / beta-catenin binding / nuclear matrix / transcription corepressor activity / UCH proteinases / cellular response to UV / nucleosome / unfolded protein binding / positive regulation of canonical Wnt signaling pathway / protein folding / HATs acetylate histones / ATPase binding / spermatogenesis / regulation of apoptotic process / DNA recombination / DNA helicase / transcription coactivator activity / protein stabilization / regulation of cell cycle / Ub-specific processing proteases / ribonucleoprotein complex / chromatin remodeling / cadherin binding / RNA polymerase II cis-regulatory region sequence-specific DNA binding / cell division / DNA repair / centrosome / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / protein homodimerization activity / positive regulation of transcription by RNA polymerase II / ATP hydrolysis activity / extracellular exosome / nucleoplasm / ATP binding / identical protein binding / membrane / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
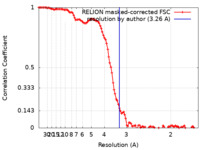
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.26 Å | |||||||||
 データ登録者 データ登録者 | Lopez-Perrote A / Llorca O / Garcia-Martin C | |||||||||
| 資金援助 |  スペイン, 1件 スペイン, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Rep Phys Sci / 年: 2024 ジャーナル: Cell Rep Phys Sci / 年: 2024タイトル: Mechanism of allosteric inhibition of RUVBL1-RUVBL2 by the small-molecule CB-6644 著者: Garcia-Martin C / Lopez-Perrote A / Boskovic J / Llorca O | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19817.map.gz emd_19817.map.gz | 79.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19817-v30.xml emd-19817-v30.xml emd-19817.xml emd-19817.xml | 22.6 KB 22.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19817_fsc.xml emd_19817_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19817.png emd_19817.png | 585.7 KB | ||
| Filedesc metadata |  emd-19817.cif.gz emd-19817.cif.gz | 7.1 KB | ||
| その他 |  emd_19817_additional_1.map.gz emd_19817_additional_1.map.gz emd_19817_half_map_1.map.gz emd_19817_half_map_1.map.gz emd_19817_half_map_2.map.gz emd_19817_half_map_2.map.gz | 10 MB 78.9 MB 78.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19817 http://ftp.pdbj.org/pub/emdb/structures/EMD-19817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19817 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19817 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9emcMC  9emaC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19817.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19817.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | CryoEM map (Refine3D) for RUVBL1/2-ATP complex | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8272 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Sharpened map (Postprocess) for RUVBL1/2-ATP complex
| ファイル | emd_19817_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map (Postprocess) for RUVBL1/2-ATP complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 (unfiltered) for RUVBL1/2-ATP complex
| ファイル | emd_19817_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 (unfiltered) for RUVBL1/2-ATP complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 (unfiltered) for RUVBL1/2-ATP complex
| ファイル | emd_19817_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 (unfiltered) for RUVBL1/2-ATP complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Hetero-hexameric RUVBL1-RUVBL2 complex bound to ATP and the CB-66...
| 全体 | 名称: Hetero-hexameric RUVBL1-RUVBL2 complex bound to ATP and the CB-6644 inhibitor |
|---|---|
| 要素 |
|
-超分子 #1: Hetero-hexameric RUVBL1-RUVBL2 complex bound to ATP and the CB-66...
| 超分子 | 名称: Hetero-hexameric RUVBL1-RUVBL2 complex bound to ATP and the CB-6644 inhibitor タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 310.47 kDa/nm |
-分子 #1: RuvB-like 1
| 分子 | 名称: RuvB-like 1 / タイプ: protein_or_peptide / ID: 1 詳細: Sequence contains three extra aminoacids (GSH) at the N-terminus due to protease cleavage that do not affect the structure and activity of the protein コピー数: 3 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 50.579188 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMKIEEVK STTKTQRIAS HSHVKGLGLD ESGLAKQAAS GLVGQENARE ACGVIVELIK SKKMAGRAVL LAGPPGTGKT ALALAIAQE LGSKVPFCPM VGSEVYSTEI KKTEVLMENF RRAIGLRIKE TKEVYEGEVT ELTPCETENP MGGYGKTISH V IIGLKTAK ...文字列: GSHMKIEEVK STTKTQRIAS HSHVKGLGLD ESGLAKQAAS GLVGQENARE ACGVIVELIK SKKMAGRAVL LAGPPGTGKT ALALAIAQE LGSKVPFCPM VGSEVYSTEI KKTEVLMENF RRAIGLRIKE TKEVYEGEVT ELTPCETENP MGGYGKTISH V IIGLKTAK GTKQLKLDPS IFESLQKERV EAGDVIYIEA NSGAVKRQGR CDTYATEFDL EAEEYVPLPK GDVHKKKEII QD VTLHDLD VANARPQGGQ DILSMMGQLM KPKKTEITDK LRGEINKVVN KYIDQGIAEL VPGVLFVDEV HMLDIECFTY LHR ALESSI APIVIFASNR GNCVIRGTED ITSPHGIPLD LLDRVMIIRT MLYTPQEMKQ IIKIRAQTEG INISEEALNH LGEI GTKTT LRYSVQLLTP ANLLAKINGK DSIEKEHVEE ISELFYDAKS SAKILADQQD KYMK UniProtKB: RuvB-like 1 |
-分子 #2: RuvB-like 2
| 分子 | 名称: RuvB-like 2 / タイプ: protein_or_peptide / ID: 2 詳細: A strectch of 18 extra residues is present at the N-terminus due to cloning design that do not affect the structure and activity of the protein コピー数: 3 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 53.047488 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MADLNWISAG HAIADVGTMA TVTATTKVPE IRDVTRIERI GAHSHIRGLG LDDALEPRQA SQGMVGQLAA RRAAGVVLEM IREGKIAGR AVLIAGQPGT GKTAIAMGMA QALGPDTPFT AIAGSEIFSL EMSKTEALTQ AFRRSIGVRI KEETEIIEGE V VEIQIDRP ...文字列: MADLNWISAG HAIADVGTMA TVTATTKVPE IRDVTRIERI GAHSHIRGLG LDDALEPRQA SQGMVGQLAA RRAAGVVLEM IREGKIAGR AVLIAGQPGT GKTAIAMGMA QALGPDTPFT AIAGSEIFSL EMSKTEALTQ AFRRSIGVRI KEETEIIEGE V VEIQIDRP ATGTGSKVGK LTLKTTEMET IYDLGTKMIE SLTKDKVQAG DVITIDKATG KISKLGRSFT RARDYDAMGS QT KFVQCPD GELQKRKEVV HTVSLHEIDV INSRTQGFLA LFSGDTGEIK SEVREQINAK VAEWREEGKA EIIPGVLFID EVH MLDIES FSFLNRALES DMAPVLIMAT NRGITRIRGT SYQSPHGIPI DLLDRLLIVS TTPYSEKDTK QILRIRCEEE DVEM SEDAY TVLTRIGLET SLRYAIQLIT AASLVCRKRK GTEVQVDDIK RVYSLFLDES RSTQYMKEYQ DAFLFNELKG ETMDT S UniProtKB: RuvB-like 2 |
-分子 #3: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 6 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R0.6/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 10.0 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOCONTINUUM (6k x 4k) 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: in silico model / 詳細: ModelAngelo |
|---|---|
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE |
| 得られたモデル |  PDB-9emc: |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X