+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | human PLD3 homodimer structure | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Exonuclease / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報spleen exonuclease / Synthesis of PG / single-stranded DNA 5'-3' DNA exonuclease activity / myotube differentiation / phospholipase D activity / immune system process / regulation of cytokine production involved in inflammatory response / Role of phospholipids in phagocytosis / lysosomal lumen / late endosome membrane ...spleen exonuclease / Synthesis of PG / single-stranded DNA 5'-3' DNA exonuclease activity / myotube differentiation / phospholipase D activity / immune system process / regulation of cytokine production involved in inflammatory response / Role of phospholipids in phagocytosis / lysosomal lumen / late endosome membrane / early endosome membrane / inflammatory response / lysosomal membrane / Golgi membrane / endoplasmic reticulum membrane / extracellular exosome 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
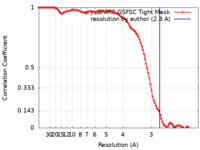
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Lammens K | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Immunity / 年: 2024 ジャーナル: Immunity / 年: 2024タイトル: Lysosomal endonuclease RNase T2 and PLD exonucleases cooperatively generate RNA ligands for TLR7 activation. 著者: Marleen Bérouti / Katja Lammens / Matthias Heiss / Larissa Hansbauer / Stefan Bauernfried / Jan Stöckl / Francesca Pinci / Ignazio Piseddu / Wilhelm Greulich / Meiyue Wang / Christophe Jung ...著者: Marleen Bérouti / Katja Lammens / Matthias Heiss / Larissa Hansbauer / Stefan Bauernfried / Jan Stöckl / Francesca Pinci / Ignazio Piseddu / Wilhelm Greulich / Meiyue Wang / Christophe Jung / Thomas Fröhlich / Thomas Carell / Karl-Peter Hopfner / Veit Hornung /  要旨: Toll-like receptor 7 (TLR7) is essential for recognition of RNA viruses and initiation of antiviral immunity. TLR7 contains two ligand-binding pockets that recognize different RNA degradation ...Toll-like receptor 7 (TLR7) is essential for recognition of RNA viruses and initiation of antiviral immunity. TLR7 contains two ligand-binding pockets that recognize different RNA degradation products: pocket 1 recognizes guanosine, while pocket 2 coordinates pyrimidine-rich RNA fragments. We found that the endonuclease RNase T2, along with 5' exonucleases PLD3 and PLD4, collaboratively generate the ligands for TLR7. Specifically, RNase T2 generated guanosine 2',3'-cyclic monophosphate-terminated RNA fragments. PLD exonuclease activity further released the terminal 2',3'-cyclic guanosine monophosphate (2',3'-cGMP) to engage pocket 1 and was also needed to generate RNA fragments for pocket 2. Loss-of-function studies in cell lines and primary cells confirmed the critical requirement for PLD activity. Biochemical and structural studies showed that PLD enzymes form homodimers with two ligand-binding sites important for activity. Previously identified disease-associated PLD mutants failed to form stable dimers. Together, our data provide a mechanistic basis for the detection of RNA fragments by TLR7. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19798.map.gz emd_19798.map.gz | 97 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19798-v30.xml emd-19798-v30.xml emd-19798.xml emd-19798.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_19798_fsc.xml emd_19798_fsc.xml | 9.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_19798.png emd_19798.png | 87.7 KB | ||
| Filedesc metadata |  emd-19798.cif.gz emd-19798.cif.gz | 5.4 KB | ||
| その他 |  emd_19798_half_map_1.map.gz emd_19798_half_map_1.map.gz emd_19798_half_map_2.map.gz emd_19798_half_map_2.map.gz | 95.3 MB 95.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19798 http://ftp.pdbj.org/pub/emdb/structures/EMD-19798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19798 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19798 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8s86MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19798.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19798.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.045 Å | ||||||||||||||||||||
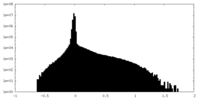
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map B
| ファイル | emd_19798_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
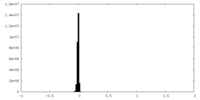
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_19798_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
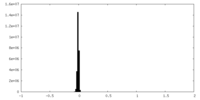
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PLD3 dimer
| 全体 | 名称: PLD3 dimer |
|---|---|
| 要素 |
|
-超分子 #1: PLD3 dimer
| 超分子 | 名称: PLD3 dimer / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: 5'-3' exonuclease PLD3
| 分子 | 名称: 5'-3' exonuclease PLD3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: spleen exonuclease |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 48.058016 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: WEYGDLHLFG PNQRPAPCYD PCEAVLVESI PEGLDFPNAS TGNPSTSQAW LGLLAGAHSS LDIASFYWTL TNNDTHTQEP SAQQGEEVL RQLQTLAPKG VNVRIAVSKP SGPQPQADLQ ALLQSGAQVR MVDMQKLTHG VLHTKFWVVD QTHFYLGSAN M DWRSLTQV ...文字列: WEYGDLHLFG PNQRPAPCYD PCEAVLVESI PEGLDFPNAS TGNPSTSQAW LGLLAGAHSS LDIASFYWTL TNNDTHTQEP SAQQGEEVL RQLQTLAPKG VNVRIAVSKP SGPQPQADLQ ALLQSGAQVR MVDMQKLTHG VLHTKFWVVD QTHFYLGSAN M DWRSLTQV KELGVVMYNC SCLARDLTKI FEAYWFLGQA GSSIPSTWPR FYDTRYNQET PMEICLNGTP ALAYLASAPP PL CPSGRTP DLKALLNVVD NARSFIYVAV MNYLPTLEFS HPHRFWPAID DGLRRATYER GVKVRLLISC WGHSEPSMRA FLL SLAALR DNHTHSDIQV KLFVVPADEA QARIPYARVN HNKYMVTERA TYIGTSNWSG NYFTETAGTS LLVTQNGRGG LRSQ LEAIF LRDWDSPYSH DLDTSADSVG NACRLL UniProtKB: 5'-3' exonuclease PLD3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 5.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 45.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.9 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X