+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the human DDB1-DDA1-DCAF15 E3 ubiquitin ligase bound to compound furan 24 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | E3 ligase / Complex / LIGASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of natural killer cell activation / positive regulation by virus of viral protein levels in host cell / epigenetic programming in the zygotic pronuclei / spindle assembly involved in female meiosis / Cul4-RING E3 ubiquitin ligase complex / UV-damage excision repair / biological process involved in interaction with symbiont / regulation of mitotic cell cycle phase transition / WD40-repeat domain binding / Cul4A-RING E3 ubiquitin ligase complex ...regulation of natural killer cell activation / positive regulation by virus of viral protein levels in host cell / epigenetic programming in the zygotic pronuclei / spindle assembly involved in female meiosis / Cul4-RING E3 ubiquitin ligase complex / UV-damage excision repair / biological process involved in interaction with symbiont / regulation of mitotic cell cycle phase transition / WD40-repeat domain binding / Cul4A-RING E3 ubiquitin ligase complex / Cul4B-RING E3 ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / immune system process / negative regulation of reproductive process / negative regulation of developmental process / small molecule binding / viral release from host cell / cullin family protein binding / ectopic germ cell programmed cell death / proteasomal protein catabolic process / positive regulation of viral genome replication / positive regulation of gluconeogenesis / nucleotide-excision repair / Recognition of DNA damage by PCNA-containing replication complex / DNA Damage Recognition in GG-NER / regulation of circadian rhythm / Transcription-Coupled Nucleotide Excision Repair (TC-NER) / Formation of TC-NER Pre-Incision Complex / Dual Incision in GG-NER / Wnt signaling pathway / Formation of Incision Complex in GG-NER / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / protein polyubiquitination / positive regulation of protein catabolic process / cellular response to UV / rhythmic process / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / Neddylation / site of double-strand break / protein-macromolecule adaptor activity / ubiquitin-dependent protein catabolic process / proteasome-mediated ubiquitin-dependent protein catabolic process / damaged DNA binding / chromosome, telomeric region / protein ubiquitination / DNA repair / DNA damage response / protein-containing complex binding / negative regulation of apoptotic process / nucleolus / apoptotic process / protein-containing complex / DNA binding / extracellular space / extracellular exosome / nucleoplasm / nucleus / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Shilliday F / Lucas SCC / Richter M / Michaelides IN / Fusani L | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: J Med Chem / 年: 2024 ジャーナル: J Med Chem / 年: 2024タイトル: Optimization of Potent Ligands for the E3 Ligase DCAF15 and Evaluation of Their Use in Heterobifunctional Degraders. 著者: Simon C C Lucas / Afshan Ahmed / S Neha Ashraf / Argyrides Argyrou / Matthias R Bauer / Gian Marco De Donatis / Sylvain Demanze / Frederik Eisele / Lucia Fusani / Andreas Hock / Ganesh ...著者: Simon C C Lucas / Afshan Ahmed / S Neha Ashraf / Argyrides Argyrou / Matthias R Bauer / Gian Marco De Donatis / Sylvain Demanze / Frederik Eisele / Lucia Fusani / Andreas Hock / Ganesh Kadamur / Shuyou Li / Abigail Macmillan-Jones / Iacovos N Michaelides / Christopher Phillips / Marie Rehnström / Magdalena Richter / Monica C Rodrigo-Brenni / Fiona Shilliday / Peng Wang / R Ian Storer /    要旨: Unlocking novel E3 ligases for use in heterobifunctional PROTAC degraders is of high importance to the pharmaceutical industry. Over-reliance on the current suite of ligands used to recruit E3 ...Unlocking novel E3 ligases for use in heterobifunctional PROTAC degraders is of high importance to the pharmaceutical industry. Over-reliance on the current suite of ligands used to recruit E3 ligases could limit the potential of their application. To address this, potent ligands for DCAF15 were optimized using cryo-EM supported, structure-based design to improve on micromolar starting points. A potent binder, compound , was identified and subsequently conjugated into PROTACs against multiple targets. Following attempts on degrading a number of proteins using DCAF15 recruiting PROTACs, only degradation of BRD4 was observed. Deconvolution of the mechanism of action showed that this degradation was not mediated by DCAF15, thereby highlighting both the challenges faced when trying to expand the toolbox of validated E3 ligase ligands for use in PROTAC degraders and the pitfalls of using BRD4 as a model substrate. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_19407.map.gz emd_19407.map.gz | 51.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-19407-v30.xml emd-19407-v30.xml emd-19407.xml emd-19407.xml | 17.9 KB 17.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_19407.png emd_19407.png | 41.8 KB | ||
| Filedesc metadata |  emd-19407.cif.gz emd-19407.cif.gz | 6.6 KB | ||
| その他 |  emd_19407_half_map_1.map.gz emd_19407_half_map_1.map.gz emd_19407_half_map_2.map.gz emd_19407_half_map_2.map.gz | 95.5 MB 95.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-19407 http://ftp.pdbj.org/pub/emdb/structures/EMD-19407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19407 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-19407 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_19407_validation.pdf.gz emd_19407_validation.pdf.gz | 1.2 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_19407_full_validation.pdf.gz emd_19407_full_validation.pdf.gz | 1.2 MB | 表示 | |
| XML形式データ |  emd_19407_validation.xml.gz emd_19407_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_19407_validation.cif.gz emd_19407_validation.cif.gz | 15.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19407 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19407 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19407 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-19407 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8royMC  8roxC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_19407.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_19407.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.645 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_19407_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
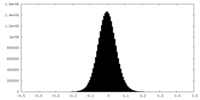
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_19407_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
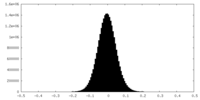
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of the human DDB1-DDA1-DCAF15 E3 ubiquitin ligase bound t...
| 全体 | 名称: Complex of the human DDB1-DDA1-DCAF15 E3 ubiquitin ligase bound to compound furan 24 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of the human DDB1-DDA1-DCAF15 E3 ubiquitin ligase bound t...
| 超分子 | 名称: Complex of the human DDB1-DDA1-DCAF15 E3 ubiquitin ligase bound to compound furan 24 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: DDB1- and CUL4-associated factor 15
| 分子 | 名称: DDB1- and CUL4-associated factor 15 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 66.822609 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMAPSSKS ERNSGAGSGG GGPGGAGGKR AAGRRREHVL KQLERVKISG QLSPRLFRKL PPRVCVSLKN IVDEDFLYAG HIFLGFSKC GRYVLSYTSS SGDDDFSFYI YHLYWWEFNV HSKLKLVRQV RLFQDEEIYS DLYLTVCEWP SDASKVIVFG F NTRSANGM ...文字列: GSHMAPSSKS ERNSGAGSGG GGPGGAGGKR AAGRRREHVL KQLERVKISG QLSPRLFRKL PPRVCVSLKN IVDEDFLYAG HIFLGFSKC GRYVLSYTSS SGDDDFSFYI YHLYWWEFNV HSKLKLVRQV RLFQDEEIYS DLYLTVCEWP SDASKVIVFG F NTRSANGM LMNMMMMSDE NHRDIYVSTV AVPPPGRCAA CQDASRAHPG DPNAQCLRHG FMLHTKYQVV YPFPTFQPAF QL KKDQVVL LNTSYSLVAC AVSVHSAGDR SFCQILYDHS TCPLAPASPP EPQSPELPPA LPSFCPEAAP ARSSGSPEPS PAI AKAKEF VADIFRRAKE AKGGVPEEAR PALCPGPSGS RCRAHSEPLA LCGETAPRDS PPASEAPASE PGYVNYTKLY YVLE SGEGT EPEDELEDDK ISLPFVVTDL RGRNLRPMRE RTAVQGQYLT VEQLTLDFEY VINEVIRHDA TWGHQFCSFS DYDIV ILEV CPETNQVLIN IGLLLLAFPS PTEEGQLRPK TYHTSLKVAW DLNTGIFETV SVGDLTEVKG QTSGSVWSSY RKSCVD MVM KWLVPESSGR YVNRMTNEAL HKGCSLKVLA DSERYTWIVL UniProtKB: DDB1- and CUL4-associated factor 15 |
-分子 #2: DNA damage-binding protein 1
| 分子 | 名称: DNA damage-binding protein 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 93.347078 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYNYVVTAQ KPTAVNGCVT GHFTSAEDLN LLIAKNTRLE IYVVTAEGLR PVKEVGMYGK IAVMELFRPK GESKDLLFIL TAKYNACIL EYKQSGESID IITRAHGNVQ DRIGRPSETG IIGIIDPECR MIGLRLYDGL FKVIPLDRDN KELKAFNIRL E ELHVIDVK ...文字列: MSYNYVVTAQ KPTAVNGCVT GHFTSAEDLN LLIAKNTRLE IYVVTAEGLR PVKEVGMYGK IAVMELFRPK GESKDLLFIL TAKYNACIL EYKQSGESID IITRAHGNVQ DRIGRPSETG IIGIIDPECR MIGLRLYDGL FKVIPLDRDN KELKAFNIRL E ELHVIDVK FLYGCQAPTI CFVYQDPQGR HVKTYEVSLR EKEFNKGPWK QENVEAEASM VIAVPEPFGG AIIIGQESIT YH NGDKYLA IAPPIIKQST IVCHNRVDPN GSRYLLGDME GRLFMLLLEK EEQMDGTVTL KDLRVELLGE TSIAECLTYL DNG VVFVGS RLGDSQLVKL NVDSNEQGSY VVAMETFTNL GPIVDMCVVD LERQGQGQLV TCSGAFKEGS LRIIRNGIGG NGNS GEIQK LHIRTVPLYE SPRKICYQEV SQCFGVLSSR IEVQDTSGGT TALRPSASTQ ALSSSVSSSK LFSSSTAPHE TSFGE EVEV HNLLIIDQHT FEVLHAHQFL QNEYALSLVS CKLGKDPNTY FIVGTAMVYP EEAEPKQGRI VVFQYSDGKL QTVAEK EVK GAVYSMVEFN GKLLASINST VRLYEWTTEK ELRTECNHYN NIMALYLKTK GDFILVGDLM RSVLLLAYKP MEGNFEE IA RDFNPNWMSA VEILDDDNFL GAENAFNLFV CQKDSAATTD EERQHLQEVG LFHLGEFVNV FCHGSLVMQN LGETSTPT Q GSVLFGTVNG MIGLVTSLSE SWYNLLLDMQ NRLNKVIKSV GKIEHSFWRS FHTERKTEPA TGFIDGDLIE SFLDISRPK MQEVVANLQY DDGSGMKREA TADDLIKVVE ELTRIH UniProtKB: DNA damage-binding protein 1, DNA damage-binding protein 1 |
-分子 #3: DET1- and DDB1-associated protein 1
| 分子 | 名称: DET1- and DDB1-associated protein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 11.855297 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MADFLKGLPV YNKSNFSRFH ADSVCKASNR RPSVYLPTRE YPSEQIIVTE KTNILLRYLH QQWDKKNAAK KRDQEQVELE GESSAPPRK VARTDSPDMH EDT UniProtKB: DET1- and DDB1-associated protein 1 |
-分子 #4: 1-[5-[[3,4-bis(chloranyl)-1~{H}-indol-7-yl]sulfamoyl]-3-methyl-fu...
| 分子 | 名称: 1-[5-[[3,4-bis(chloranyl)-1~{H}-indol-7-yl]sulfamoyl]-3-methyl-furan-2-yl]carbonyl-~{N}-methyl-piperidine-4-carboxamide タイプ: ligand / ID: 4 / コピー数: 1 / 式: A1H18 |
|---|---|
| 分子量 | 理論値: 513.394 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 405189 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)