+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | INTS9-INTS11-BRAT1-WDR73 complex | |||||||||||||||||||||||||||
 マップデータ マップデータ | Overall map | |||||||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||||||||
 キーワード キーワード | Chaperone / nuclear import / Integrator complex / TRANSCRIPTION | |||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報snRNA 3'-end processing / snRNA processing / mitochondrion localization / integrator complex / regulation of transcription elongation by RNA polymerase II / 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ / response to ionizing radiation / nucleus organization / cleavage furrow / RNA polymerase II transcribes snRNA genes ...snRNA 3'-end processing / snRNA processing / mitochondrion localization / integrator complex / regulation of transcription elongation by RNA polymerase II / 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ / response to ionizing radiation / nucleus organization / cleavage furrow / RNA polymerase II transcribes snRNA genes / cytoplasmic microtubule organization / RNA endonuclease activity / negative regulation of transforming growth factor beta receptor signaling pathway / spindle pole / glucose metabolic process / cell migration / positive regulation of cell growth / cell population proliferation / blood microparticle / positive regulation of protein phosphorylation / apoptotic process / DNA damage response / negative regulation of apoptotic process / nucleoplasm / membrane / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||||||||
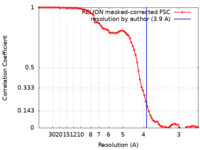
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||||||||||||||||||||
 データ登録者 データ登録者 | Sabath K / Qiu C / Jonas S | |||||||||||||||||||||||||||
| 資金援助 |  スイス, European Union, 8件 スイス, European Union, 8件
| |||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2024 ジャーナル: Mol Cell / 年: 2024タイトル: Assembly mechanism of Integrator's RNA cleavage module. 著者: Kevin Sabath / Chunhong Qiu / Stefanie Jonas /  要旨: The modular Integrator complex is a transcription regulator that is essential for embryonic development. It attenuates coding gene expression via premature transcription termination and performs 3'- ...The modular Integrator complex is a transcription regulator that is essential for embryonic development. It attenuates coding gene expression via premature transcription termination and performs 3'-processing of non-coding RNAs. For both activities, Integrator requires endonuclease activity that is harbored by an RNA cleavage module consisting of INTS4-9-11. How correct assembly of Integrator modules is achieved remains unknown. Here, we show that BRAT1 and WDR73 are critical biogenesis factors for the human cleavage module. They maintain INTS9-11 inactive during maturation by physically blocking the endonuclease active site and prevent premature INTS4 association. Furthermore, BRAT1 facilitates import of INTS9-11 into the nucleus, where it is joined by INTS4. Final BRAT1 release requires locking of the mature cleavage module conformation by inositol hexaphosphate (IP). Our data explain several neurodevelopmental disorders caused by BRAT1, WDR73, and INTS11 mutations as Integrator assembly defects and reveal that IP is an essential co-factor for cleavage module maturation. | |||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18833.map.gz emd_18833.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18833-v30.xml emd-18833-v30.xml emd-18833.xml emd-18833.xml | 24.1 KB 24.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18833_fsc.xml emd_18833_fsc.xml | 7.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18833.png emd_18833.png | 72.6 KB | ||
| Filedesc metadata |  emd-18833.cif.gz emd-18833.cif.gz | 7.2 KB | ||
| その他 |  emd_18833_additional_1.map.gz emd_18833_additional_1.map.gz emd_18833_additional_2.map.gz emd_18833_additional_2.map.gz emd_18833_half_map_1.map.gz emd_18833_half_map_1.map.gz emd_18833_half_map_2.map.gz emd_18833_half_map_2.map.gz | 1 MB 1.4 MB 31.5 MB 31.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18833 http://ftp.pdbj.org/pub/emdb/structures/EMD-18833 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18833 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18833 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18833_validation.pdf.gz emd_18833_validation.pdf.gz | 727.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18833_full_validation.pdf.gz emd_18833_full_validation.pdf.gz | 726.8 KB | 表示 | |
| XML形式データ |  emd_18833_validation.xml.gz emd_18833_validation.xml.gz | 13.4 KB | 表示 | |
| CIF形式データ |  emd_18833_validation.cif.gz emd_18833_validation.cif.gz | 19.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18833 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18833 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18833 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18833 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18833.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18833.map.gz / 形式: CCP4 / 大きさ: 40.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
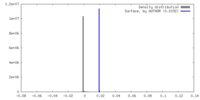
| 注釈 | Overall map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.32 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Locally refined cryo-EM map around INTS11-WDR73
| ファイル | emd_18833_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
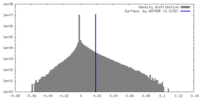
| 注釈 | Locally refined cryo-EM map around INTS11-WDR73 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Locally refined cryo-EM map around INTS9-BRAT1
| ファイル | emd_18833_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally refined cryo-EM map around INTS9-BRAT1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1
| ファイル | emd_18833_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
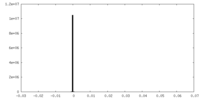
| 注釈 | Half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2
| ファイル | emd_18833_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
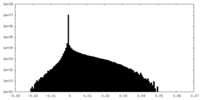
| 注釈 | Half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Protein Complex
| 全体 | 名称: Protein Complex |
|---|---|
| 要素 |
|
-超分子 #1: Protein Complex
| 超分子 | 名称: Protein Complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: BRCA1-associated ATM activator 1
| 分子 | 名称: BRCA1-associated ATM activator 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 88.766328 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GPSQSNMDPE CAQLLPALCA VLVDPRQPVA DDTCLEKLLD WFKTVTEGES SVVLLQEHPC LVELLSHVLK VQDLSSGVLS FSLRLAGTF AAQENCFQYL QQGELLPGLF GEPGPLGRAT WAVPTVRSGW IQGLRSLAQH PSALRFLADH GAVDTIFSLQ G DSSLFVAS ...文字列: GPSQSNMDPE CAQLLPALCA VLVDPRQPVA DDTCLEKLLD WFKTVTEGES SVVLLQEHPC LVELLSHVLK VQDLSSGVLS FSLRLAGTF AAQENCFQYL QQGELLPGLF GEPGPLGRAT WAVPTVRSGW IQGLRSLAQH PSALRFLADH GAVDTIFSLQ G DSSLFVAS AASQLLVHVL ALSMRGGAEG QPCLPGGDWP ACAQKIMDHV EESLCSAATP KVTQALNVLT TTFGRCQSPW TE ALWVRLS PRVACLLERD PIPAAHSFVD LLLCVARSPV FSSSDGSLWE TVARALSCLG PTHMGPLALG ILKLEHCPQA LRT QAFQVL LQPLACVLKA TVQAPGPPGL LDGTADDATT VDTLLASKSS CAGLLCRTLA HLEELQPLPQ RPSPWPQASL LGAT VTVLR LCDGSAAPAS SVGGHLCGTL AGCVRVQRAA LDFLGTLSQG TGPQELVTQA LAVLLECLES PGSSPTVLKK AFQAT LRWL LSSPKTPGCS DLGPLIPQFL RELFPVLQKR LCHPCWEVRD SALEFLTQLS RHWGGQADFR CALLASEVPQ LALQLL QDP ESYVRASAVT AMGQLSSQGL HAPTSPEHAE ARQSLFLELL HILSVDSEGF PRRAVMQVFT EWLRDGHADA AQDTEQF VA TVLQAASRDL DWEVRAQGLE LALVFLGQTL GPPRTHCPYA VALPEVAPAQ PLTEALRALC HVGLFDFAFC ALFDCDRP V AQKSCDLLLF LRDKIASYSS LREARGSPNT ASAEATLPRW RAGEQAQPPG DQEPEAVLAM LRSLDLEGLR STLAESSDH VEKSPQSLLQ DMLATGGFLQ GDEADCY UniProtKB: BRCA1-associated ATM activator 1 |
-分子 #2: Integrator complex subunit 9
| 分子 | 名称: Integrator complex subunit 9 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 73.891219 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MKLYCLSGHP TLPCNVLKFK STTIMLDCGL DMTSTLNFLP LPLVQSPRLS NLPGWSLKDG NAFLDKELKE CSGHVFVDSV PEFCLPETE LIDLSTVDVI LISNYHCMMA LPYITEHTGF TGTVYATEPT VQIGRLLMEE LVNFIERVPK AQSASLWKNK D IQRLLPSP ...文字列: MKLYCLSGHP TLPCNVLKFK STTIMLDCGL DMTSTLNFLP LPLVQSPRLS NLPGWSLKDG NAFLDKELKE CSGHVFVDSV PEFCLPETE LIDLSTVDVI LISNYHCMMA LPYITEHTGF TGTVYATEPT VQIGRLLMEE LVNFIERVPK AQSASLWKNK D IQRLLPSP LKDAVEVSTW RRCYTMQEVN SALSKIQLVG YSQKIELFGA VQVTPLSSGY ALGSSNWIIQ SHYEKVSYVS GS SLLTTHP QPMDQASLKN SDVLVLTGLT QIPTANPDGM VGEFCSNLAL TVRNGGNVLV PCYPSGVIYD LLECLYQYID SAG LSSVPL YFISPVANSS LEFSQIFAEW LCHNKQSKVY LPEPPFPHAE LIQTNKLKHY PSIHGDFSND FRQPCVVFTG HPSL RFGDV VHFMELWGKS SLNTVIFTEP DFSYLEALAP YQPLAMKCIY CPIDTRLNFI QVSKLLKEVQ PLHVVCPEQY TQPPP AQSH RMDLMIDCQP PAMSYRRAEV LALPFKRRYE KIEIMPELAD SLVPMEIKPG ISLATVSAVL HTKDNKHLLQ PPPRPA QPT SGKKRKRVSD DVPDCKVLKP LLSGSIPVEQ FVQTLEKHGF SDIKVEDTAK GHIVLLQEAE TLIQIEEDST HIICDND EM LRVRLRDLVL KFLQKF UniProtKB: Integrator complex subunit 9 |
-分子 #3: Integrator complex subunit 11
| 分子 | 名称: Integrator complex subunit 11 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 68.997906 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GPSDPGPKRA EFMPEIRVTP LGAGQDVGRS CILVSIAGKN VMLDCGMHMG FNDDRRFPDF SYITQNGRLT DFLDCVIISH FHLDHCGAL PYFSEMVGYD GPIYMTHPTQ AICPILLEDY RKIAVDKKGE ANFFTSQMIK DCMKKVVAVH LHQTVQVDDE L EIKAYYAG ...文字列: GPSDPGPKRA EFMPEIRVTP LGAGQDVGRS CILVSIAGKN VMLDCGMHMG FNDDRRFPDF SYITQNGRLT DFLDCVIISH FHLDHCGAL PYFSEMVGYD GPIYMTHPTQ AICPILLEDY RKIAVDKKGE ANFFTSQMIK DCMKKVVAVH LHQTVQVDDE L EIKAYYAG HVLGAAMFQI KVGSESVVYT GDYNMTPDRH LGAAWIDKCR PNLLITESTY ATTIRDSKRC RERDFLKKVH ET VERGGKV LIPVFALGRA QELCILLETF WERMNLKVPI YFSTGLTEKA NHYYKLFIPW TNQKIRKTFV QRNMFEFKHI KAF DRAFAD NPGPMVVFAT PGMLHAGQSL QIFRKWAGNE KNMVIMPGYC VQGTVGHKIL SGQRKLEMEG RQVLEVKMQV EYMS FSAHA DAKGIMQLVG QAEPESVLLV HGEAKKMEFL KQKIEQELRV NCYMPANGET VTLPTSPSIP VGISLGLLKR EMAQG LLPE AKKPRLLHGT LIMKDSNFRL VSSEQALKEL GLAEHQLRFT CRVHLHDTRK EQETALRVYS HLKSVLKDHC VQHLPD GSV TVESVLLQAA APSEDPGTKV LLVSWTYQDE ELGSFLTSLL KKGLPQAPS UniProtKB: Integrator complex subunit 11 |
-分子 #4: WD repeat-containing protein 73
| 分子 | 名称: WD repeat-containing protein 73 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 42.391574 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: GPSQSNSMDP GDDWLVESLR LYQDFYAFDL SGATRVLEWI DDKGVFVAGY ESLKKNEILH LKLPLRLSVK ENKGLFPERD FKVRHGGFS DRSIFDLKHV PHTRLLVTSG LPGCYLQVWQ VAEDSDVIKA VSTIAVHEKE ESLWPRVAVF STLAPGVLHG A RLRSLQVV ...文字列: GPSQSNSMDP GDDWLVESLR LYQDFYAFDL SGATRVLEWI DDKGVFVAGY ESLKKNEILH LKLPLRLSVK ENKGLFPERD FKVRHGGFS DRSIFDLKHV PHTRLLVTSG LPGCYLQVWQ VAEDSDVIKA VSTIAVHEKE ESLWPRVAVF STLAPGVLHG A RLRSLQVV DLESRKTTYT SDVSDSEELS SLQVLDADTF AFCCASGRLG LVDTRQKWAP LENRSPGPGS GGERWCAEVG SW GQGPGPS IASLGSDGRL CLLDPRDLCH PVSSVQCPVS VPSPDPELLR VTWAPGLKNC LAISGFDGTV QVYDATSWDG TRS QDGTRS QVEPLFTHRG HIFLDGNGMD PAPLVTTHTW HPCRPRTLLS ATNDASLHVW DWVDLCAPR UniProtKB: WD repeat-containing protein 73 |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 63.2 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.4 µm / 最小 デフォーカス(公称値): 1.3 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)