+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Chimeric Adenovirus-derived dodecamer | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Adenovirus / capsid protein / virus-like particle / VIRUS LIKE PARTICLE | |||||||||
| 機能・相同性 | Adenovirus penton base protein / Adenovirus penton base protein / T=25 icosahedral viral capsid / endocytosis involved in viral entry into host cell / host cell nucleus / virion attachment to host cell / structural molecule activity / metal ion binding / Penton protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Human adenovirus sp. (ヒトアデノウイルス) / Human adenovirus sp. (ヒトアデノウイルス) /  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.2 Å | |||||||||
 データ登録者 データ登録者 | Buzas D / Borucu U / Bufton J / Kapadalakere SY / Toelzer C | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2024 ジャーナル: Structure / 年: 2024タイトル: Engineering the ADDobody protein scaffold for generation of high-avidity ADDomer super-binders. 著者: Dora Buzas / Huan Sun / Christine Toelzer / Sathish K N Yadav / Ufuk Borucu / Gunjan Gautam / Kapil Gupta / Joshua C Bufton / Julien Capin / Richard B Sessions / Frederic Garzoni / Imre ...著者: Dora Buzas / Huan Sun / Christine Toelzer / Sathish K N Yadav / Ufuk Borucu / Gunjan Gautam / Kapil Gupta / Joshua C Bufton / Julien Capin / Richard B Sessions / Frederic Garzoni / Imre Berger / Christiane Schaffitzel /  要旨: Adenovirus-derived nanoparticles (ADDomer) comprise 60 copies of adenovirus penton base protein (PBP). ADDomer is thermostable, rendering the storage, transport, and deployment of ADDomer-based ...Adenovirus-derived nanoparticles (ADDomer) comprise 60 copies of adenovirus penton base protein (PBP). ADDomer is thermostable, rendering the storage, transport, and deployment of ADDomer-based therapeutics independent of a cold chain. To expand the scope of ADDomers for new applications, we engineered ADDobodies, representing PBP crown domain, genetically separated from PBP multimerization domain. We inserted heterologous sequences into hyper-variable loops, resulting in monomeric, thermostable ADDobodies expressed at high yields in Escherichia coli. The X-ray structure of an ADDobody prototype validated our design. ADDobodies can be used in ribosome display experiments to select a specific binder against a target, with an enrichment factor of ∼10-fold per round. ADDobodies can be re-converted into ADDomers by genetically reconnecting the selected ADDobody with the PBP multimerization domain from a different species, giving rise to a multivalent nanoparticle, called Chimera, confirmed by a 2.2 Å electron cryo-microscopy structure. Chimera comprises 60 binding sites, resulting in ultra-high, picomolar avidity to the target. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18323.map.gz emd_18323.map.gz | 36.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18323-v30.xml emd-18323-v30.xml emd-18323.xml emd-18323.xml | 14.3 KB 14.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18323_fsc.xml emd_18323_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18323.png emd_18323.png | 77.2 KB | ||
| マスクデータ |  emd_18323_msk_1.map emd_18323_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18323.cif.gz emd-18323.cif.gz | 5.7 KB | ||
| その他 |  emd_18323_half_map_1.map.gz emd_18323_half_map_1.map.gz emd_18323_half_map_2.map.gz emd_18323_half_map_2.map.gz | 190.8 MB 190.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18323 http://ftp.pdbj.org/pub/emdb/structures/EMD-18323 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18323 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18323 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18323_validation.pdf.gz emd_18323_validation.pdf.gz | 894.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18323_full_validation.pdf.gz emd_18323_full_validation.pdf.gz | 894.1 KB | 表示 | |
| XML形式データ |  emd_18323_validation.xml.gz emd_18323_validation.xml.gz | 20.8 KB | 表示 | |
| CIF形式データ |  emd_18323_validation.cif.gz emd_18323_validation.cif.gz | 27.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18323 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18323 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18323 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18323 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8qbxMC  8coiC  8qb3C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18323.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18323.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||
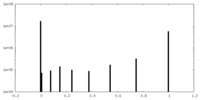
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18323_msk_1.map emd_18323_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
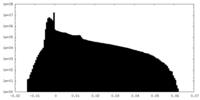
| 密度ヒストグラム |
-ハーフマップ: half-map1
| ファイル | emd_18323_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
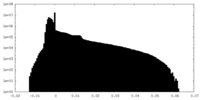
| 密度ヒストグラム |
-ハーフマップ: half-map2
| ファイル | emd_18323_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : unidentified adenovirus
| 全体 | 名称:  unidentified adenovirus (ウイルス) unidentified adenovirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: unidentified adenovirus
| 超分子 | 名称: unidentified adenovirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10535 / 生物種: unidentified adenovirus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes |
|---|
-分子 #1: Penton protein
| 分子 | 名称: Penton protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 60 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human adenovirus sp. (ヒトアデノウイルス) Human adenovirus sp. (ヒトアデノウイルス) |
| 分子量 | 理論値: 63.195812 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MMRRAYPEGP PPSYESVMQQ AMAAAAAMQP PLEAPYVPPR YLAPTEGRNS IRYSELSPLY DTTRLYLVDN KSADIASLNY QNDHSNFLT TVVQNNDFTP TEASTQTINF DERSRWGGQL KTIMHTNMPN VNEYMFSNKF KARVMVSRKA PEGEFVTVND G PVNDTYDH ...文字列: MMRRAYPEGP PPSYESVMQQ AMAAAAAMQP PLEAPYVPPR YLAPTEGRNS IRYSELSPLY DTTRLYLVDN KSADIASLNY QNDHSNFLT TVVQNNDFTP TEASTQTINF DERSRWGGQL KTIMHTNMPN VNEYMFSNKF KARVMVSRKA PEGEFVTVND G PVNDTYDH KEDILKYEWF EFILPEGNFS ATMTIDLMNN AIIDNYLEIG RQNGVLESDI GVKFDTRNFR LGWDPETKLI MP GVYTYEA FHPDIVLLPG CGVDFTESRL SNLLGIRKRH PFQEGFKIMY EDLEGGNIPA LLDVTAYEES KKDTTTARET TTL AVAEET SEDVDDDITR GDTYITELEK QKREAAAAEV SRKKELKIQP LEKDSKSRSY NVLEDKINTA YRSWYLSYNY GNPE KGIRS WTLLTTSDVT CGVEQVYWSL PDMMQDPVTF RSTRQVSNYP VVGAELMPVF SKSFYNEQAV YSQQLRQATS LTHVF NRFP ENQILIRPPA PTITTVSENV PALTDHGTLP LRSSIRGVQR VTVTDARRRT CPYVYKALGI VAPRVLSSRT F UniProtKB: Penton protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 1.06 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z
Z Y
Y X
X