+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human KMN network (outer kinetochore) | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | OUTER KINETOCHORE / KMN / COMPLEX / CELL CYCLE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Knl1/Spc105 complex / positive regulation of meiosis I spindle assembly checkpoint / homologous chromosome orientation in meiotic metaphase I / MIS12/MIND type complex / skeletal muscle satellite cell proliferation / Ndc80 complex / leucine zipper domain binding / acrosome assembly / regulation of mitotic cell cycle spindle assembly checkpoint / attachment of spindle microtubules to kinetochore ...Knl1/Spc105 complex / positive regulation of meiosis I spindle assembly checkpoint / homologous chromosome orientation in meiotic metaphase I / MIS12/MIND type complex / skeletal muscle satellite cell proliferation / Ndc80 complex / leucine zipper domain binding / acrosome assembly / regulation of mitotic cell cycle spindle assembly checkpoint / attachment of spindle microtubules to kinetochore / kinetochore assembly / attachment of mitotic spindle microtubules to kinetochore / protein localization to kinetochore / mitotic spindle assembly checkpoint signaling / mitotic sister chromatid segregation / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / Deposition of new CENPA-containing nucleosomes at the centromere / Resolution of Sister Chromatid Cohesion / mitotic spindle organization / acrosomal vesicle / chromosome segregation / RHO GTPases Activate Formins / kinetochore / fibrillar center / spindle pole / Separation of Sister Chromatids / azurophil granule lumen / microtubule binding / transcription regulator complex / transcription by RNA polymerase II / transcription coactivator activity / nuclear body / nuclear speck / cell division / intracellular membrane-bounded organelle / Neutrophil degranulation / nucleolus / Golgi apparatus / extracellular region / nucleoplasm / nucleus / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
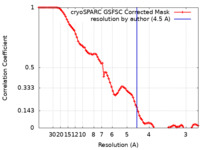
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.5 Å | |||||||||
 データ登録者 データ登録者 | Raisch T / Polley S / Vetter I / Musacchio A / Raunser S | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Insights into human outer kinetochore assembly and force transmission from a structure-function analysis of the KMN network 著者: Polley S / Raisch T / Aponte C / Koerner M / Terbeck M / Raunser S / Graeter F / Vetter I / Musacchio A | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18179.map.gz emd_18179.map.gz | 59.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18179-v30.xml emd-18179-v30.xml emd-18179.xml emd-18179.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18179_fsc.xml emd_18179_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18179.png emd_18179.png | 72.1 KB | ||
| マスクデータ |  emd_18179_msk_1.map emd_18179_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18179.cif.gz emd-18179.cif.gz | 6.5 KB | ||
| その他 |  emd_18179_additional_1.map.gz emd_18179_additional_1.map.gz emd_18179_additional_2.map.gz emd_18179_additional_2.map.gz emd_18179_half_map_1.map.gz emd_18179_half_map_1.map.gz emd_18179_half_map_2.map.gz emd_18179_half_map_2.map.gz | 32.1 MB 57.2 MB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18179 http://ftp.pdbj.org/pub/emdb/structures/EMD-18179 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18179 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18179 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18179_validation.pdf.gz emd_18179_validation.pdf.gz | 997.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18179_full_validation.pdf.gz emd_18179_full_validation.pdf.gz | 997.3 KB | 表示 | |
| XML形式データ |  emd_18179_validation.xml.gz emd_18179_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_18179_validation.cif.gz emd_18179_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18179 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18179 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18179 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18179 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8q5hMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18179.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18179.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.36 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18179_msk_1.map emd_18179_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Non-sharpened map
| ファイル | emd_18179_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Non-sharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Map sharpened by DeepEMhancer
| ファイル | emd_18179_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map sharpened by DeepEMhancer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_18179_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_18179_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : KMN network
| 全体 | 名称: KMN network |
|---|---|
| 要素 |
|
-超分子 #1: KMN network
| 超分子 | 名称: KMN network / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Kinetochore protein Spc24
| 分子 | 名称: Kinetochore protein Spc24 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.344515 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MELKEIEADL ERQEKEVDED TTVTIPSAVY VAQLYHQVSK IEWDYECEPG MVKGIHHGPS VAQPIHLDST QLSRKFISDY LWSLVDTEW UniProtKB: Kinetochore protein Spc24 |
-分子 #2: Kinetochore protein Spc25
| 分子 | 名称: Kinetochore protein Spc25 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 16.285332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKQELEVLTA NIQDLKEEYS RKKETISTAN KANAERLKRL QKSADLYKDR LGLEIRKIYG EKLQFIFTNI DPKNPESPFM FSLHLNEAR DYEVSDSAPH LEGLAEFQEN VRKTNNFSAF LANVRKAFTA TVYNHHHHHH UniProtKB: Kinetochore protein Spc25 |
-分子 #3: Protein MIS12 homolog
| 分子 | 名称: Protein MIS12 homolog / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.170922 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MSVDPMTYEA QFFGFTPQTC MLRIYIAFQD YLFEVMQAVE QVILKKLDGI PDCDISPVQI RKCTEKFLCF MKGHFDNLFS KMEQLFLQL ILRIPSNILL PEDKCKETPY SEEDFQHLQK EIEQLQEKYK TELCTKQALL AELEEQKIVQ AKLKQTLTFF D ELHNVGRD ...文字列: MSVDPMTYEA QFFGFTPQTC MLRIYIAFQD YLFEVMQAVE QVILKKLDGI PDCDISPVQI RKCTEKFLCF MKGHFDNLFS KMEQLFLQL ILRIPSNILL PEDKCKETPY SEEDFQHLQK EIEQLQEKYK TELCTKQALL AELEEQKIVQ AKLKQTLTFF D ELHNVGRD HGTSDFRESL VSLVQNSRKL QNIRDNVEKE SKRLKIS UniProtKB: Protein MIS12 homolog |
-分子 #4: Polyamine-modulated factor 1
| 分子 | 名称: Polyamine-modulated factor 1 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.368311 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAEASSANLG SGCEEKRHEG SSSESVPPGT TISRVKLLDT MVDTFLQKLV AAGSYQRFTD CYKCFYQLQP AMTQQIYDKF IAQLQTSIR EEISDIKEEG NLEAVLNALD KIVEEGKVRK EPAWRPSGIP EKDLHSVMAP YFLQQRDTLR RHVQKQEAEN Q QLADAVLA ...文字列: MAEASSANLG SGCEEKRHEG SSSESVPPGT TISRVKLLDT MVDTFLQKLV AAGSYQRFTD CYKCFYQLQP AMTQQIYDKF IAQLQTSIR EEISDIKEEG NLEAVLNALD KIVEEGKVRK EPAWRPSGIP EKDLHSVMAP YFLQQRDTLR RHVQKQEAEN Q QLADAVLA GRRQVEELQL QVQAQQQAWQ ALHREQRELV AVLREPE UniProtKB: Polyamine-modulated factor 1 |
-分子 #5: Kinetochore-associated protein DSN1 homolog
| 分子 | 名称: Kinetochore-associated protein DSN1 homolog / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 40.951062 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MTSVTRSEII DEKGPVMSKT HDHQLESSLS PVEVFAKTSA SLEMNQGVSE ERIHLGSSPK KGGNCDLSHQ ERLQSKSLHL SPQEQSASY QDRRQSWRRA SMKETNRRKS LHPIHQGITE LSRSISVDLA ESKRLGCLLL SSFQFSIQKL EPFLRDTKGF S LESFRAKA ...文字列: MTSVTRSEII DEKGPVMSKT HDHQLESSLS PVEVFAKTSA SLEMNQGVSE ERIHLGSSPK KGGNCDLSHQ ERLQSKSLHL SPQEQSASY QDRRQSWRRA SMKETNRRKS LHPIHQGITE LSRSISVDLA ESKRLGCLLL SSFQFSIQKL EPFLRDTKGF S LESFRAKA SSLSEELKHF ADGLETDGTL QKCFEDSNGK ASDFSLEASV AEMKEYITKF SLERQTWDQL LLHYQQEAKE IL SRGSTEA KITEVKVEPM TYLGSSQNEV LNTKPDYQKI LQNQSKVFDC MELVMDELQG SVKQLQAFMD ESTQCFQKVS VQL GKRSMQ QLDPSPARKL LKLQLQNPPA IHGSGSGSCQ HHHHHH UniProtKB: Kinetochore-associated protein DSN1 homolog |
-分子 #6: Kinetochore scaffold 1
| 分子 | 名称: Kinetochore scaffold 1 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 38.644453 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GAMGGSMGKL VQSAQNEREK LQIKIDEMDK ILKKIDNCLT EMETETKNLE DEEKNNPVEE WDSEMRAAEK ELEQLKTEEE ELQRNLLEL EVQKEQTLAQ IDFMQKQRNR TEELLDQLSL SEWDVVEWSD DQAVFTFVYD TIQLTITFEE SVVGFPFLDK R YRKIVDVN ...文字列: GAMGGSMGKL VQSAQNEREK LQIKIDEMDK ILKKIDNCLT EMETETKNLE DEEKNNPVEE WDSEMRAAEK ELEQLKTEEE ELQRNLLEL EVQKEQTLAQ IDFMQKQRNR TEELLDQLSL SEWDVVEWSD DQAVFTFVYD TIQLTITFEE SVVGFPFLDK R YRKIVDVN FQSLLDEDQA PPSSLLVHKL IFQYVEEKES WKKTCTTQHQ LPKMLEEFSL VVHHCRLLGE EIEYLKRWGP NY NLMNIDI NNNELRLLFS SSAAFAKFEI TLFLSAYYPS VPLPSTIQNH VGNTSQDDIA TILSKVPLEN NYLKNVVKQI YQD LFQDCH FYH UniProtKB: Kinetochore scaffold 1 |
-分子 #7: Kinetochore-associated protein NSL1 homolog
| 分子 | 名称: Kinetochore-associated protein NSL1 homolog / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 32.208951 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MAGSPELVVL DPPWDKELAA GTESQALVSA TPREDFRVRC TSKRAVTEML QLCGRFVQKL GDALPEEIRE PALRDAQWTF ESAVQENIS INGQAWQEAS DNCFMDSDIK VLEDQFDEII VDIATKRKQY PRKILECVIK TIKAKQEILK QYHPVVHPLD L KYDPDPAP ...文字列: MAGSPELVVL DPPWDKELAA GTESQALVSA TPREDFRVRC TSKRAVTEML QLCGRFVQKL GDALPEEIRE PALRDAQWTF ESAVQENIS INGQAWQEAS DNCFMDSDIK VLEDQFDEII VDIATKRKQY PRKILECVIK TIKAKQEILK QYHPVVHPLD L KYDPDPAP HMENLKCRGE TVAKEISEAM KSLPALIEQG EGFSQVLRMQ PVIHLQRIHQ EVFSSCHRKP DAKPENFITQ IE TTPTETA SRKTSDMVLK RKQTKDCPQR KWYPLRPKKI NLDT UniProtKB: Kinetochore-associated protein NSL1 homolog |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 63.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X