+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cas1-Cas2 CRISPR integrase bound to prespacer and target DNA, Streptococcus thermophilus DGCC 7710 CRISPR3 system | |||||||||
 マップデータ マップデータ | Map autosharpened using phenix.auto_sharpen_1.21rc1-4903 and b_iso_to_d_cut | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Cas1-Cas2 integrase / CRISPR-Cas / prespacer / CRISPR leader / CRISPR repeat / spacer acquisition / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報maintenance of CRISPR repeat elements / RNA endonuclease activity / DNA endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / synthetic construct (人工物) Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / synthetic construct (人工物) | |||||||||
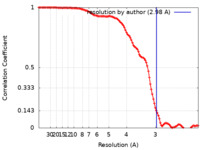
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.98 Å | |||||||||
 データ登録者 データ登録者 | Sasnauskas G / Gaizauskaite U / Tamulaitiene G | |||||||||
| 資金援助 | リトアニア, 1件
| |||||||||
 引用 引用 |  ジャーナル: To be published ジャーナル: To be publishedタイトル: Structural basis for spacer acquisition in a type II-A CRISPR-Cas system 著者: Sasnauskas G / Gaizauskaite U / Tamulaitiene G | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18113.map.gz emd_18113.map.gz | 109.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18113-v30.xml emd-18113-v30.xml emd-18113.xml emd-18113.xml | 23.4 KB 23.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18113_fsc.xml emd_18113_fsc.xml | 14.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18113.png emd_18113.png | 134.2 KB | ||
| マスクデータ |  emd_18113_msk_1.map emd_18113_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18113.cif.gz emd-18113.cif.gz | 6.7 KB | ||
| その他 |  emd_18113_additional_1.map.gz emd_18113_additional_1.map.gz emd_18113_half_map_1.map.gz emd_18113_half_map_1.map.gz emd_18113_half_map_2.map.gz emd_18113_half_map_2.map.gz | 62.7 MB 116 MB 115.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18113 http://ftp.pdbj.org/pub/emdb/structures/EMD-18113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18113 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18113 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_18113_validation.pdf.gz emd_18113_validation.pdf.gz | 846.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_18113_full_validation.pdf.gz emd_18113_full_validation.pdf.gz | 845.6 KB | 表示 | |
| XML形式データ |  emd_18113_validation.xml.gz emd_18113_validation.xml.gz | 19 KB | 表示 | |
| CIF形式データ |  emd_18113_validation.cif.gz emd_18113_validation.cif.gz | 24.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18113 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18113 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-18113 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18113.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18113.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map autosharpened using phenix.auto_sharpen_1.21rc1-4903 and b_iso_to_d_cut | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18113_msk_1.map emd_18113_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
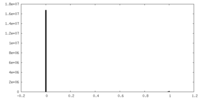
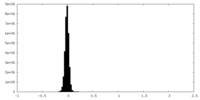
| 投影像・断面図 |
| ||||||||||||
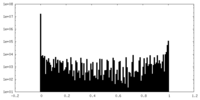
| 密度ヒストグラム |
-追加マップ: Unsharpened main map
| ファイル | emd_18113_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened main map | ||||||||||||
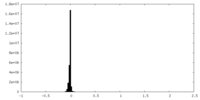
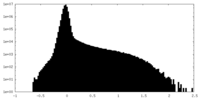
| 投影像・断面図 |
| ||||||||||||
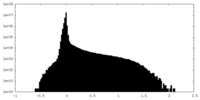
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18113_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
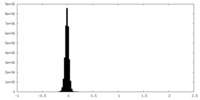
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18113_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
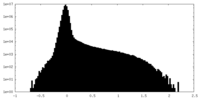
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cas1-Cas2 CRISPR integrase bound to prespacer and target DNA, Str...
| 全体 | 名称: Cas1-Cas2 CRISPR integrase bound to prespacer and target DNA, Streptococcus thermophilus DGCC 7710 CRISPR3 system |
|---|---|
| 要素 |
|
-超分子 #1: Cas1-Cas2 CRISPR integrase bound to prespacer and target DNA, Str...
| 超分子 | 名称: Cas1-Cas2 CRISPR integrase bound to prespacer and target DNA, Streptococcus thermophilus DGCC 7710 CRISPR3 system タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
-分子 #1: CRISPR-associated endoribonuclease Cas2
| 分子 | 名称: CRISPR-associated endoribonuclease Cas2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 13.43156 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSYRYMRMIL MFDMPTDTAE ERKAYRKFRK FLLSEGFIMH QFSVYSKLLL NHTANTAMVG RLKANNPKKG NITILTVTEK QFARMIYLY GDKNTSIANS EERLVFLGDN YCDED UniProtKB: CRISPR-associated endoribonuclease Cas2 |
-分子 #2: CRISPR-associated endonuclease Cas1
| 分子 | 名称: CRISPR-associated endonuclease Cas1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 4 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus DGCC 7710 (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 35.363461 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MAGWRTVVVN IHSKLSYKNN HLIFRNSYKT EMIHLSEIDI LLLETTDIVL TTMLVKRLVD ENILVIFCDD KRLPTAFLTP YYARHDSSL QIARQIAWKE NVKCEVWTAI IAQKILNQSY YLGECSFFEK SQSIMELYHG LERFDPSNRE GHSARIYFNT L FGNDFTRE ...文字列: MAGWRTVVVN IHSKLSYKNN HLIFRNSYKT EMIHLSEIDI LLLETTDIVL TTMLVKRLVD ENILVIFCDD KRLPTAFLTP YYARHDSSL QIARQIAWKE NVKCEVWTAI IAQKILNQSY YLGECSFFEK SQSIMELYHG LERFDPSNRE GHSARIYFNT L FGNDFTRE SDNDINAALD YGYTLLLSMF AREVVVCGCM TQIGLKHANQ FNQFNLASDI MEPFRPIIDR IVYQNRHNNF VK IKKELFS IFSETYLYNG KEMYLSNIVS DYTKKVIKAL NQLGEEIPEF RILESGWSHP QFEKA UniProtKB: CRISPR-associated endonuclease Cas1 |
-分子 #3: Prespacer DNA, chain G
| 分子 | 名称: Prespacer DNA, chain G / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 8.062258 KDa |
| 配列 | 文字列: (DA)(DA)(DA)(DC)(DA)(DC)(DC)(DA)(DG)(DA) (DA)(DC)(DG)(DA)(DG)(DT)(DA)(DG)(DT)(DA) (DA)(DA)(DT)(DT)(DG)(DG) |
-分子 #4: Prespacer DNA, chain H
| 分子 | 名称: Prespacer DNA, chain H / タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 7.917082 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DA)(DC)(DT)(DA)(DC)(DT)(DC) (DG)(DT)(DT)(DC)(DT)(DG)(DG)(DT)(DG)(DT) (DT)(DT)(DC)(DT)(DC)(DG) |
-分子 #5: Integration target, chain I
| 分子 | 名称: Integration target, chain I / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.729506 KDa |
| 配列 | 文字列: (DT)(DA)(DC)(DG)(DA)(DG)(DG)(DT)(DT)(DT) (DT)(DG)(DG)(DA)(DA)(DC)(DC)(DA)(DT)(DT) (DC)(DG)(DA)(DA)(DA)(DC)(DA)(DA)(DC) (DA)(DC)(DA)(DG)(DC)(DT)(DC)(DT)(DA)(DA) (DA) (DA)(DC)(DC)(DT)(DC)(DG)(DT)(DA) |
-分子 #6: Integration target, chain J
| 分子 | 名称: Integration target, chain J / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.835518 KDa |
| 配列 | 文字列: (DT)(DA)(DC)(DG)(DA)(DG)(DG)(DT)(DT)(DT) (DT)(DA)(DG)(DA)(DG)(DC)(DT)(DG)(DT)(DG) (DT)(DT)(DG)(DT)(DT)(DT)(DC)(DG)(DA) (DA)(DT)(DG)(DG)(DT)(DT)(DC)(DC)(DA)(DA) (DA) (DA)(DC)(DC)(DT)(DC)(DG)(DT)(DA) |
-分子 #7: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 7 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 3129 / 平均露光時間: 46.33 sec. / 平均電子線量: 29.7 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: OTHER / 撮影モード: OTHER / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 92000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 得られたモデル |  PDB-8q2n: |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X