+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Full-length bacterial polysaccharide co-polymerase WzzE mutant R267A from E. coli. C4 symmetry | ||||||||||||
 マップデータ マップデータ | E. coli WzzE mutant R267A, C4 symmetry, unsharpened | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Complex / Lipopolysaccharide / bacterial polysaccharide co-polymerase / MEMBRANE PROTEIN | ||||||||||||
| 機能・相同性 | ECA polysaccharide chain length modulation protein WzzE / enterobacterial common antigen biosynthetic process / Polysaccharide chain length determinant N-terminal domain / Chain length determinant protein / : / protein tyrosine kinase activity / plasma membrane / ECA polysaccharide chain length modulation protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  | ||||||||||||
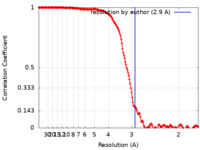
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||||||||
 データ登録者 データ登録者 | Wiseman B / Hogbom M | ||||||||||||
| 資金援助 |  スウェーデン, 3件 スウェーデン, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: Alternating L4 loop architecture of the bacterial polysaccharide co-polymerase WzzE. 著者: Benjamin Wiseman / Göran Widmalm / Martin Högbom /  要旨: Lipopolysaccharides such as the enterobacterial common antigen are important components of the enterobacterial cell envelope that act as a protective barrier against the environment and are often ...Lipopolysaccharides such as the enterobacterial common antigen are important components of the enterobacterial cell envelope that act as a protective barrier against the environment and are often polymerized by the inner membrane bound Wzy-dependent pathway. By employing cryo-electron microscopy we show that WzzE, the co-polymerase component of this pathway that is responsible for the length modulation of the enterobacterial common antigen, is octameric with alternating up-down conformations of its L4 loops. The alternating up-down nature of these essential loops, located at the top of the periplasmic bell, are modulated by clashing helical faces between adjacent protomers that flank the L4 loops around the octameric periplasmic bell. This alternating arrangement and a highly negatively charged binding face create a dynamic environment in which the polysaccharide chain is extended, and suggest a ratchet-type mechanism for polysaccharide elongation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17387.map.gz emd_17387.map.gz | 194.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17387-v30.xml emd-17387-v30.xml emd-17387.xml emd-17387.xml | 21.4 KB 21.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17387_fsc.xml emd_17387_fsc.xml | 17.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17387.png emd_17387.png | 83.6 KB | ||
| Filedesc metadata |  emd-17387.cif.gz emd-17387.cif.gz | 6 KB | ||
| その他 |  emd_17387_additional_1.map.gz emd_17387_additional_1.map.gz emd_17387_half_map_1.map.gz emd_17387_half_map_1.map.gz emd_17387_half_map_2.map.gz emd_17387_half_map_2.map.gz | 373.6 MB 367.2 MB 367.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17387 http://ftp.pdbj.org/pub/emdb/structures/EMD-17387 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17387 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17387 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17387_validation.pdf.gz emd_17387_validation.pdf.gz | 731.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17387_full_validation.pdf.gz emd_17387_full_validation.pdf.gz | 731 KB | 表示 | |
| XML形式データ |  emd_17387_validation.xml.gz emd_17387_validation.xml.gz | 25 KB | 表示 | |
| CIF形式データ |  emd_17387_validation.cif.gz emd_17387_validation.cif.gz | 32.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17387 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17387 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17387 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17387 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8p3oMC  8bhwC  8p3pC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17387.map.gz / 形式: CCP4 / 大きさ: 396.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17387.map.gz / 形式: CCP4 / 大きさ: 396.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli WzzE mutant R267A, C4 symmetry, unsharpened | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8676 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: E. coli WzzE mutant R267A, C4 symmetry, sharpened
| ファイル | emd_17387_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli WzzE mutant R267A, C4 symmetry, sharpened | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: E. coli WzzE mutant R267A, C4 symmetry, half map B
| ファイル | emd_17387_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli WzzE mutant R267A, C4 symmetry, half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: E. coli WzzE mutant R267A, C4 symmetry, half map A
| ファイル | emd_17387_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | E. coli WzzE mutant R267A, C4 symmetry, half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Bacterial Polysaccharide Co-polymerase WzzE
| 全体 | 名称: Bacterial Polysaccharide Co-polymerase WzzE |
|---|---|
| 要素 |
|
-超分子 #1: Bacterial Polysaccharide Co-polymerase WzzE
| 超分子 | 名称: Bacterial Polysaccharide Co-polymerase WzzE / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Homo octameric complex |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 320 KDa |
-分子 #1: ECA polysaccharide chain length modulation protein
| 分子 | 名称: ECA polysaccharide chain length modulation protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 41.324906 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTQPMPGKPA EDAENELDIR GLFRTLWAGK LWIIGMGLAF ALIALAYTFF ARQEWSSTAI TDRPTVNMLG GYYSQQQFLR NLDVRSNMA SADQPSVMDE AYKEFVMQLA SWDTRREFWL QTDYYKQRMV GNSKADAALL DEMINNIQFI PGDFTRAVND S VKLIAETA ...文字列: MTQPMPGKPA EDAENELDIR GLFRTLWAGK LWIIGMGLAF ALIALAYTFF ARQEWSSTAI TDRPTVNMLG GYYSQQQFLR NLDVRSNMA SADQPSVMDE AYKEFVMQLA SWDTRREFWL QTDYYKQRMV GNSKADAALL DEMINNIQFI PGDFTRAVND S VKLIAETA PDANNLLRQY VAFASQRAAS HLNDELKGAW AARTIQMKAQ VKRQEEVAKA IYDRRMNSIE QALKIAEQHN IS RSATDVP AEELPDSEMF LLGRPMLQAA LENLQAVGPA FDLDYDQNRA MLNTLNVGPT LDPRFQTYRY LRTPEEPVKR DSP RRAFLM IMWGIVGGLI GAGVALTRRC SKEFRVPGSH HHHHHHH UniProtKB: ECA polysaccharide chain length modulation protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: C-flat-2/2 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 9573 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 1.9000000000000001 µm 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)