+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Setaria italica NRAT in complex with a nanobody | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NRAT / NRAMP / SLC11 / Metal uptake / Aluminium transporter / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 | aluminum ion transmembrane transporter activity / aluminum cation transport / NRAMP family / Natural resistance-associated macrophage protein-like / manganese ion transmembrane transporter activity / cadmium ion transmembrane transporter activity / response to aluminum ion / plasma membrane / Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Setaria italica (アワ) / Setaria italica (アワ) /  | |||||||||
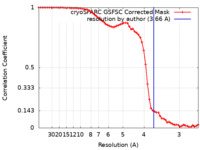
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.66 Å | |||||||||
 データ登録者 データ登録者 | Ramanadane K / Liziczai M / Markovic D / Straub MS / Rosalen GT / Udovcic A / Dutzler R / Manatschal C | |||||||||
| 資金援助 |  スイス, 2件 スイス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2023 ジャーナル: Elife / 年: 2023タイトル: Structural and functional properties of a plant NRAMP-related aluminum transporter. 著者: Karthik Ramanadane / Márton Liziczai / Dragana Markovic / Monique S Straub / Gian T Rosalen / Anto Udovcic / Raimund Dutzler / Cristina Manatschal /  要旨: The transport of transition metal ions by members of the SLC11/NRAMP family constitutes a ubiquitous mechanism for the uptake of Fe and Mn across all kingdoms of life. Despite the strong conservation ...The transport of transition metal ions by members of the SLC11/NRAMP family constitutes a ubiquitous mechanism for the uptake of Fe and Mn across all kingdoms of life. Despite the strong conservation of the family, two of its branches have evolved a distinct substrate preference with one mediating Mg uptake in prokaryotes and another the transport of Al into plant cells. Our previous work on the SLC11 transporter from revealed the basis for its Mg selectivity (Ramanadane et al., 2022). Here, we have addressed the structural and functional properties of a putative Al transporter from . We show that the protein transports diverse divalent metal ions and binds the trivalent ions Al and Ga, which are both presumable substrates. Its cryo-electron microscopy (cryo-EM) structure displays an occluded conformation that is closer to an inward- than an outward-facing state, with a binding site that is remodeled to accommodate the increased charge density of its transported substrate. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
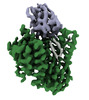
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17000.map.gz emd_17000.map.gz | 11.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17000-v30.xml emd-17000-v30.xml emd-17000.xml emd-17000.xml | 16.7 KB 16.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17000_fsc.xml emd_17000_fsc.xml | 7.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17000.png emd_17000.png | 105.1 KB | ||
| マスクデータ |  emd_17000_msk_1.map emd_17000_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-17000.cif.gz emd-17000.cif.gz | 6.2 KB | ||
| その他 |  emd_17000_half_map_1.map.gz emd_17000_half_map_1.map.gz emd_17000_half_map_2.map.gz emd_17000_half_map_2.map.gz | 20.7 MB 20.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17000 http://ftp.pdbj.org/pub/emdb/structures/EMD-17000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17000 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17000 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_17000_validation.pdf.gz emd_17000_validation.pdf.gz | 746.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_17000_full_validation.pdf.gz emd_17000_full_validation.pdf.gz | 746.2 KB | 表示 | |
| XML形式データ |  emd_17000_validation.xml.gz emd_17000_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_17000_validation.cif.gz emd_17000_validation.cif.gz | 16.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17000 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17000 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-17000 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ontMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17000.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17000.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.302 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_17000_msk_1.map emd_17000_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
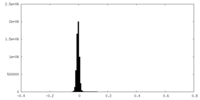
| 投影像・断面図 |
| ||||||||||||
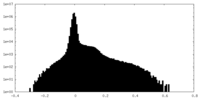
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_17000_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17000_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex between SiNRAT and a nanobody used as a fiducial marker f...
| 全体 | 名称: Complex between SiNRAT and a nanobody used as a fiducial marker for structure determination |
|---|---|
| 要素 |
|
-超分子 #1: Complex between SiNRAT and a nanobody used as a fiducial marker f...
| 超分子 | 名称: Complex between SiNRAT and a nanobody used as a fiducial marker for structure determination タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  Setaria italica (アワ) Setaria italica (アワ) |
-分子 #1: NRAMP related aluminium transporter
| 分子 | 名称: NRAMP related aluminium transporter / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Setaria italica (アワ) Setaria italica (アワ) |
| 分子量 | 理論値: 60.147227 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSERAREVGE TGREARHGVL QSGSDTGDHK DKTVELEKDE QFQGQPKWRK FLAHVGPGAL VAIGFLDPSN LETDMQAGAD FKYELLWVV LVGMIFALLI QTLAANLGVK TGRHLAELCR EEYPRYVNIC LWIIAELAVI SDDIPEVLGT AFAFNILLKI P VWAGVILT ...文字列: MSERAREVGE TGREARHGVL QSGSDTGDHK DKTVELEKDE QFQGQPKWRK FLAHVGPGAL VAIGFLDPSN LETDMQAGAD FKYELLWVV LVGMIFALLI QTLAANLGVK TGRHLAELCR EEYPRYVNIC LWIIAELAVI SDDIPEVLGT AFAFNILLKI P VWAGVILT VFSTLLLLGV QRFGARKLEF IIAAFMFTMA ACFFGELSYL RPSAKEVVKG MFVPSLQGKG AAANAIALFG AI ITPYNLF LHSALVLSRK TPRSVKSIRA ACRYFLIECS LAFIVAFLIN VSVVVVAGTI CNADNLSPTD SNTCSDLTLQ SAP MLLRNV LGRSSSVVYA VALLASGQST TISCTFAGQV IMQGFLDMKM KNWVRNLITR VIAIAPSLIV SIVSGPSGAG KLII FSSMV LSFEMPFALI PLLKFCNSSK KVGPLKESIY TVVIAWILSF ALIVVNTYFL VWTYVDWLVH NHLPKYANAL VSIVV FALM AAYLVFVVYL TFRRDTVSTY VPVSERAQGQ VEAGGAQAVA SAADADQPAP FRKDLADASM ALEVLFQG UniProtKB: Uncharacterized protein |
-分子 #2: Nanobody1
| 分子 | 名称: Nanobody1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.071546 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QWQLVESGGG LVQAGGSLRL SCVGSGRAFS SGAMGWFRQT PGQEREFVAA ISWSGGSTVY AESVKGRFTI SMDNAKNTVY LRMNSLQPE DTAVYYCAAG TSTFALRRSP EYWGKGTPVT VSS |
-分子 #3: DIUNDECYL PHOSPHATIDYL CHOLINE
| 分子 | 名称: DIUNDECYL PHOSPHATIDYL CHOLINE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: PLC |
|---|---|
| 分子量 | 理論値: 622.834 Da |
| Chemical component information |  ChemComp-PLC: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 1 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 69.68 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



 Z
Z Y
Y X
X