+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of bridges of the yeast ER-mitochondria encounter structure (ERMES) | ||||||||||||||||||||||||
 マップデータ マップデータ | Subtomogram average map of ERMES bridges, imaged in yeast cells. | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | ERMES / lipid transfer protein / membrane contact sites / SMP domains / LIPID TRANSPORT | ||||||||||||||||||||||||
| 生物種 |  | ||||||||||||||||||||||||
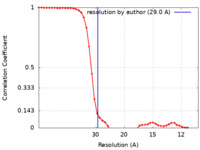
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 29.0 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Wozny MR / Di Luca A / Morado DR / Picco A / Khaddaj R / Campomanes P / Ivanovic L / Hoffmann PC / Miller EA / Vanni S / Kukulski W | ||||||||||||||||||||||||
| 資金援助 |  英国, 英国,  カナダ, カナダ,  スイス, European Union, 7件 スイス, European Union, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: In situ architecture of the ER-mitochondria encounter structure. 著者: Michael R Wozny / Andrea Di Luca / Dustin R Morado / Andrea Picco / Rasha Khaddaj / Pablo Campomanes / Lazar Ivanović / Patrick C Hoffmann / Elizabeth A Miller / Stefano Vanni / Wanda Kukulski /      要旨: The endoplasmic reticulum and mitochondria are main hubs of eukaryotic membrane biogenesis that rely on lipid exchange via membrane contact sites, but the underpinning mechanisms remain poorly ...The endoplasmic reticulum and mitochondria are main hubs of eukaryotic membrane biogenesis that rely on lipid exchange via membrane contact sites, but the underpinning mechanisms remain poorly understood. In yeast, tethering and lipid transfer between the two organelles is mediated by the endoplasmic reticulum-mitochondria encounter structure (ERMES), a four-subunit complex of unresolved stoichiometry and architecture. Here we determined the molecular organization of ERMES within Saccharomyces cerevisiae cells using integrative structural biology by combining quantitative live imaging, cryo-correlative microscopy, subtomogram averaging and molecular modelling. We found that ERMES assembles into approximately 25 discrete bridge-like complexes distributed irregularly across a contact site. Each bridge consists of three synaptotagmin-like mitochondrial lipid binding protein domains oriented in a zig-zag arrangement. Our molecular model of ERMES reveals a pathway for lipids. These findings resolve the in situ supramolecular architecture of a major inter-organelle lipid transfer machinery and provide a basis for the mechanistic understanding of lipid fluxes in eukaryotic cells. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16873.map.gz emd_16873.map.gz | 6.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16873-v30.xml emd-16873-v30.xml emd-16873.xml emd-16873.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16873_fsc.xml emd_16873_fsc.xml | 5.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16873.png emd_16873.png | 134 KB | ||
| その他 |  emd_16873_additional_1.map.gz emd_16873_additional_1.map.gz emd_16873_additional_2.map.gz emd_16873_additional_2.map.gz emd_16873_additional_3.map.gz emd_16873_additional_3.map.gz emd_16873_additional_4.map.gz emd_16873_additional_4.map.gz emd_16873_half_map_1.map.gz emd_16873_half_map_1.map.gz emd_16873_half_map_2.map.gz emd_16873_half_map_2.map.gz | 387.7 KB 388.4 KB 6.1 MB 6.1 MB 6.1 MB 6.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16873 http://ftp.pdbj.org/pub/emdb/structures/EMD-16873 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16873 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16873 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16873_validation.pdf.gz emd_16873_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16873_full_validation.pdf.gz emd_16873_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_16873_validation.xml.gz emd_16873_validation.xml.gz | 9.8 KB | 表示 | |
| CIF形式データ |  emd_16873_validation.cif.gz emd_16873_validation.cif.gz | 13.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16873 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16873 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16873 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16873 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16873.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16873.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average map of ERMES bridges, imaged in yeast cells. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.368 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Half map a, masked. Together with masked half...
| ファイル | emd_16873_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map a, masked. Together with masked half map b, this was used for resolution estimate. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
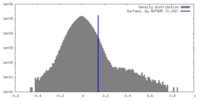
| 密度ヒストグラム |
-追加マップ: Half map b, masked. Together with masked half...
| ファイル | emd_16873_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map b, masked. Together with masked half map a, this was used for resolution estimate. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
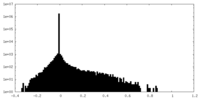
| 密度ヒストグラム |
-追加マップ: Subtomogram average map of ERMES bridges aligned on...
| ファイル | emd_16873_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average map of ERMES bridges aligned on the outer mitochondrial membrane, for determination of the angle between the bridge and the outer mitochondrial membrane normal. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
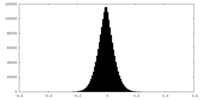
| 密度ヒストグラム |
-追加マップ: Subtomogram average map of ERMES bridges aligned on...
| ファイル | emd_16873_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average map of ERMES bridges aligned on the ER membrane, for determination of the angle between the bridge and the ER normal. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
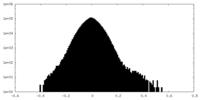
| 密度ヒストグラム |
-ハーフマップ: Half map b, unmasked
| ファイル | emd_16873_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map b, unmasked | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map a, unmasked.
| ファイル | emd_16873_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map a, unmasked. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Budding yeast cells expressing Mdm34-mNeonGreen, a component of t...
| 全体 | 名称: Budding yeast cells expressing Mdm34-mNeonGreen, a component of the ER-mitochondria encounter structure (ERMES). |
|---|---|
| 要素 |
|
-超分子 #1: Budding yeast cells expressing Mdm34-mNeonGreen, a component of t...
| 超分子 | 名称: Budding yeast cells expressing Mdm34-mNeonGreen, a component of the ER-mitochondria encounter structure (ERMES). タイプ: cell / ID: 1 / 親要素: 0 詳細: Yeast cells were cryo-FIB-milled, imaged by cryo-FM and electron cryo-tomograms were acquired at localisations of Mdm34-mNeonGreen fluorescent signals. |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 6 詳細: Synthetic complete medium without tryptophan, with 2% glucose and 15% dextran |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
| 詳細 | Plunge-frozen grids with yeast cells were subjected to cryo-FIB milling followed by cryo-fluorescence microscopy and cryo-ET. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 1.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 6.0 µm / 最小 デフォーカス(公称値): 3.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)