+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RcpA-TadD with C13 symmetry from the Pseudomonas aeruginosa Tight Adherence Secretion System | |||||||||
 マップデータ マップデータ | RcpA-TadD with C13 symmetry from the Pseudomonas aeruginosa Tigh Adherence Secretion System | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Secretin / Pilotin / RcpA / TadD / TAD / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Tassinari M / Low HH | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Assembly mechanism of a Tad secretion system secretin-pilotin complex. 著者: Matteo Tassinari / Marta Rudzite / Alain Filloux / Harry H Low /    要旨: The bacterial Tight adherence Secretion System (TadSS) assembles surface pili that drive cell adherence, biofilm formation and bacterial predation. The structure and mechanism of the TadSS is mostly ...The bacterial Tight adherence Secretion System (TadSS) assembles surface pili that drive cell adherence, biofilm formation and bacterial predation. The structure and mechanism of the TadSS is mostly unknown. This includes characterisation of the outer membrane secretin through which the pilus is channelled and recruitment of its pilotin. Here we investigate RcpA and TadD lipoprotein from Pseudomonas aeruginosa. Light microscopy reveals RcpA colocalising with TadD in P. aeruginosa and when heterologously expressed in Escherichia coli. We use cryogenic electron microscopy to determine how RcpA and TadD assemble a secretin channel with C13 and C14 symmetries. Despite low sequence homology, we show that TadD shares a similar fold to the type 4 pilus system pilotin PilF. We establish that the C-terminal four residues of RcpA bind TadD - an interaction essential for secretin formation. The binding mechanism between RcpA and TadD appears distinct from known secretin-pilotin pairings in other secretion systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16810.map.gz emd_16810.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16810-v30.xml emd-16810-v30.xml emd-16810.xml emd-16810.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16810_fsc.xml emd_16810_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16810.png emd_16810.png | 71.3 KB | ||
| その他 |  emd_16810_half_map_1.map.gz emd_16810_half_map_1.map.gz emd_16810_half_map_2.map.gz emd_16810_half_map_2.map.gz | 156.7 MB 135.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16810 http://ftp.pdbj.org/pub/emdb/structures/EMD-16810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16810 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16810 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16810_validation.pdf.gz emd_16810_validation.pdf.gz | 654.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16810_full_validation.pdf.gz emd_16810_full_validation.pdf.gz | 653.7 KB | 表示 | |
| XML形式データ |  emd_16810_validation.xml.gz emd_16810_validation.xml.gz | 20 KB | 表示 | |
| CIF形式データ |  emd_16810_validation.cif.gz emd_16810_validation.cif.gz | 26.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16810 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16810 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16810 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16810 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8odnMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16810.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16810.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RcpA-TadD with C13 symmetry from the Pseudomonas aeruginosa Tigh Adherence Secretion System | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: RcpA-TadD with C13 symmetry from the Pseudomonas aeruginosa...
| ファイル | emd_16810_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RcpA-TadD with C13 symmetry from the Pseudomonas aeruginosa Tigh Adherence Secretion System | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
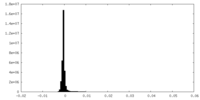
| 密度ヒストグラム |
-ハーフマップ: RcpA-TadD with C13 symmetry from the Pseudomonas aeruginosa...
| ファイル | emd_16810_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RcpA-TadD with C13 symmetry from the Pseudomonas aeruginosa Tigh Adherence Secretion System | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
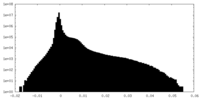
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RcpA-TadD with C13 symmetry
| 全体 | 名称: RcpA-TadD with C13 symmetry |
|---|---|
| 要素 |
|
-超分子 #1: RcpA-TadD with C13 symmetry
| 超分子 | 名称: RcpA-TadD with C13 symmetry / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 900 KDa |
-分子 #1: RcpA
| 分子 | 名称: RcpA / タイプ: protein_or_peptide / ID: 1 / コピー数: 13 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 45.507402 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHRSTGIGVS RWLGGLLGVA LALPALALPQ GCIELLAQAP RVDVVQGQQR DLRLAVPIER LAIGDPKIAD VQLLDRRGFL VTGKEQGST SLLIWTGCSP EPLRSLVEVE GRGSVDTRGA PAFTVGAAEE LPNQVQTDIR FVEVSRSKLK QASTSFVRRG G NLWVLGAP ...文字列: MHRSTGIGVS RWLGGLLGVA LALPALALPQ GCIELLAQAP RVDVVQGQQR DLRLAVPIER LAIGDPKIAD VQLLDRRGFL VTGKEQGST SLLIWTGCSP EPLRSLVEVE GRGSVDTRGA PAFTVGAAEE LPNQVQTDIR FVEVSRSKLK QASTSFVRRG G NLWVLGAP GSLGDIKVNA DGSGLGGTFG TGSSGFNLIF GGGKWLSFMN ALEGSGFAYT LARPSLVAMS GQSASFLAGG EF PIPVPNG TNDNVTIEYK EFGIRLTLTP TVMNNRRIAL KVAPEVSELD YSAGIQSGGV AVPALRVRRT DTSVMLADGE SFV ISGLTS SNSVSNVDKF PWLGDIPILG AFFRSTKLDK DDRELLMIVT PHLVQPLAAD AQLPDLPGEG LRHYDPGFSR LYFL ERGEY DGQQNDTGLS DSAWSHPQFE K UniProtKB: RcpA |
-分子 #2: TPR repeat-containing protein PA4299
| 分子 | 名称: TPR repeat-containing protein PA4299 / タイプ: protein_or_peptide / ID: 2 / コピー数: 13 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PAO1 (緑膿菌) Pseudomonas aeruginosa PAO1 (緑膿菌) |
| 分子量 | 理論値: 27.759648 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKALIGIGLC AALLGGCAAL PGRDGPRECS QQLGQEQELQ MNMVRDMIRE GRLHAALANL ESMPPGLLDV REERALILRR IGDPRARAE YQALLETCKA PEAHHGLGLL ALRNGDSARA VLELREAARL RPTESRFRND LGVALLKRGD RVGARFEFIT A LELQQGGK ...文字列: MKALIGIGLC AALLGGCAAL PGRDGPRECS QQLGQEQELQ MNMVRDMIRE GRLHAALANL ESMPPGLLDV REERALILRR IGDPRARAE YQALLETCKA PEAHHGLGLL ALRNGDSARA VLELREAARL RPTESRFRND LGVALLKRGD RVGARFEFIT A LELQQGGK LPATNLLGLL YLQGDREDAQ RLIERLQLDA RDIRAAEARA RSWGAVPTPG AAPASDDPLA ELPAEANMHT AM ANEAPGS DYKDDDDK UniProtKB: TPR repeat-containing protein PA4299 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 50 mM Hepes pH 8, 100 mM NaCl, 1 mM EDTA, 0.06% bDDM | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR 詳細: The grid was coated with graphene oxide prior to use | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X