+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
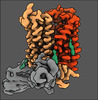
| タイトル | PHT1 in the outward facing conformation, bound to Sb27 | |||||||||
 マップデータ マップデータ | Map sharpened in cryoSPARC. Used for final model refinement. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | PHT1 / Peptide transporter / Histidine transporter / Sybody 27 / SLE / SLC15A4 / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 | :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   | |||||||||
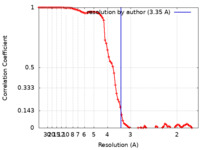
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||
 データ登録者 データ登録者 | Custodio T / Killer M / Loew C | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Molecular basis of TASL recruitment by the peptide/histidine transporter 1, PHT1. 著者: Tânia F Custódio / Maxime Killer / Dingquan Yu / Virginia Puente / Daniel P Teufel / Alexander Pautsch / Gisela Schnapp / Marc Grundl / Jan Kosinski / Christian Löw /  要旨: PHT1 is a histidine /oligopeptide transporter with an essential role in Toll-like receptor innate immune responses. It can act as a receptor by recruiting the adaptor protein TASL which leads to type ...PHT1 is a histidine /oligopeptide transporter with an essential role in Toll-like receptor innate immune responses. It can act as a receptor by recruiting the adaptor protein TASL which leads to type I interferon production via IRF5. Persistent stimulation of this signalling pathway is known to be involved in the pathogenesis of systemic lupus erythematosus (SLE). Understanding how PHT1 recruits TASL at the molecular level, is therefore clinically important for the development of therapeutics against SLE and other autoimmune diseases. Here we present the Cryo-EM structure of PHT1 stabilized in the outward-open conformation. By combining biochemical and structural modeling techniques we propose a model of the PHT1-TASL complex, in which the first 16 N-terminal TASL residues fold into a helical structure that bind in the central cavity of the inward-open conformation of PHT1. This work provides critical insights into the molecular basis of PHT1/TASL mediated type I interferon production. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16758.map.gz emd_16758.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16758-v30.xml emd-16758-v30.xml emd-16758.xml emd-16758.xml | 20.4 KB 20.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16758_fsc.xml emd_16758_fsc.xml | 9.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16758.png emd_16758.png | 149 KB | ||
| マスクデータ |  emd_16758_msk_1.map emd_16758_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16758.cif.gz emd-16758.cif.gz | 6 KB | ||
| その他 |  emd_16758_additional_1.map.gz emd_16758_additional_1.map.gz emd_16758_additional_2.map.gz emd_16758_additional_2.map.gz emd_16758_half_map_1.map.gz emd_16758_half_map_1.map.gz emd_16758_half_map_2.map.gz emd_16758_half_map_2.map.gz | 56.8 MB 30.9 MB 59.3 MB 59.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16758 http://ftp.pdbj.org/pub/emdb/structures/EMD-16758 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16758 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16758 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16758_validation.pdf.gz emd_16758_validation.pdf.gz | 790 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16758_full_validation.pdf.gz emd_16758_full_validation.pdf.gz | 789.5 KB | 表示 | |
| XML形式データ |  emd_16758_validation.xml.gz emd_16758_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  emd_16758_validation.cif.gz emd_16758_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16758 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16758 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16758 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16758 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8cniMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16758.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16758.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map sharpened in cryoSPARC. Used for final model refinement. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
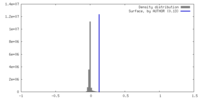
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16758_msk_1.map emd_16758_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
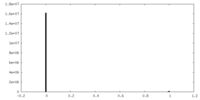
| 密度ヒストグラム |
-追加マップ: Map post processed with DeepEMhancer. Tight Target model....
| ファイル | emd_16758_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map post processed with DeepEMhancer. Tight Target model. Used for display and initial docking of sybody 27 only. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
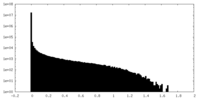
| 密度ヒストグラム |
-追加マップ: Unsharpened full map. Final Non Uniform Refinement in cryoSPARC.
| ファイル | emd_16758_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened full map. Final Non Uniform Refinement in cryoSPARC. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
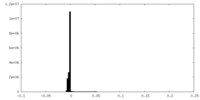
| 密度ヒストグラム |
-ハーフマップ: half map A
| ファイル | emd_16758_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half map B
| ファイル | emd_16758_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PHT1-Sb27
| 全体 | 名称: PHT1-Sb27 |
|---|---|
| 要素 |
|
-超分子 #1: PHT1-Sb27
| 超分子 | 名称: PHT1-Sb27 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: Solute carrier family 15 member 4
| 超分子 | 名称: Solute carrier family 15 member 4 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Sybody 27
| 超分子 | 名称: Sybody 27 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Solute carrier family 15 member 4
| 分子 | 名称: Solute carrier family 15 member 4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 63.566996 KDa |
| 組換発現 | 生物種:  Homo sapiens subsp. 'Denisova' (ヒト) Homo sapiens subsp. 'Denisova' (ヒト) |
| 配列 | 文字列: MEEAEQSERS PLLGSGERGR AAIGAFHGRR LACAAVLLAE LLERVAFYGI TSNLVLFLNG PPYDWEGAQA SQALLLFMGI TYLVSPFGG WLADALLGKF GTILLSMALY LLGMLAFPVI AAPHTRQGLC GDIPLYPVEN CSSPATNATL APCSQVGTTR Y CAAATFVG ...文字列: MEEAEQSERS PLLGSGERGR AAIGAFHGRR LACAAVLLAE LLERVAFYGI TSNLVLFLNG PPYDWEGAQA SQALLLFMGI TYLVSPFGG WLADALLGKF GTILLSMALY LLGMLAFPVI AAPHTRQGLC GDIPLYPVEN CSSPATNATL APCSQVGTTR Y CAAATFVG LVLVGLGVGS VKANITPFGA DQVKDRGPEA TRRFFNWFYW SINLGAILSL GGIAYIQQNV SFVIGYSIPA IC IGISFMV FLCGQSFFIT KPPDGSAFTD MFKILAYSCC SRKRHMEHST NSEGQGVLQQ PRKQSLFEMA KLSRGGPFRE DKV EDVKAL VKIIPVFLAL IPYWTVYFQM QTTYVLQSLH LKIPEIANDT NSVHTFPAAW LTMFDAVLIL ILIPLKDKLV DPVL KRNGL LPSSLKRIAV GMFFVMCSAF AAGILESNRL KIVKVKTINQ TIGNVTYHAA DMPIWWQIPQ YVLIGFSEIF ASIAG LEFA YSAAPKSMQS AIMGLFFFFS GIGSFVGSGL LALVSIKEIG WMSNHTDFGN INGCQLNYYF FLLAAIQGAT LLLFLI VSV KYDHQKSKMN DVAANGRI UniProtKB: UNIPROTKB: F1NG54 |
-分子 #2: Sybody 27
| 分子 | 名称: Sybody 27 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.504001 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVESGGG SVQAGGSLRL SCAASGDIET IWYLGWFRQA PGKEREGVAA LSTVTGSTYY ADSVKGRFTV SLDNAKNTVY LQMNSLKPE DTALYYCAAA YTGWMAPLWQ WVYSYWGQGT QVTVS |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 75.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: experimental model |
|---|---|
| 得られたモデル |  PDB-8cni: |
 ムービー
ムービー コントローラー
コントローラー



 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)