[日本語] English
 万見
万見- EMDB-16727: Human 80S ribosome at the 'eEF2, ap/P, pe/E' state in untreated cells -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human 80S ribosome at the 'eEF2, ap/P, pe/E' state in untreated cells | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | human / 80S ribosome / translation / tRNA / RIBOSOME | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 9.6 Å | |||||||||
 データ登録者 データ登録者 | Xing H / Welsch S / Turonova B / Beck M | |||||||||
| 資金援助 |  ドイツ, 1件 ドイツ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2023 ジャーナル: Science / 年: 2023タイトル: Translation dynamics in human cells visualized at high resolution reveal cancer drug action. 著者: Huaipeng Xing / Reiya Taniguchi / Iskander Khusainov / Jan Philipp Kreysing / Sonja Welsch / Beata Turoňová / Martin Beck /  要旨: Ribosomes catalyze protein synthesis by cycling through various functional states. These states have been extensively characterized in vitro, but their distribution in actively translating human ...Ribosomes catalyze protein synthesis by cycling through various functional states. These states have been extensively characterized in vitro, but their distribution in actively translating human cells remains elusive. We used a cryo-electron tomography-based approach and resolved ribosome structures inside human cells with high resolution. These structures revealed the distribution of functional states of the elongation cycle, a Z transfer RNA binding site, and the dynamics of ribosome expansion segments. Ribosome structures from cells treated with Homoharringtonine, a drug used against chronic myeloid leukemia, revealed how translation dynamics were altered in situ and resolve the small molecules within the active site of the ribosome. Thus, structural dynamics and drug effects can be assessed at high resolution within human cells. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16727.map.gz emd_16727.map.gz | 115.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16727-v30.xml emd-16727-v30.xml emd-16727.xml emd-16727.xml | 11.8 KB 11.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_16727.png emd_16727.png | 80.4 KB | ||
| その他 |  emd_16727_half_map_1.map.gz emd_16727_half_map_1.map.gz emd_16727_half_map_2.map.gz emd_16727_half_map_2.map.gz | 115.9 MB 115.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16727 http://ftp.pdbj.org/pub/emdb/structures/EMD-16727 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16727 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16727 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16727_validation.pdf.gz emd_16727_validation.pdf.gz | 1.4 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16727_full_validation.pdf.gz emd_16727_full_validation.pdf.gz | 1.4 MB | 表示 | |
| XML形式データ |  emd_16727_validation.xml.gz emd_16727_validation.xml.gz | 13.9 KB | 表示 | |
| CIF形式データ |  emd_16727_validation.cif.gz emd_16727_validation.cif.gz | 16.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16727 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16727 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16727 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16727 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16727.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16727.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.188 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_16727_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
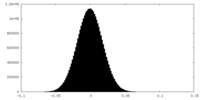
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16727_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
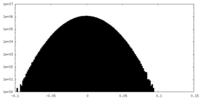
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human 80S ribosome at the 'eEF2, ap/P, pe/E' state in untreated cells
| 全体 | 名称: Human 80S ribosome at the 'eEF2, ap/P, pe/E' state in untreated cells |
|---|---|
| 要素 |
|
-超分子 #1: Human 80S ribosome at the 'eEF2, ap/P, pe/E' state in untreated cells
| 超分子 | 名称: Human 80S ribosome at the 'eEF2, ap/P, pe/E' state in untreated cells タイプ: complex / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 2.46 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 9.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用したサブトモグラム数: 2037 |
|---|---|
| 抽出 | トモグラム数: 358 / 使用した粒子像数: 286400 |
| 最終 角度割当 | タイプ: OTHER |
 ムービー
ムービー コントローラー
コントローラー






























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)