+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the fd bacteriophage capsid major coat protein pVIII | ||||||||||||||||||||||||
 マップデータ マップデータ | Cryo-EM map of the intact fd bacteriophage capsid | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
 キーワード キーワード | Bacteriophage / fd / inovirus / ff / helical / phage / filamentous / VIRUS | ||||||||||||||||||||||||
| 機能・相同性 | Phage major coat protein, Gp8 / Bacteriophage M13, G8P, capsid domain superfamily / Capsid protein G8P / helical viral capsid / host cell membrane / membrane / Capsid protein G8P 機能・相同性情報 機能・相同性情報 | ||||||||||||||||||||||||
| 生物種 |   Enterobacteria phage fd (ファージ) Enterobacteria phage fd (ファージ) | ||||||||||||||||||||||||
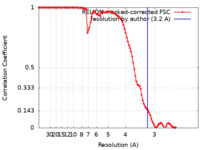
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Boehning J / Bharat TAM | ||||||||||||||||||||||||
| 資金援助 |  英国, 英国,  フランス, フランス,  米国, European Union, 7件 米国, European Union, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Biophysical basis of filamentous phage tactoid-mediated antibiotic tolerance in P. aeruginosa. 著者: Jan Böhning / Miles Graham / Suzanne C Letham / Luke K Davis / Ulrike Schulze / Phillip J Stansfeld / Robin A Corey / Philip Pearce / Abul K Tarafder / Tanmay A M Bharat /  要旨: Inoviruses are filamentous phages infecting numerous prokaryotic phyla. Inoviruses can self-assemble into mesoscale structures with liquid-crystalline order, termed tactoids, which protect bacterial ...Inoviruses are filamentous phages infecting numerous prokaryotic phyla. Inoviruses can self-assemble into mesoscale structures with liquid-crystalline order, termed tactoids, which protect bacterial cells in Pseudomonas aeruginosa biofilms from antibiotics. Here, we investigate the structural, biophysical, and protective properties of tactoids formed by the P. aeruginosa phage Pf4 and Escherichia coli phage fd. A cryo-EM structure of the capsid from fd revealed distinct biochemical properties compared to Pf4. Fd and Pf4 formed tactoids with different morphologies that arise from differing phage geometries and packing densities, which in turn gave rise to different tactoid emergent properties. Finally, we showed that tactoids formed by either phage protect rod-shaped bacteria from antibiotic treatment, and that direct association with a tactoid is required for protection, demonstrating the formation of a diffusion barrier by the tactoid. This study provides insights into how filamentous molecules protect bacteria from extraneous substances in biofilms and in host-associated infections. #1:  ジャーナル: Biorxiv / 年: 2023 ジャーナル: Biorxiv / 年: 2023タイトル: Biophysical basis of phage liquid crystalline droplet-mediated antibiotic tolerance in pathogenic bacteria 著者: Bohning J / Graham M / Letham S / Davis L / Schulze U / Stansfeld P / Corey R / Pearce P / Tarafder A / Bharat T | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16657.map.gz emd_16657.map.gz | 96.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16657-v30.xml emd-16657-v30.xml emd-16657.xml emd-16657.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16657_fsc.xml emd_16657_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16657.png emd_16657.png | 101.5 KB | ||
| Filedesc metadata |  emd-16657.cif.gz emd-16657.cif.gz | 5.9 KB | ||
| その他 |  emd_16657_half_map_1.map.gz emd_16657_half_map_1.map.gz emd_16657_half_map_2.map.gz emd_16657_half_map_2.map.gz | 81.1 MB 80.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16657 http://ftp.pdbj.org/pub/emdb/structures/EMD-16657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16657 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16657_validation.pdf.gz emd_16657_validation.pdf.gz | 997.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16657_full_validation.pdf.gz emd_16657_full_validation.pdf.gz | 997.2 KB | 表示 | |
| XML形式データ |  emd_16657_validation.xml.gz emd_16657_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  emd_16657_validation.cif.gz emd_16657_validation.cif.gz | 23.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16657 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16657 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16657 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16657 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8ch5MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16657.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16657.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of the intact fd bacteriophage capsid | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.092 Å | ||||||||||||||||||||||||||||||||||||
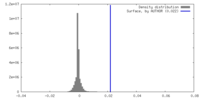
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half-map 2
| ファイル | emd_16657_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
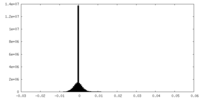
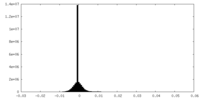
| 密度ヒストグラム |
-ハーフマップ: Half-map 1
| ファイル | emd_16657_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
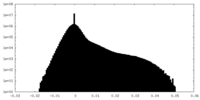
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Enterobacteria phage fd
| 全体 | 名称:  Enterobacteria phage fd (ファージ) Enterobacteria phage fd (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Enterobacteria phage fd
| 超分子 | 名称: Enterobacteria phage fd / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Purified by PEG precipitation from the supernatant of infected E. coli cells. NCBI-ID: 10864 / 生物種: Enterobacteria phage fd / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-分子 #1: Major capsid protein pVIII
| 分子 | 名称: Major capsid protein pVIII / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 5.244 KDa |
| 配列 | 文字列: AEGDDPAKAA FDSLQASATE YIGYAWAMVV VIVGATIGIK LFKKFTSKAS UniProtKB: Capsid protein G8P |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: Phosphate-buffered saline |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/RHODIUM / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR / 詳細: 15 mA |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | fd phage in PBS |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 平均電子線量: 53.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 79.94 |
|---|---|
| 得られたモデル |  PDB-8ch5: |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)