+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AQP7_inhibitor | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | aquaglyceroporin / glycerol channel / dimer of tetramers / inhibitor / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Transport of glycerol from adipocytes to the liver by Aquaporins / glycerol channel activity / urea transmembrane transporter activity / Passive transport by Aquaporins / glycerol transmembrane transport / water transport / water channel activity / lipid droplet / cytoplasmic vesicle membrane / cell-cell junction ...Transport of glycerol from adipocytes to the liver by Aquaporins / glycerol channel activity / urea transmembrane transporter activity / Passive transport by Aquaporins / glycerol transmembrane transport / water transport / water channel activity / lipid droplet / cytoplasmic vesicle membrane / cell-cell junction / cell cortex / basolateral plasma membrane / plasma membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.2 Å | |||||||||
 データ登録者 データ登録者 | Huang P / Venskutonyte R / Gourdon P / Lindkvist-Petersson K | |||||||||
| 資金援助 |  スウェーデン, 2件 スウェーデン, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2024 ジャーナル: Proc Natl Acad Sci U S A / 年: 2024タイトル: Molecular basis for human aquaporin inhibition. 著者: Peng Huang / Hannah Åbacka / Carter J Wilson / Malene Lykke Wind / Michael Rűtzler / Anna Hagström-Andersson / Pontus Gourdon / Bert L de Groot / Raminta Venskutonytė / Karin Lindkvist-Petersson /    要旨: Cancer invasion and metastasis are known to be potentiated by the expression of aquaporins (AQPs). Likewise, the expression levels of AQPs have been shown to be prognostic for survival in patients ...Cancer invasion and metastasis are known to be potentiated by the expression of aquaporins (AQPs). Likewise, the expression levels of AQPs have been shown to be prognostic for survival in patients and have a role in tumor growth, edema, angiogenesis, and tumor cell migration. Thus, AQPs are key players in cancer biology and potential targets for drug development. Here, we present the single-particle cryo-EM structure of human AQP7 at 3.2-Å resolution in complex with the specific inhibitor compound Z433927330. The structure in combination with MD simulations shows that the inhibitor binds to the endofacial side of AQP7. In addition, cancer cells treated with Z433927330 show reduced proliferation. The data presented here serve as a framework for the development of AQP inhibitors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_16510.map.gz emd_16510.map.gz | 97.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-16510-v30.xml emd-16510-v30.xml emd-16510.xml emd-16510.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_16510_fsc.xml emd_16510_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_16510.png emd_16510.png | 31.5 KB | ||
| マスクデータ |  emd_16510_msk_1.map emd_16510_msk_1.map | 103 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-16510.cif.gz emd-16510.cif.gz | 6.3 KB | ||
| その他 |  emd_16510_half_map_1.map.gz emd_16510_half_map_1.map.gz emd_16510_half_map_2.map.gz emd_16510_half_map_2.map.gz | 95.3 MB 95.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-16510 http://ftp.pdbj.org/pub/emdb/structures/EMD-16510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-16510 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_16510_validation.pdf.gz emd_16510_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_16510_full_validation.pdf.gz emd_16510_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_16510_validation.xml.gz emd_16510_validation.xml.gz | 18.3 KB | 表示 | |
| CIF形式データ |  emd_16510_validation.cif.gz emd_16510_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16510 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16510 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16510 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-16510 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8c9hMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_16510.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_16510.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8464 Å | ||||||||||||||||||||||||||||||||||||
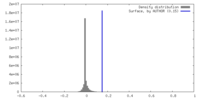
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_16510_msk_1.map emd_16510_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
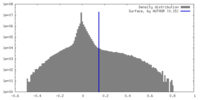
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_16510_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_16510_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AQP7 dimer of tetramers with inhibitor
| 全体 | 名称: AQP7 dimer of tetramers with inhibitor |
|---|---|
| 要素 |
|
-超分子 #1: AQP7 dimer of tetramers with inhibitor
| 超分子 | 名称: AQP7 dimer of tetramers with inhibitor / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 / 詳細: sample was prepared in GDN detergent |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Aquaporin-7
| 分子 | 名称: Aquaporin-7 / タイプ: protein_or_peptide / ID: 1 / 詳細: LIG represents the inhibitor / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 37.264395 KDa |
| 組換発現 | 生物種:  Komagataella pastoris (菌類) Komagataella pastoris (菌類) |
| 配列 | 文字列: MVQASGHRRS TRGSKMVSWS VIAKIQEILQ RKMVREFLAE FMSTYVMMVF GLGSVAHMVL NKKYGSYLGV NLGFGFGVTM GVHVAGRIS GAHMNAAVTF ANCALGRVPW RKFPVYVLGQ FLGSFLAAAT IYSLFYTAIL HFSGGQLMVT GPVATAGIFA T YLPDHMTL ...文字列: MVQASGHRRS TRGSKMVSWS VIAKIQEILQ RKMVREFLAE FMSTYVMMVF GLGSVAHMVL NKKYGSYLGV NLGFGFGVTM GVHVAGRIS GAHMNAAVTF ANCALGRVPW RKFPVYVLGQ FLGSFLAAAT IYSLFYTAIL HFSGGQLMVT GPVATAGIFA T YLPDHMTL WRGFLNEAWL TGMLQLCLFA ITDQENNPAL PGTEALVIGI LVVIIGVSLG MNTGYAINPS RDLPPRIFTF IA GWGKQVF SNGENWWWVP VVAPLLGAYL GGIIYLVFIG STIPREPLKL EDSVAYEDHG ITVLPKMGSH EPTISPLTPV SVS PANRSS VHPAPPLHES MALEHF UniProtKB: Aquaporin-7 |
-分子 #2: ethyl 4-[(4-pyrazol-1-ylphenyl)methylcarbamoylamino]benzoate
| 分子 | 名称: ethyl 4-[(4-pyrazol-1-ylphenyl)methylcarbamoylamino]benzoate タイプ: ligand / ID: 2 / コピー数: 8 / 式: T60 |
|---|---|
| 分子量 | 理論値: 364.398 Da |
| Chemical component information | 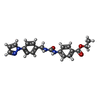 ChemComp-T60: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 詳細: 20mA for glow discharge | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK II / 詳細: 3s blot, 3s wait, 0s drain time, 0 blot force. | ||||||||||||
| 詳細 | 25uM inhibitor was supplemented into protein solution before grid freezing. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 9002 / 平均露光時間: 2.16 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 105000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 詳細 | Initial model (PDB ID:8AMX) was fitted locally into map by Chimera. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 108 |
| 得られたモデル |  PDB-8c9h: |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)